Поиск ДНК-белковых контактов в заданной структуре
Использование команд define для задания множества атомов в Jmol'e
Для выполнения задания были:
- Определено множество атомов кислорода 2'-дезоксирибозы (~2'_ribose_O)
- Определено множество атомов кислорода в остатке фосфорной кислоты (~O_in_phospho)
- Определено множество атомов азота в азотистых основаниях (~N_in_bases)
- Создан
 скрипт-файл 1_Jmol_script.txt с определениями этих множеств
скрипт-файл 1_Jmol_script.txt с определениями этих множеств - Создан
 скрипт-файл 2_Jmol_script.txt, вызов которого в JMol даст последовательное изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов ~2'_ribose_O, затем ~O_in_phospho и ~N_in_bases
скрипт-файл 2_Jmol_script.txt, вызов которого в JMol даст последовательное изображение всей структуры, только ДНК в проволочной модели, той же модели, но с выделенными шариками множеством атомов ~2'_ribose_O, затем ~O_in_phospho и ~N_in_bases - Создана
 рассширенная версия пердыдущего скрипта, результатом работы которой стали Рис. 1 - Рис. 5.
рассширенная версия пердыдущего скрипта, результатом работы которой стали Рис. 1 - Рис. 5.
1_Jmol_script.txt
2_Jmol_script.txt
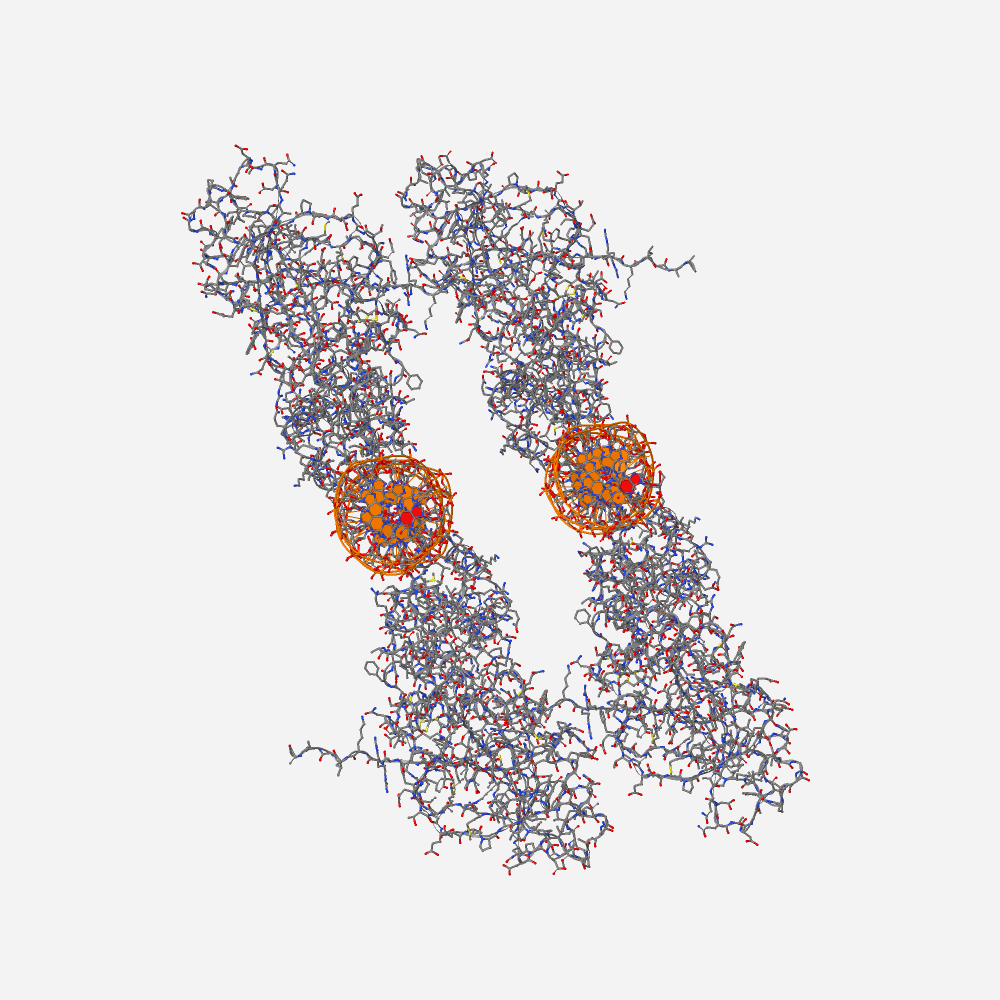
Рис. 1. Изображение всей структуры, содержащейся в pdb 1D5Y. Раскраска атомов по схеме cpk.
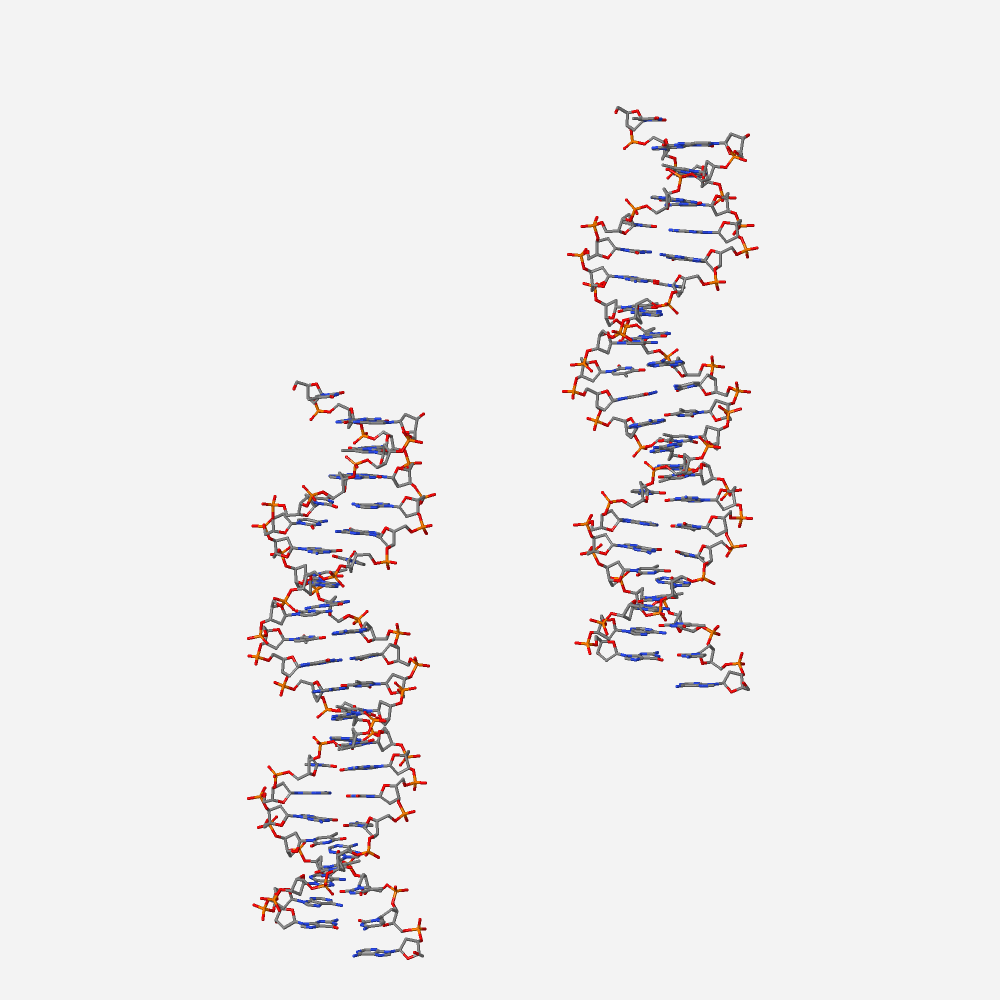
Рис. 2. Изображение только ДНК из структуры pdb 1D5Y в проволочной модели. Раскраска атомов по схеме cpk.

Рис. 3. Изображение только ДНК из структуры pdb 1D5Y, атомы кислорода 2'-дезоксирибозы выделены большим размером. Раскраска атомов по схеме cpk.
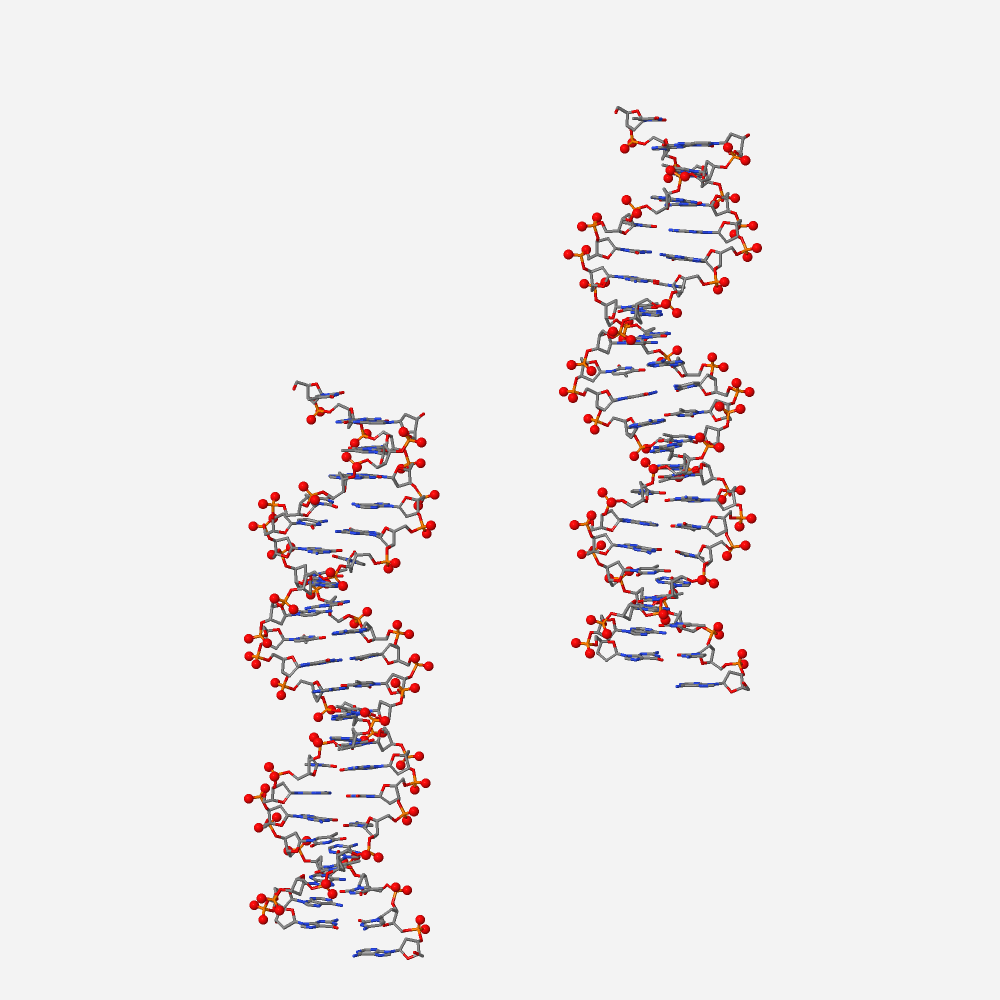
Рис. 4. Изображение только ДНК из структуры pdb 1D5Y, атомы атомов кислорода в остатке фосфорной кислоты выделены большим размером. Раскраска атомов по схеме cpk.
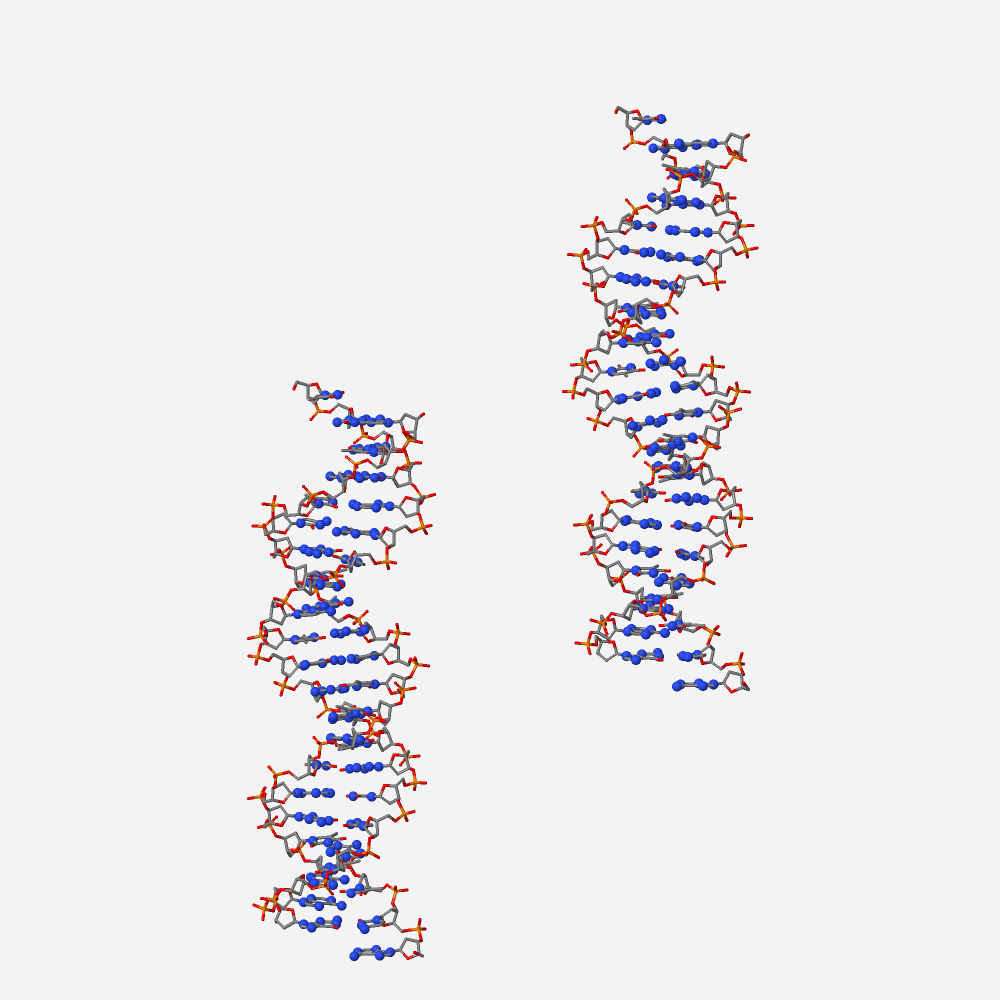
Рис. 5. Изображение только ДНК из структуры pdb 1D5Y, атомы азота в азотистых основаниях выделены большим размером. Раскраска атомов по схеме cpk.
Описание ДНК-белковых контактов в заданной структуре. Сравнение количества контактов разной природы.
Введем понятия и допущения:
- Будем считать полярными атомы кислорода и азота, а неполярными атомы углерода, фосфора и серы.
- Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК меньше 3.5 Å
- Назовем неполярным контактом ситуацию в которой расстояние между неполярными атомами меньше 4.5 Å .
| контакты атома белка с | полярные | неполярные | всего |
| остатками 2'-дезоксирибозы | 6 | 31 | 37 |
| остатками фосфорной кислоты | 31 | 19 | 50 |
| остатками азотистых оснований со стороны большой бороздки | 6 | 14 | 20 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 0 | 0 |
Как видно из Таблицы 1 наиболее вовлечены в различные взаимодействия с белками остатки фосфорной кислоты, однако при этом в неполярные взаимодействия гораздо больше вовлечены атомы 2'-дезоксирибозы. Отдельно стоит отметить, что в взаимодействиях с белком атомы малой бороздки вообще не участвуют.
Получение популярной схемы ДНК-белковых контактов с помощью программы nucplot
Для получения схемы ДНК-белковых взаимодействий была запущена на сервере следующая последовательность команд:
получение схемы ДНК-белковых взаимодействий
remediator --old --pdb 1D5Y.pdb >old_1D5Y.pdb
nucplot old_1D5Y.pdb
convert nucplot.ps nucplot.png
Результат выполнения этих команд представлен на Рис. 6- Рис. 11
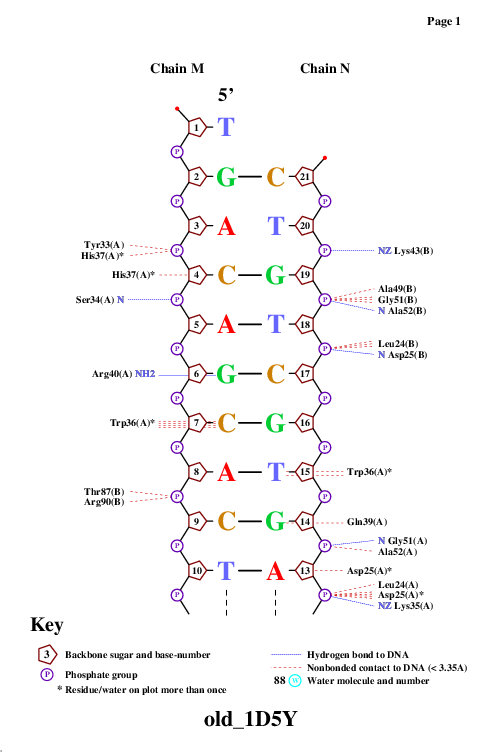
Рис. 6 Популярная схема ДНК-белковых взаимодействий в структуре 1D5Y pdb. Лист 1.
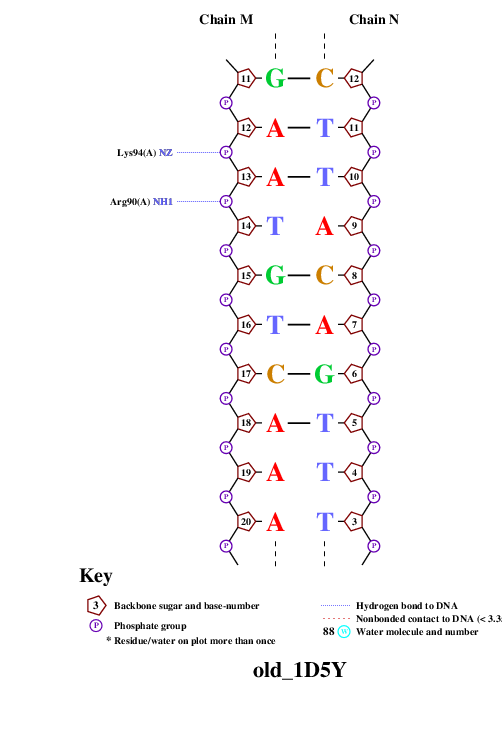
Рис. 7 Популярная схема ДНК-белковых взаимодействий в структуре 1D5Y pdb. Лист 2.
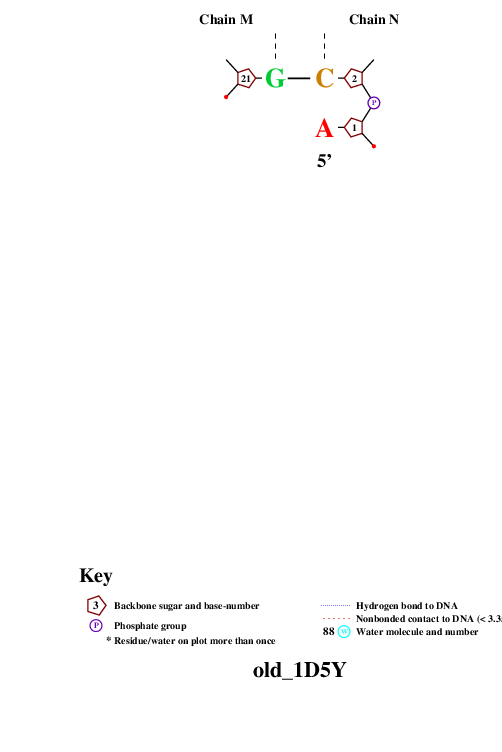
Рис. 8 Популярная схема ДНК-белковых взаимодействий в структуре 1D5Y pdb. Лист 3.
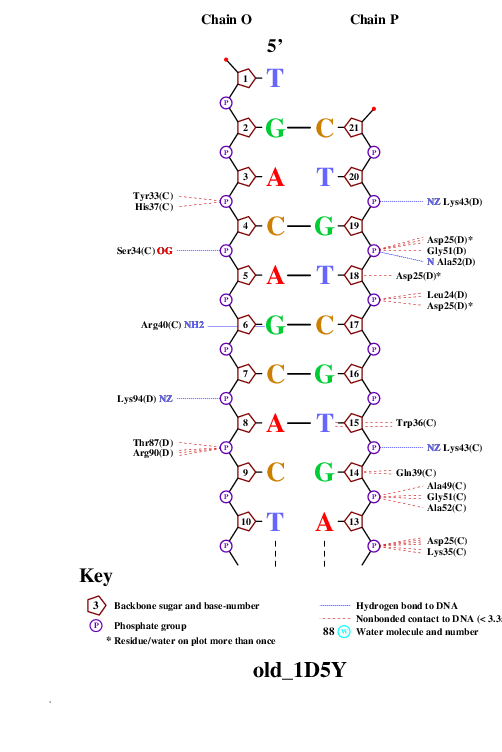
Рис. 9 Популярная схема ДНК-белковых взаимодействий в структуре 1D5Y pdb. Лист 4.
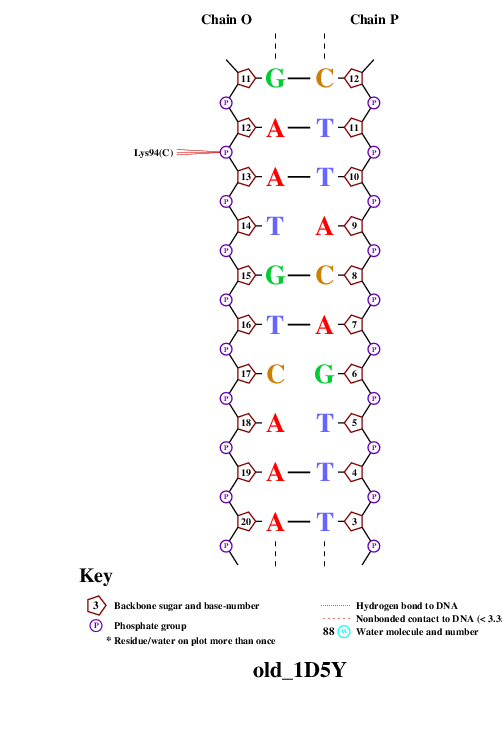
Рис. 10 Популярная схема ДНК-белковых взаимодействий в структуре 1D5Y pdb. Лист 5.
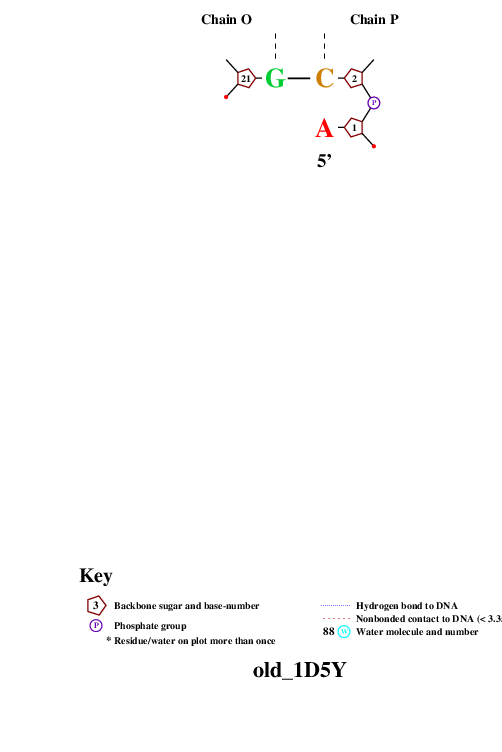
Рис. 11 Популярная схема ДНК-белковых взаимодействий в структуре 1D5Y pdb. Лист 6.
Поиск на схеме ДНК-белковых взаимодйствий важных аминокислотных остатков
Найден аминокислотный остаток с наибольшим числом указанных на схеме контактов с ДНК: Trp36(A)
Он представлен на Рис. 12, для получения которого использовался скрипт 5_Jmol_script.txt
5_Jmol_script.txt
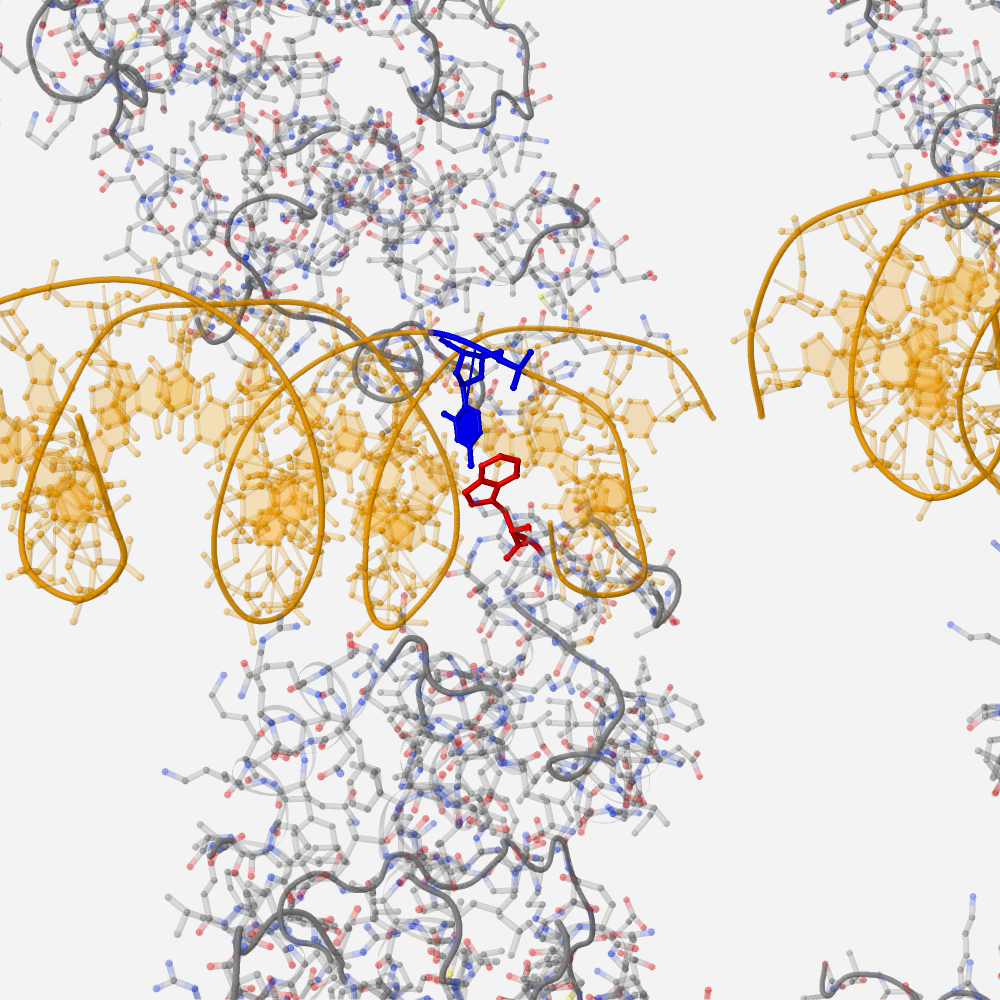
Рис. 12 Участок взаимодействия Trp36(A) ( покрашен красным) с ДНК: [DC]7:M (покрашен синим).
Найден аминокислотный остаток, наиболее важный для распознавания последовательности ДНК: это тоже Trp36(A)
Это обусловлено тем, что он связывается именно с азотистым основанием в цепи ДНК. Данный участок представлен на Рис. 12.
Предсказание вторичной структуры заданной тРНК
Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
Для предсказания вторичной структуры тРНК была  скачана последовательность тРНК из pdb 1EHZ и использована программа einverted из пакета EMBOSS.
скачана последовательность тРНК из pdb 1EHZ и использована программа einverted из пакета EMBOSS.
При стандартных настройках ничего путного не получилось. В виду наличия вычислительных мощьностей было решено перебором подобрать оптимальные настройки
Для перебора был использован следующий скрипт:
перебор
В результате было получено большое к-во файлов, после чего были удалены дубликаты. И осуществлен поиск по оставшимся файлам при помощи следующих реглулярных выражений:
^\s{6}1[a|t|g|c|\s|-]*7\s{6}$
^\s{6}10[a|t|g|c|\s|-]*13\s{6}$
^\s{6}49[a|t|g|c|\s|-]*53\s{6}$
^\s{6}38[a|t|g|c|\s|-]*44\s{6}$
Анализ данных показал, что более-менее результаты могут быть получены комадной einverted при следующих настройках:
Предсказание вторичной структуры тРНК по алгоритму Зукера
Реальная и предсказанная вторичная структура тРНК из файла 1EHZ.pdb
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5'-1-7-3' 5'-66-72-3' Всего 7 пар (6 канонич.) | ни при каких настройках целиком не находится | нашлись 6 канонических |
| D-стебель | 5'-10-13-3' 5'-22-25-3' Всего 4 пары (все канонич.) | нашлись все 4 пары | нашлись все 4 канонические пары |
| T-стебель | 5'-49-53-3' 5'-61-65-3' Всего 5 (все канонич.) | нашлись все 5 пар | нашлись все 5 канонических пар |
| Антикодоновый стебель | 5'-38-44-3' 5'-26-32-3' Всего 7 пар (5 канонических) | ни при каких настройках целиком не находится | нашлись 5 канонических пар пар |
| Общее число каноничекских не модифицированных пар нуклеотидов | 20 | 9 | 20 |
Таблица 2. Сравнение реальной и предсказанной структуры тРНК из pdb 1EHZ.
Результат предсказания вторичной структуры по алгоритму Зукера
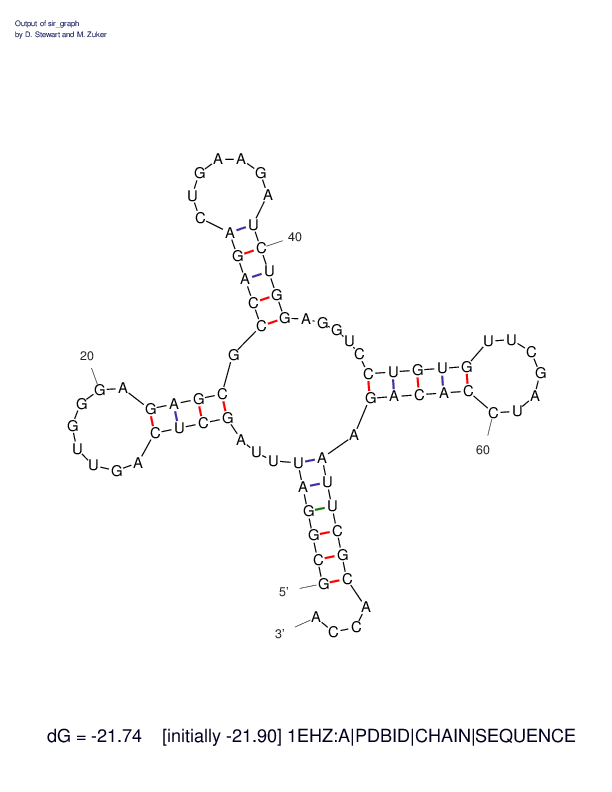
Рис. 12 Результат предсказания вторичной структуры тРНК из pdb 1EHZ по алгоритму Зукера.
