Построение дерева по нуклеотидным последовательностям
Для работы были  получены последовательности в формате fasta 16S РНК бактерий с мнемониками: BACAN,CLOB1,ENTFA,FINM2,GEOKA,LACAC,LISMO,STAA1. Особенности 16S РНК показаны в Таблице 1.
получены последовательности в формате fasta 16S РНК бактерий с мнемониками: BACAN,CLOB1,ENTFA,FINM2,GEOKA,LACAC,LISMO,STAA1. Особенности 16S РНК показаны в Таблице 1.
| мнемоника вида | AC embl | координаты | цепь |
| BACAN | AE016879 | 9335:10841 | + |
| CLOB1 | CP000726 | 9282:10783 | + |
| ENTFA | AE016830 | 248466:249987 | + |
| FINM2 | AP008971 | 197837:199361 | + |
| GEOKA | BA000043 | 10421:11973 | + |
| LACAC | CP000033 | 59255:60826 | + |
| LISMO | AL591980 | 96266:97811 | - |
| STAA1 | AP009324 | 531922:533476 | + |
Таблица 1. Данные о найденных 16S РНК.
Полученные последовательности были  выровнены программой t-coffee. На основе выравнивания в программе MEGA было
выровнены программой t-coffee. На основе выравнивания в программе MEGA было  построено филогенетическое дерево по алгоритму максимального правдоподобия, которое представлено на Рисунке 1.
построено филогенетическое дерево по алгоритму максимального правдоподобия, которое представлено на Рисунке 1.
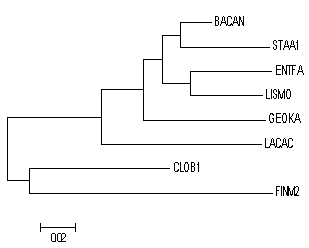
Рисунок 1. Дерево бактерий, построенное программой MEGA по алгоритму максимального правдоподобия на основе множественного выравнивания программой t-coffee 16S РНК.
Сравнение топологии полученного дерева с реальным:
Правильное дерево
/-CLOB1
/-|
| \-FINM2
|
--| /-LACAC
| /-|
| | \-ENTFA
\-|
| /-STAA1
\-|
| /-LISMO
\-|
| /-GEOKA
\-|
\-BACAN
Полученное дерево
/-BACAN
/-|
| \-STAA1
/-|
| | /-ENTFA
/-| \-|
| | \-LISMO
/-| |
| | \-GEOKA
| |
--| \-LACAC
|
| /-CLOB1
\-|
\-FINM2
Нетривиальные ветви Правильного дерева
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'ENTFA', 'GEOKA', 'LACAC', 'LISMO', 'STAA1'}vs{'CLOB1', 'FINM2'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Нетривиальные ветви Полученного дерева
{'BACAN', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'FINM2'}
{'BACAN', 'ENTFA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'GEOKA', 'FINM2'}
{'ENTFA', 'LISMO'}vs{'BACAN', 'GEOKA', 'FINM2', 'LACAC', 'STAA1', 'CLOB1'}
{'CLOB1', 'FINM2'}vs{'BACAN', 'GEOKA', 'LACAC', 'ENTFA', 'STAA1', 'LISMO'}
{'BACAN', 'ENTFA', 'GEOKA', 'LACAC', 'LISMO', 'STAA1'}vs{'CLOB1', 'FINM2'}
{'BACAN', 'STAA1'}vs{'GEOKA', 'FINM2', 'LACAC', 'ENTFA', 'CLOB1', 'LISMO'}
Ветви которые есть в Правильном дереве, но отсутствуют в Полученном
{'ENTFA', 'LACAC'}vs{'BACAN', 'GEOKA', 'FINM2', 'STAA1', 'CLOB1', 'LISMO'}
{'BACAN', 'GEOKA'}vs{'FINM2', 'LACAC', 'ENTFA', 'STAA1', 'CLOB1', 'LISMO'}
{'BACAN', 'GEOKA', 'LISMO'}vs{'LACAC', 'CLOB1', 'ENTFA', 'STAA1', 'FINM2'}
{'BACAN', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'ENTFA', 'FINM2'}
Ветви которые есть в Полученном дереве, но отсутствуют в Правильном
{'BACAN', 'ENTFA', 'GEOKA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'FINM2'}
{'ENTFA', 'LISMO'}vs{'BACAN', 'GEOKA', 'FINM2', 'LACAC', 'STAA1', 'CLOB1'}
{'BACAN', 'STAA1'}vs{'GEOKA', 'FINM2', 'LACAC', 'ENTFA', 'CLOB1', 'LISMO'}
{'BACAN', 'ENTFA', 'LISMO', 'STAA1'}vs{'LACAC', 'CLOB1', 'GEOKA', 'FINM2'}
Построение и анализ дерева, содержащего паралоги
Для поиска паралогов и гомологов белка CLPX_BACSU была использована программа blastp с e-value 1e-5 . Поиск проводился среди протеомов рассматриваемых бактерий. После чего найденные белки были извлечены из базы данных для анализа, после чего при помощи регулярных выражений имена были приведены в надлежащий вид. Полный код процесса представлен ниже.
Получение гомологичных\ортологичных последовательностей
~/blast/ncbi-blast/bin/makeblastdb -in proteo.fasta -dbtype prot -parse_seqids
~/blast/ncbi-blast/bin/blastp -task blastp -query ./CLPX_BACSU.fasta -db proteo.fasta -evalue 1e-5 -outfmt "6 sseqid" -out blastp.txt
~/blast/ncbi-blast/bin/blastdbcmd -db proteo.fasta -entry_batch blastp.txt -outfmt "%f" -out proteins_for_tree.fasta
Полученные  белковые последовательности в fasta формате были очищены от белков не рассматриваемых бактерий следующим скриптом:
белковые последовательности в fasta формате были очищены от белков не рассматриваемых бактерий следующим скриптом:
clean.py
В результате  получен фаста файл с результатами blastp относящимися только к белкам из бактерий с мнемоникакми 'BACAN', 'GEOKA', 'LISMO', 'STAA1','LACAC', 'CLOB1', 'ENTFA', 'FINM2'.
получен фаста файл с результатами blastp относящимися только к белкам из бактерий с мнемоникакми 'BACAN', 'GEOKA', 'LISMO', 'STAA1','LACAC', 'CLOB1', 'ENTFA', 'FINM2'.
Далее эти белки белки были выровнены программой t-coffee,  получен фаста файл с выровненными последовательностями.
получен фаста файл с выровненными последовательностями.
По полученному выравниванию при помощи программы MEGA было построено дерево гомологов белка CLPX_BACSU по алгоритму объединения соседей с поддержкой бутстрепом (500 реплик) на основе множественного выравнивания гомологичных последовательностей программой t-coffee. Консенсусное дерево представлено на Рисунке 2. На нем же подписаны ортологи и паралоги; зеленый квадрат-дупликация гена, желтый круг-видообразование.
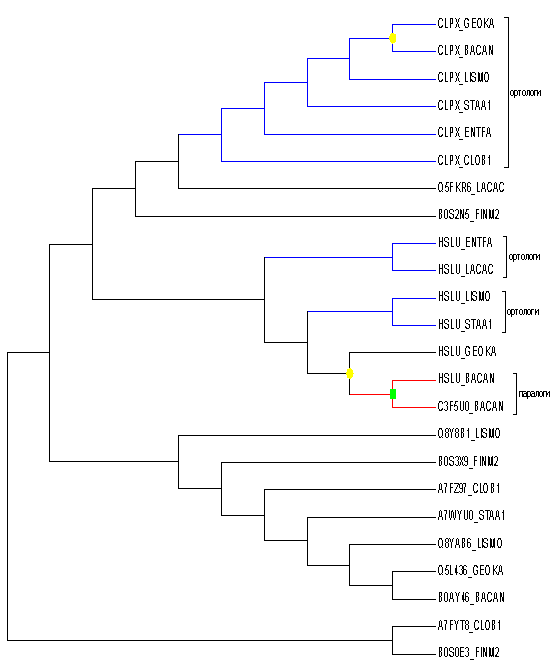
Рисунок 2. Консенсусное дерево гомологов белка CLPX_BACSU построенное программой MEGA по алгоритму по алгоритму объединения соседей с поддержкой бутстрепом (500 реплик) на основе множественного выравнивания программой t-coffee. Синие ветви-ветви ортологов, красные ветви-паралогов, желтый круг-видообразование, зеленый квадрат-дупликация гена.
