Краткое описание характеристик некоторых трансмембранных белков.
Для работы были случайно отобраны из базы данных OPM три белка с альфа-спиралями и три белка с бета-листами, отвечающими за трансмембранное положение. Основные данные взяты из самой базы данных, расчет медианы проводился при помощи библиотеки SciPy на языке программирования Python. Результаты представленны в Таблице 1.
| PDB код | Тип
(спираль, баррель) , описание |
Какая мембрана (внутренняя или внешняя, организм, органелла) |
Толщина гидрофобной части мембраны в ангстремах | Медиана числа остатков в одном трансмембранном участке |
| 1xkw | баррель Fe(III)-pyochelin рецептор |
Внешняя мембрана Грам-отрицательных бактерий | 24.8 ± 1.0 Å | 9.0 |
| 2lhf | баррель Outer membrane protein H (OprH) |
Внутренняя мембрана Грам-отрицательных бактерий | 20.6 ± 0.6 Å | 8.0 |
| 4e1t | баррель Invasin |
Внешняя мембрана Грам-отрицательных бактерий | 25.6 ± 1.4 Å | 9.0 |
| 2UUH | спираль Leukotriene C4 synthase |
Мембрана ЭПР | 29.4 ± 1.1 Å | 19.5 |
| 1xio | спираль Sensory rhodopsin |
Внутренняя мембрана Грам-отрицательных бактерий | 31.9 ± 1.5 Å | 23.0 |
| 1m0l | спираль Bacteriorhodopsin |
архебактериальная мембрана | 31.8 ± 1.1 Å | 23.0 |
Таблица 1. Описание трансмембранных белков с известной 3D структурой.
У трансмембранных бета-баррелей толщина гидрофобной части и число остатков в трансмембранном участке в целом значительно меньше, чем у трансмембранных альфа-спиралей. Это связано с тем, что число остатков в альфа-спиралях имеет минимальный порог для организации и поддержания структуры.
Отбор гомологов белка из pdb: 3ZE3
Был произведен поиск гомологов при помощи сервиса blastp по базе refseq с e-value 1. После этого из 3002 результатов отобрано 10 гомологов по организмам, наиболее далеким от исходного и\или максимально характеризующим филогенетические группы к которым принадлежат. Отдельно следует уточнить, что судя по результатам blastp последовательность белка из pdb 3ZE3 очень (действительно очень) консервативна, из-за чего и возможет выбор самых далеких из известных гомологов для создания репрезентативной выборки: они являются достаточно далекими для внесения информации о вариативности в последовательности, но в то же всемя остаются достаточно близкими для видимой гомологии. Резульат в fasta-формате.Иллюстрация филогенетического дерева некоторых организмов по данным сервиса EMBL представлена на Рис.1
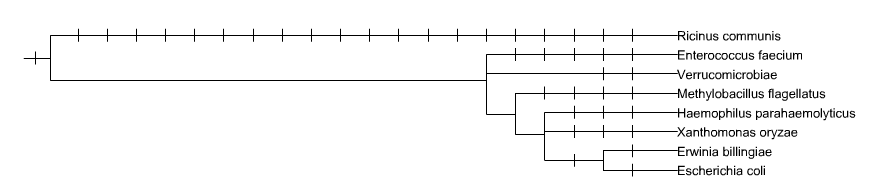
Рисунок 1. Филогенетическое дерево 8 из 10, чьи гомологи используются в анализе. (промежуточные такосномические уровни свернуты,т.е показаны чертой) Дерево построено по данным сервиса EMBL
Основная информация о белке KDGL_ECOLI из pdb: 3ZE5
На основе данных из OPM получено описание белка, представленное в Таблице 2. (рассмотрена структура 3ZE5, т.к. 3ZE3 не представлено в OPM).
| PDB ID | организм | Тип мембраны | TC-код | Угол наклона спиралей (β-тяжей) к нормали | Количество трансмембранных спиралей (β-тяжей в бочонке) |
| 3ZE3 | Escherichia coli | внутренняя | нет | 2 ± 1° (в цепи А: 4°) | 9 ( по 3 в каждой цепи) |
Таблица 2.Описание структуры трансмембранного белка KDGL_ECOLI (идентификатор PDB 3ZE5, цепь A).
Анализ множественного выравнивания трансмембранных белков
Для полученных гомологов проведено множественное выравнивание при помощи сервиса t-coffee, резульат выравнивания в fasta-формате.
Было произведено предсказание трансмембранных спиралей для гомологичного белка из организма Salmanela bongori при помощи сервиса TMHMM Результат представлен в Таблице на Рис.2 и на Рис.3. Множественное выравнивание предсталвено на Рис.3 со схемой расскарски по гидрофобности (Hydrophobicity: гидрофобные остатки показаны красным цветом) с интенсивностью по консервативности позиции (порог: 30%). Дополнительно в полях TM_REAL и TM_PREDICTED буквой М отмечены трансмембранные участи реальные у E.coli и предстказанные у S.bongori соответственно. Трехмерная структура белка KDGL_ECOLI представлена на Рис.4, раскрашена цепь B, схема раскраски соответствует Рис.3, часть белка ориентированая в n-сторону мембраны (в сторону цитоплазмы) расположена сверху.
# gi|445925046|Salmonella Length: 122 # gi|445925046|Salmonella Number of predicted TMHs: 2 # gi|445925046|Salmonella Exp number of AAs in TMHs: 49.4591 # gi|445925046|Salmonella Exp number, first 60 AAs: 10.34369 # gi|445925046|Salmonella Total prob of N-in: 0.84048 # gi|445925046|Salmonella POSSIBLE N-term signal sequence gi|445925046|Salmonella TMHMM2.0 inside 1 56 gi|445925046|Salmonella TMHMM2.0 TMhelix 57 79 gi|445925046|Salmonella TMHMM2.0 outside 80 98 gi|445925046|Salmonella TMHMM2.0 TMhelix 99 121 gi|445925046|Salmonella TMHMM2.0 inside 122 122
Таблица 3. Вывод программы TMHMM для предсказания трансмембранной организации белка gi:445925046 из Salmonella bongori.
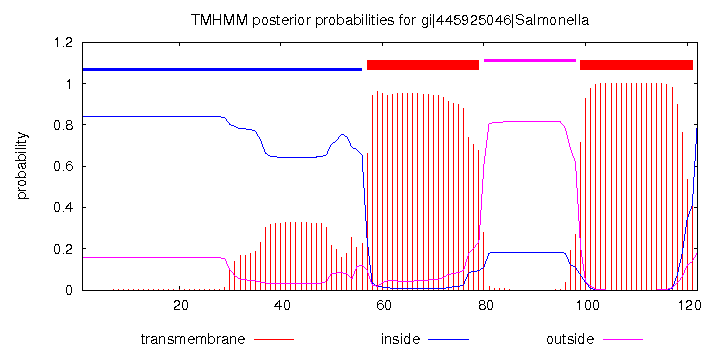
Рисунок 2. Результат программы TMHMM для предсказания трансмембранной организации белка gi:445925046 из Salmonella bongori.
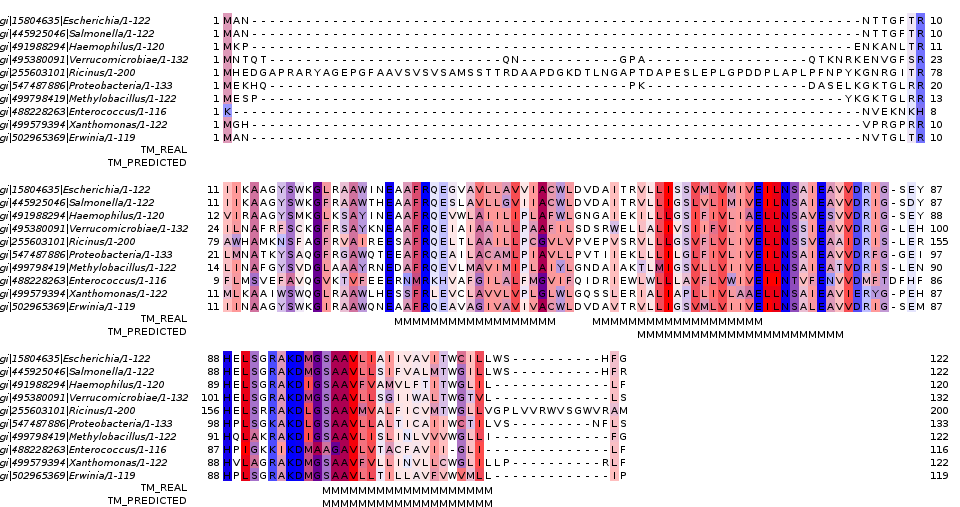
Рисунок 3. Результат выравнивание гомологичных послдеовательностей ( визуализация средствами Jalview). Цветовая схема: по гидрофобности, интенсивность по консервативности (порог: 30%). В полях TM_REAL и TM_PREDICTED буквой М отмечены трансмембранные участи реальные у E.coli и предстказанные у S.bongori соответственно.
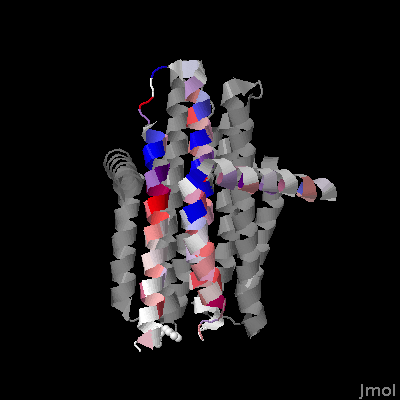
Рисунок 4. Трехмерная структура белка KDGL_ECOLI , раскрашена цепь B, схема раскраски соответствует Рис.3, часть белка ориентированая в n-сторону мембраны (в сторону цитоплазмы) расположена сверху. (Визуализация выполнена в программе Jmol)
Как видно на Рис.3 участки, относящиеся к трансмембранным спиралям имеют явно консервативные позиции и качественно похожее ( по гидрофобности) окружение. В этих участках часто встречаются аминокислоты: валин, аланин, лейцин, а так же серин. В межспиральном участке наблюдаются как консервативные, так и не консерванитвные участки. В самих спиралях при этом есть консервативные заряженные остатки (аргинин), которые возможно выполняют какую-либо функциональную роль.
При этом наблюдается совпадение предсказания TMHMM и реальной трансмембранной организации, там где оно произошло. Один из участков предсказан полностью, другой не полно, третий не предсказан совсем. Возможно третий участкок не достаточно явно функционально выражен для надлежащего предсказания.
Увеличим клетку до размеров дыни. С чем можно сравнить толщину мембраны?
Дыни имеют размер от 18-20 см в длину около 14 см в диаметре. Опишем дыню, как шар шар диаметра 16 см. Для сравнения возьмем среднюю эукариотическую клетку: её размер - 10-50 мкм. Положим размер равным 30 мкм. Толщина мембраны составляет - 7-8 нм. Тогда толщина мембраны клетки размера дыни будет: (7.5*10^(-9) * 16*10^(-2))/30*10^(-6) м = 4*10-5 м. Мембрану такой толщины можно сравнить со слоем толщиной в одну среднюю эукариотическую клетку.
Проверка правила "positive inside" для трансмембранных белков
Для белка KDGL_ECOLI проверку правила positive inside для трансмембранных белков (часть белка, расположенная с n-стороны от мембраны, содержит больше положительно заряженных остатков) можно сделать по выравниванию, представленному на Рис.3, т.к. организация структуры выглядит так: всё что не трансмембраннно, находится с n-стороны. Как видно из Рис.3, положительно заряженные аминокислоты встречаются на протяжении всей цепи белка, но все же их больше не в трансмембранной зоне,т.е. согласно рассматриваемой структуре (3ZE5) их больше с n-стороны от мембраны. Получается для данного белка правило выполняется.
Проверка работы сервиса, предсказывающего ТМ бета-баррель по последовательности
Для предсказания исползован сервис PRED-TMBB. Для работы выбран белок OprH (pdb 2lhf) из Pseudomonas aeruginosa. Средствами ПО blast найден гомолог из Pseudomonas stutzeri: мембранный белок ref|YP_001170938.1. Далее сервисом PRED-TMBB проведено предсказание для обоих белков. Как видно из Рис.5 и Рис.6 сервис не очень точно предсказал организацию Oprh (перепредсказал некоторые участки, и сдвинул трансмембранные области по сравнению с известной сруктурой) при этом у гомолога участки предсказаны сравнительно верно, но довольно скудно.
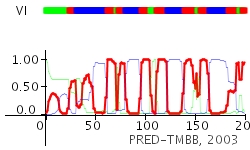
Рисунок 5. Результат предсказания трансмембранной организации белка Oprh (pdb 2lhf) сервисом PRED-TMBB
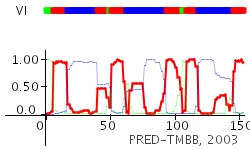
Рисунок 6. Результат предсказания трансмембранной организации белка гомологичного Oprh сервисом PRED-TMBB
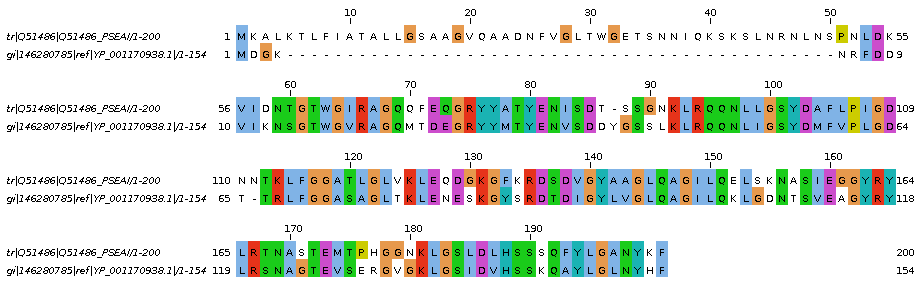
Рисунок 7. Результат выравеиваеия белка Oprh и его гомолога сервисом t-coffee. Визуализация Jalview, цветовая схема clustalx
Проверка гипотезы, что остатки бета-барреля, обращенные в мембрану, гидрофобны
Для работы выбран белок OprH (pdb 2lhf) из Pseudomonas aeruginosa. При помощи сервиса Sheep оплучена карта расположения на правления остатков.
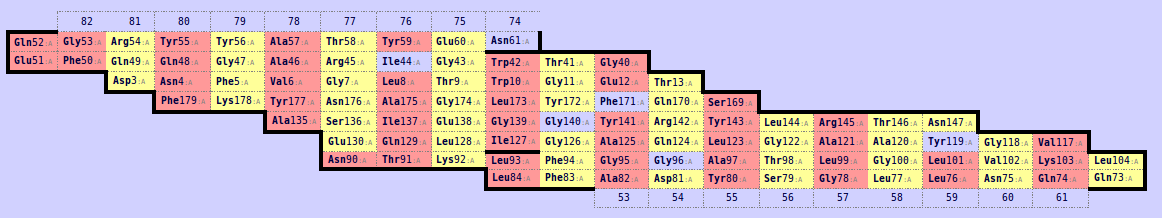
Рисунок 8. Карта остатков цепи белка из PDB 2lhf, полученная в SheeP (SheeP sheet map). Красным цветом выделены остатки, боковые цепи которых "смотрят" наружу баррели, жёлтым – внутрь.
Как видно из Рис.8, в целом, гипотеза верна: остати бата-барреля, обращеные в мембрану, гидрофобны. При этом наблюдаютя некоторые исключения, возможно вызванные функциональной особенностью.
