d1: определение вторичной структуры
Сравнение предсказания
Для определения вторичной структуры были использованы серверные версии программ DSSP и Stride на структуре белка PDBid 2Z3B, результат для
 DSSP и
DSSP и  Stride.
Stride.
Запуск DSSP
mkdssp -i 2Z3B.pdb -o 2Z3B.dssp
Запуск Stirde
stride 2Z3B.pdb -f2Z3B.stride -h
По результатам запуска программ получено сравнение их предсказания с данными, представленными в PDB-файле, представленное в Таблице 1.
| Элемент | PDB | DSSP | Stride | ||||
|---|---|---|---|---|---|---|---|
| Начало | Конец | Начало | Конец | Начало | Конец | ||
| 1 | Тяж | 8 | 13 | 8 | 13 | 7 | 13 |
| 2 | Тяж | 48 | 52 | 48 | 52 | 48 | 52 |
| 3 | Спираль | 55 | 73 | 56 | 72 | 56 | 73 |
| 4 | Спираль | 76 | 91 | 77 | 90 | 77 | 90 |
Таблица 1. Сравнение разметки вторичной структуры полученной различными способами. Координаты даны для цепи А белка.
Как видно из Таблицы 1 предсказание вторичной структуры, в целом, схожы с записью в pdb, но существуют небольшие разлиция на концах разметки.
Карта β-листов
С помощью программы SheeP была построена карта β-листов структуры 2Z3B один из элементов которой представлен на Рисунке 1.

Рисунок 1. Карта одного из β-листов структуры 2Z3B. (Sheet sht_2, map_2)
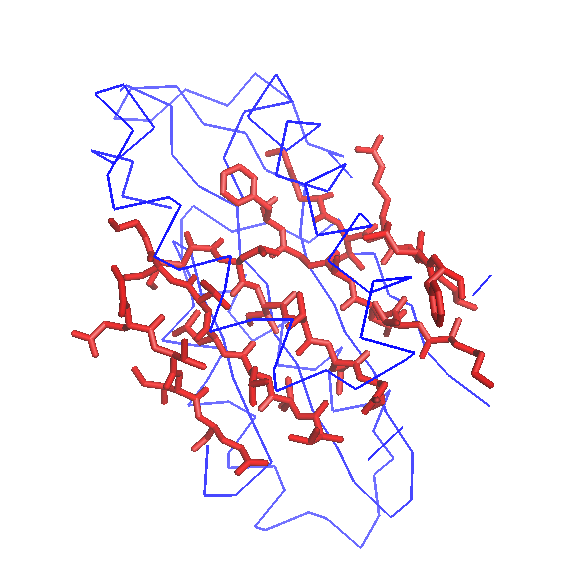
Рисунок 2. Карта одного из β-листов структуры 2Z3B, (Sheet sht_2, map_2). Изображена только цепь А, остов синим, описываемый β-лист -красным.
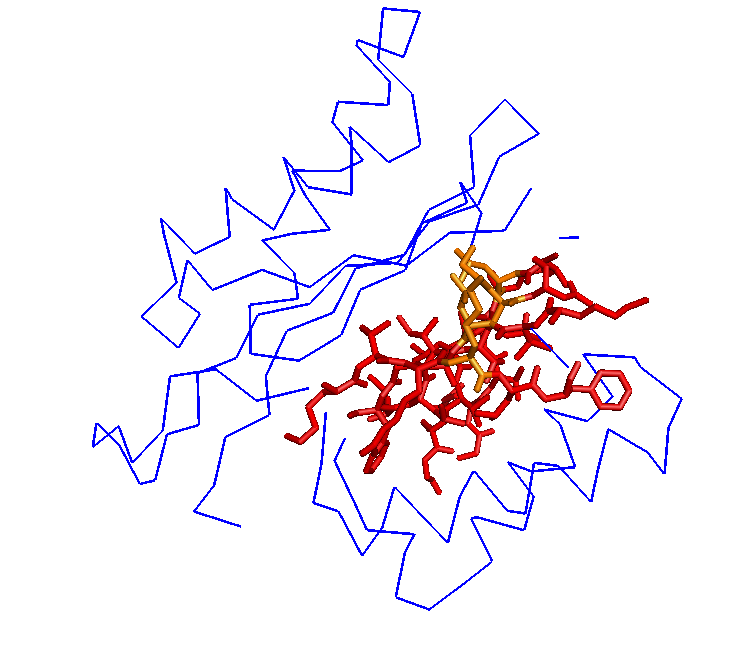
Рисунок 3. Карта одного из β-листов с выделенным гребнем из структуры 2Z3B, (Sheet sht_2, map_2). Изображена только цепь А, остов синим, описываемый β-лист -красным, описываемый гребень (образованный Lys42, Leu49, Met104, Leu109) -- оранжевым.
Как видно из Рисунка 1-3, найденный β-лист совпадает со структурой, а рассмотреннный β-тяж действительно формирует β-тяж, а так же направлен внутрь белка, в гидрофобную область.
Карта водородных связей
Для β-листа построена карта (Рисунок 4) и карта водородных связей.

Рисунок 4. Карта одного из β-листов, для которого показаны водородные связи ниже.
Водородные связи в β-листе
N[ile175:A]O N[leu176:A]O N[glu177:A]O N[glu178:A]O N[leu179:A]O
| | | | |
O[gly21 :A]N O[ser20 :A]N O[met19 :A]N O[ala18 :A]N O[ser17 :A]N O[arg16:A]N
| | | | | |
N[thr7 :A]O N[ile8 :A]O N[phe9 :A]O N[ala10 :A]O N[val11 :A]O N[gln12 :A]O N[his13:A]O
| | | |
O[gly129:A]N O[ile128:A]N O[ala127:A]N O[leu126:A]N O[ile125:A]N
Данная карта содержит нерегулярности (buldes), наблюдаемые, например, в левом верхнем углу карты
d2: Совмещение структур
Построение совмещения структур
Для анализа был выбран белок CLPQ_BACSU
С помощью сервиса PDBeFOLD были найдены структурные гомологи белка, из которых выбрано 4 для дальнейшей работы:
- 2z3a: Q-0.97; RMSD-0.46;Seq%-100
- 3ty6: Q-0.95; RMSD-0.58;Seq%-80
- 1jjw: Q-0.84; RMSD-1.04;Seq%59
- 1g3k: Q-0.84; RMSD-1.12;Seq%59
Поиск структурных гомологов для домена 1dek A:33-154
При помощи сервиса PDBeFold был произведен поиск гомологов для домена 1dek A:33-154,
находки были отсортированы по RMSD.
В результате было найдено 32 хита, среди которых исходного домена не наблюдалось.
Причиной этого может быть слабая чувствительность при параметрах по умолчанию.
На превый взгляд странно, что исходная последовательность не нашлась. Но судя по всему, причина этого в том, что
уровено похожести в 70% ( по умолчанию) слишком велик, т.к. домен, по которому ведётся поиск составляет около
20-30% от всей структуры. Пори таких параметрах исходная структура не проходит фильтрацию.
При запуске с порогом 15% эта структура находится с RMSD 0.00.
Совмещение по заданному выравниванию
Из базы данных SCOP для дальнейшей работы была выбрана структура 1KGC,
для которй были получены PDB-файлы с доменами из  альфа и из
альфа и из
 бета цепи
бета цепи

Рисунок 5. Структура домена Т-клеточного рецептора из цепи альфа (красная) и бета (синяя).
Далее при помощи сервиса SheeP построены карты β-листов для обоих структур (Рисунок 6 и Рисунок 7). Целевой цистеин обозначен бирюзовым.
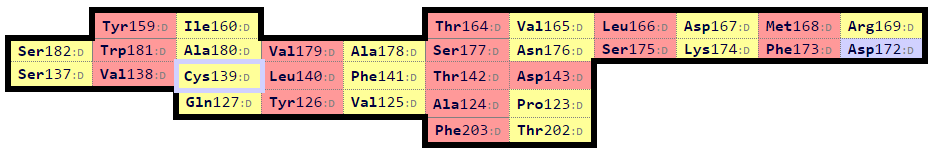
Рисунок 6. Карта β-листа из цепи альфа, полученная Sheep с последующей подстройкой ориентации, соответствующей листу из цепи бета. Консервативный остаток цистеина выделен бирюзовым.
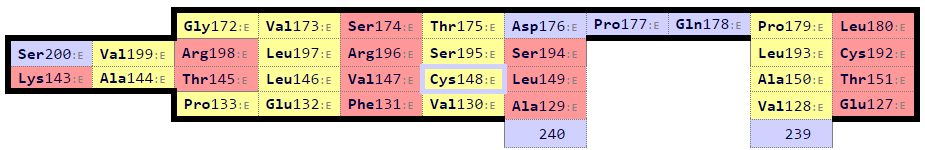
Рисунок 7. Карты β-листов из цепи бета, полученные Sheep. Консервативный остаток цистеина выделен бирюзовым.
Наблюдается соответствие между топологией тяжей β-листа цепи α и цепи β. Выберем остатки для совмещения в PyMol
Запуск DSSP
select alp, chain d and (resi 159-160 or resi 164-166 or resi 179-182 or resi 175-176 or resi 137-140 or resi 126-127 ) and name ca
select bet, chain e and (resi 174-175 or resi 178-180 or resi 194-197 or resi 192-193 or resi 146-149 or resi 129-130) and name ca
pair_fit alp, bet
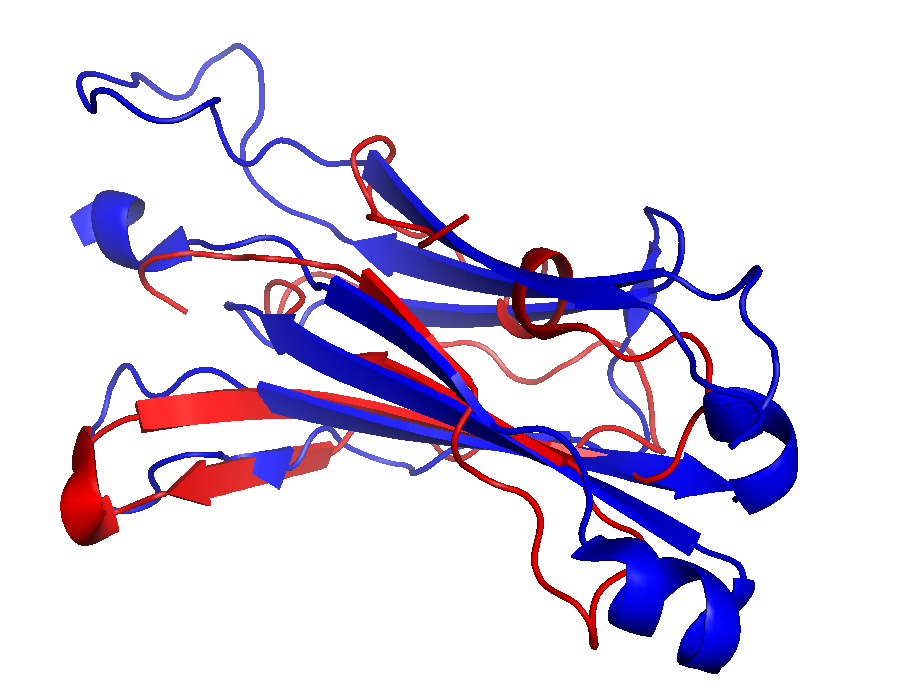
Рисунок 8.Совмещение домена Т-клеточного рецептора из цепи альфа (красная) и бета (синяя) на основе сопоставления отдельных остатков.
Исходя из Рисунка 8, можно утверждать, что топология двух β-листов весьма похожа. Наже не регулярные фрагменты структур схожи между структурами.
d3: Нахождение гидрофобных кластеров
Поиск гидрофобных кластеров в белке 2Z3B
При помощи сервиса CluD при различных параметрах осуществим поиск гидрофобных кластеров. Как видно из Рисунка 9 и 10, при изменеии параметров гидрофобные кластеры могрут разбиватсья на более мелкие или не быть детектированными, но "общая картина" гидрофобных кластеров в структуре остается прежней.
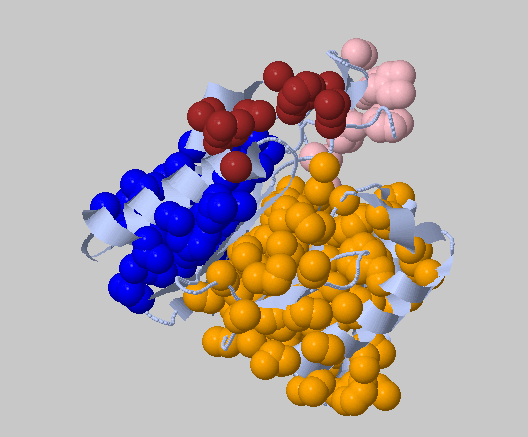
Рисунок 9. Гидрофобные кластеры в А cубъединице структуры 2Z3B. Построено в CluD, Distance threshold - 5.4 Å
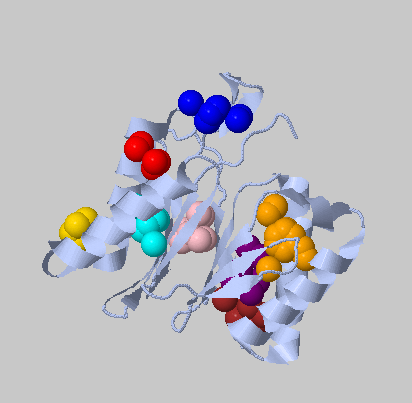
Рисунок 10. Гидрофобные кластеры в А субъединице структуры 2Z3B. Построено в CluD, Distance threshold - 4 Å
Поиск гидрофобных кластеров ДНК-белковом коплексе 3ZQL
Рассмотрим гидрофобные кластеры в ДНК-белковом коплексе 3ZQL, найденные при помощи CluD, Distance threshold - 5.4 Å. Как видно из Рисунка 11,
гидрофобные контакты важны и при взаимодействии белка с ДНК: две цепи белка формируют гидрофобные ядра, вовлеченные в контакт с гидрофобными участками ДНК.
Рисунок 11. Гидрофобные кластеры в структуре 3ZQL. Построено в CluD, Distance threshold - 5.4 Å.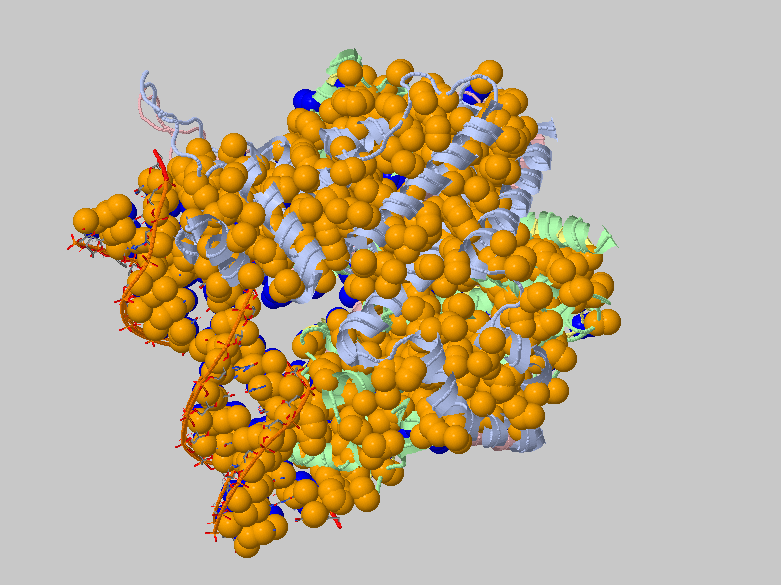
d4: построение поверхности, раскраска участка поверхности (pymol)
Создание изображения для комплекса димера пуринового репрессора с ДНК. Структура 1VPW.

Рисунок 12. Поверхность контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера.
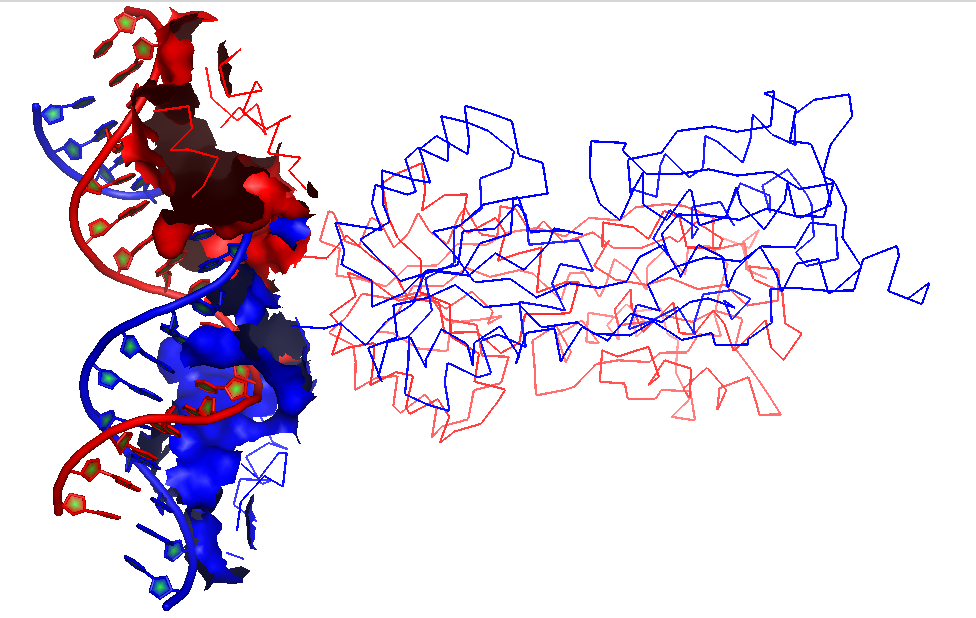
Рисунок 13. Поверхность контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка, вовлечённой в контакт.
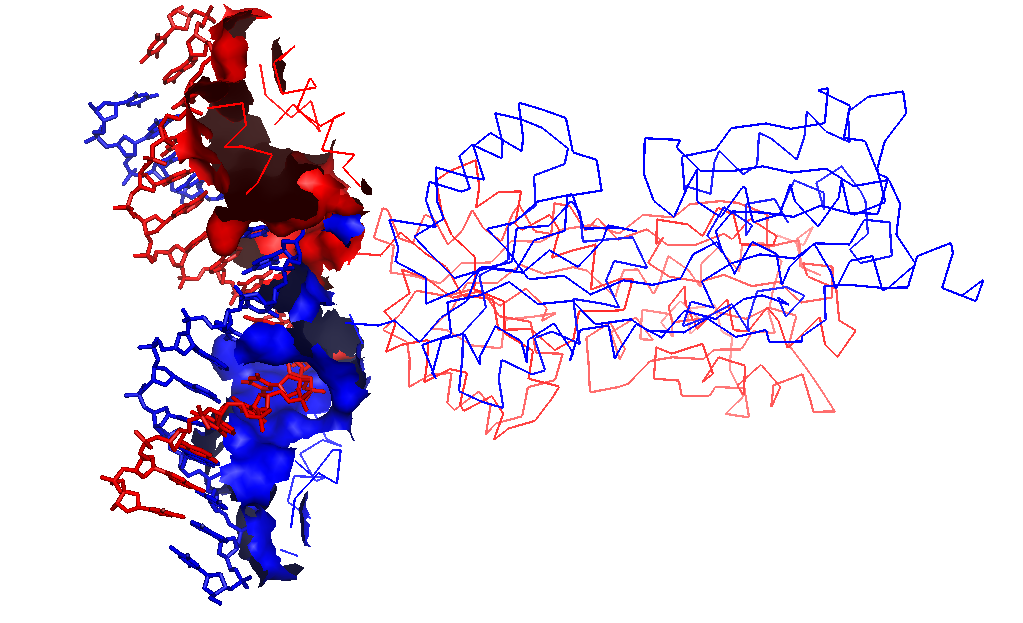
Рисунок 14.Поверхность контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали.
Изображение гидрофобных поверхностей (CluD)
С помощью сервиса Clud был произведён поиск гидрофобных кластеров между мономерами белка (параметры CluD: порог 5.4 Å , более 10 атомов). Гидрофобные кластеры отмечены на изображении поверхности контакта мономеров желтым,см. Рисунок 15.
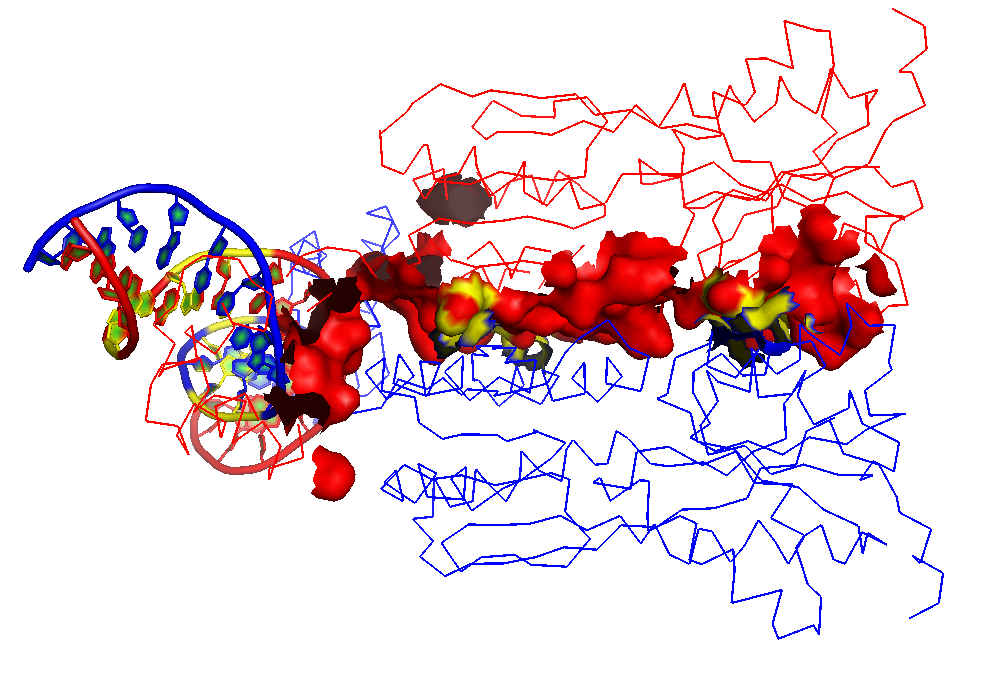
Рисунок 15.Поверхность контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали. Участки гидрофобных кластеров в контакте отмечены желтым.
d5: сравнение доменов SCOP/SCOPe, ECOD, CATH и Pfam
Для работы была выбрана структура 2Z3B, для нее был произведен поиск доменов в Pfam, SCOPe, CATH и ECOD. Результат представлен в Таблице 2.
| База данных | Найденные домены | Границы доменов |
| Pfam | PF00227 | 3-180 |
| SCOPe | d.153.1.4 | 2-181 |
| CATH | 2z3bA00 | 1-180 |
| ECOD | e2z3bA1 | 6-180 |
Таблица 2. Сравнение доменов SCOP/SCOPe, ECOD, CATH и Pfam
Как видно из Таблицы 2, основноой каталитический домен нашелся во всех базах и примерно с одинаковыми координатами.
d6: использование сайта PDB
Скачайте все последовательности белков, структуры которых определенны при помощи метода электронной микроскопии, в виде одного FASTA файла. Используйте Advances search
При помощи Advaneced Search на сайте PDB были скачаны последовательности всех белков,
определенных с помощью метода электронной микроскопии (Experimental Method, ELECTRON MICROSCOPY).
Было определено  924 искомых последовательности.
924 искомых последовательности.
Сравните список структурных гомологов, определенных для вашего белка программой PDBeFold с таким же списком из программы jFATCAT. Последний список есть прямо на странице PDB
Для моего белка 2Z3B на сайте PDB не было найдено структурных гомологов.
На основе похожести по последовательности в 50% предложено смотреть структурные гомологи белка 1G3K.
Для нового белка 1G3K найдено 35 структурных гомологов, определенных программой jFATCAT на сайте PDB.
И 112487 гомологов, определенных с поомощью PDBeFold; при этом многие гомологи являются разными цепями одного белка. Практически все что найдено в jFATCAT найдено и в PDBeFold.
