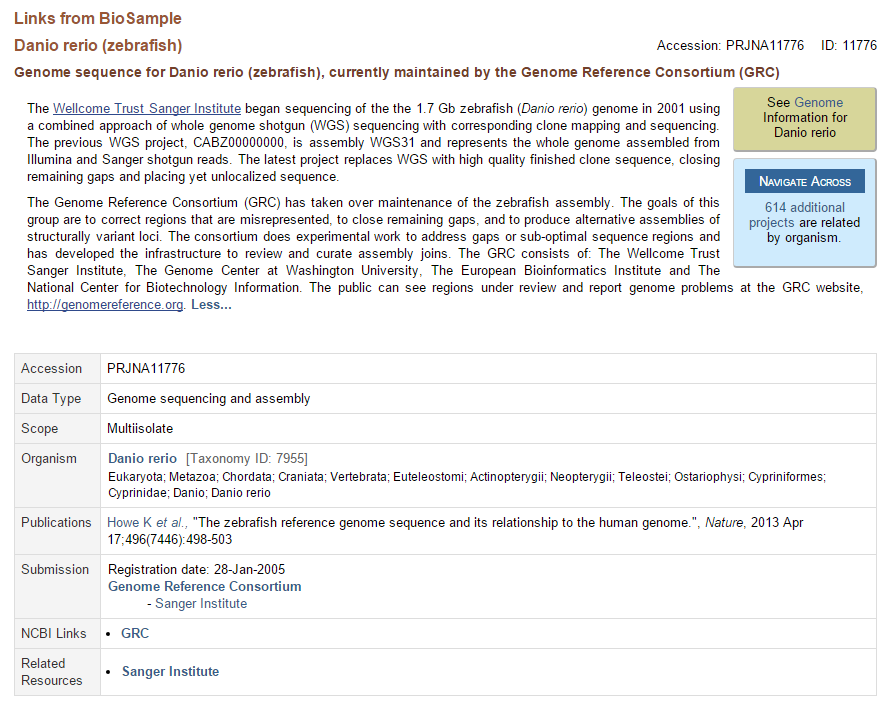
| Ключи, используемые в таблицах особенностей |
| Ключ |
Описание |
Пример |
| CDS |
Кодирующая последовательность; последовательность нуклеотидов, соответствующая аминокислотной последовательности белка |
CDS join(<1..89,193..263,389..420,481..639,698..>736)
/gene="cytC"
/codon_start=1
/product="cytC"
/protein_id="AIJ50606.1"
/db_xref="GI:672718494"
/translation="HTLIFPFLQCPRFTLIGTCHHSLRNCGVFSWYRNRRISSSQFKI
FSNRCLPFPRDAKKGAKLFQTRCAQCHTVESGGPHKVGPNLHGLFGRKTGSAEGYAYT
DANKQAGVTWDENTLFSYLENPKKFIPG" |
| centromere |
Область, где экспериментально была доказана принаждежность к центромере - участку ДНК, где связаны сестринские хроматиды и находится кинетохор |
centromere 555957..556073
/note="CEN16; Chromosome XVI centromere"
/db_xref="SGD:S000006477" |
| D-loop |
Петля смещения - область в митохондриальной ДНК, в которой короткая РНК взаимодействует с одной цепью ДНК, отстраняя комплементарную вторую цепь. Так же описывает замещение одной цепи дуплекса ДНК на другую цепь в реакции, катализируемой белком RecA |
D-loop 15424..16300
/note="control region" |
| intron |
Участок ДНК, который после транскрипции вырезается из транскрипта |
intron <1..>340
/note="J1-C intron" |
| sig_peptide |
Сигнальный пептид на N-конце последовательности |
sig_peptide 23..79
/gene="YM-1" |
| C_region |
Неизменяемый участок лёгких и тяжёлых цепей иммуноглобулинов, а также альфа-, бэта- и гамма-цепей Т-клеточного рецептора |
C_region 394..399
/gene="TCR1A" |
| mat_peptide |
Последовательность зрелого пептида или белка |
mat_peptide 55..399
/gene="TCR1A"
/product="T-cell receptor alpha chain" |
| polyA_site |
Участок, кодирующий те места РНК, куда добавятся остатки аденина вследствие посттранскрипционного полиаденилирования |
polyA_site 173
/gene="294" |
| rep_origin |
Ориджин репликации |
rep_origin 5160..5191
/note="L-strand origin of replication" |
| stem_loop |
Шпилька |
stem_loop 505..520
/gene="H4"
/note="terminator" |