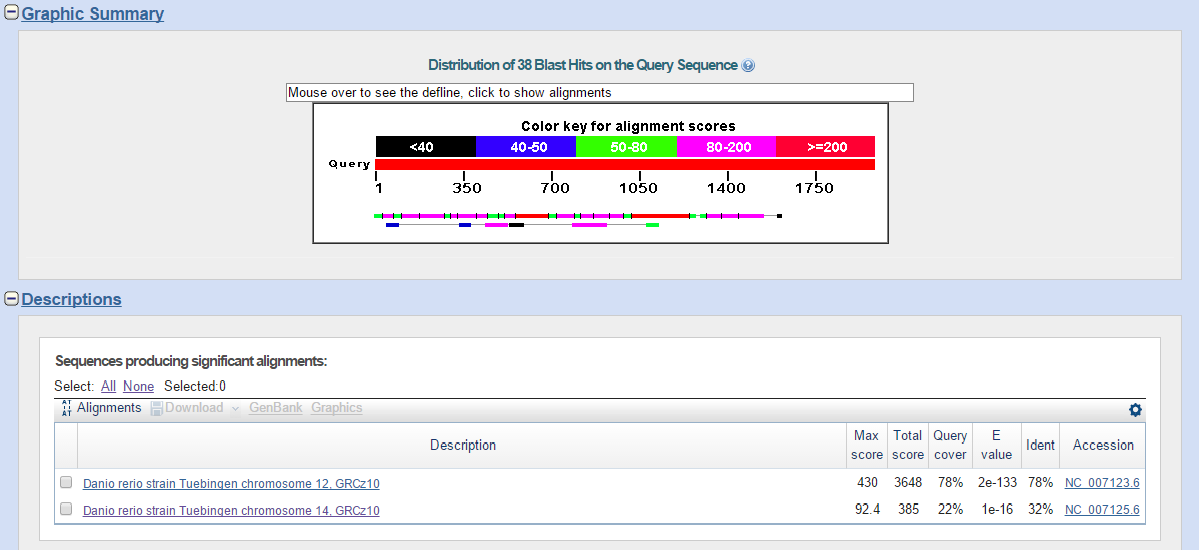
Фрагмент выдачи алгоритма discontiguous megablast
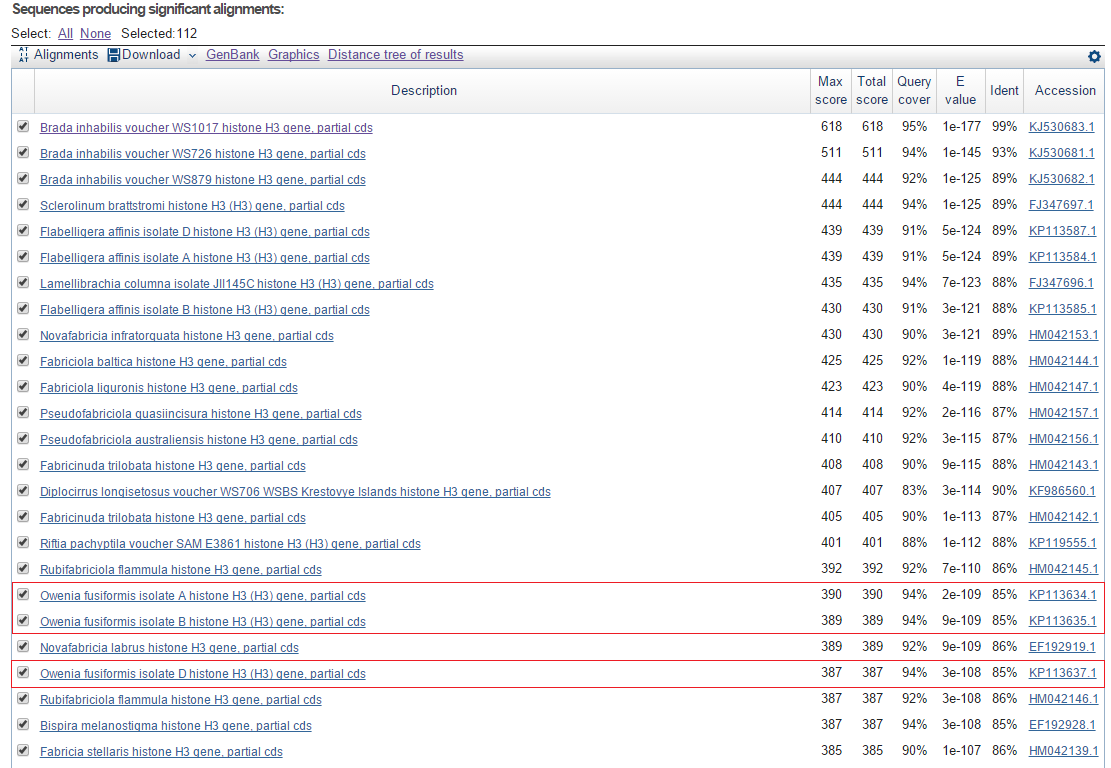
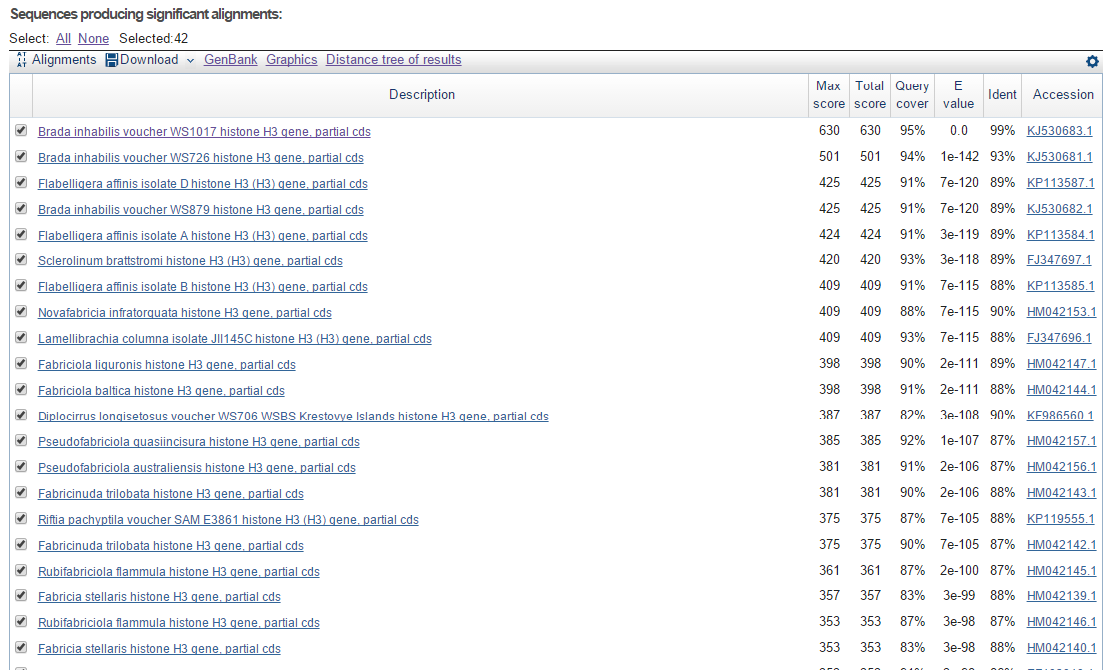
| Алгоритм | blastn | megablast | discontiguous megablast |
| Число находок | 112 | 42 | 112 |
| E-value худшей находки | 6e-54 | 5e-72 | 6e-54 |
| Сходство худшей находки | 81% | 82% | 81% |
| Query cover худшей находки | 59% | 82% | 59% |


| Белок | Число находок (хорошие/все) | Параметры лучшей находки | |||
| Запись | Процент идентичности | Query cover | E-value | ||
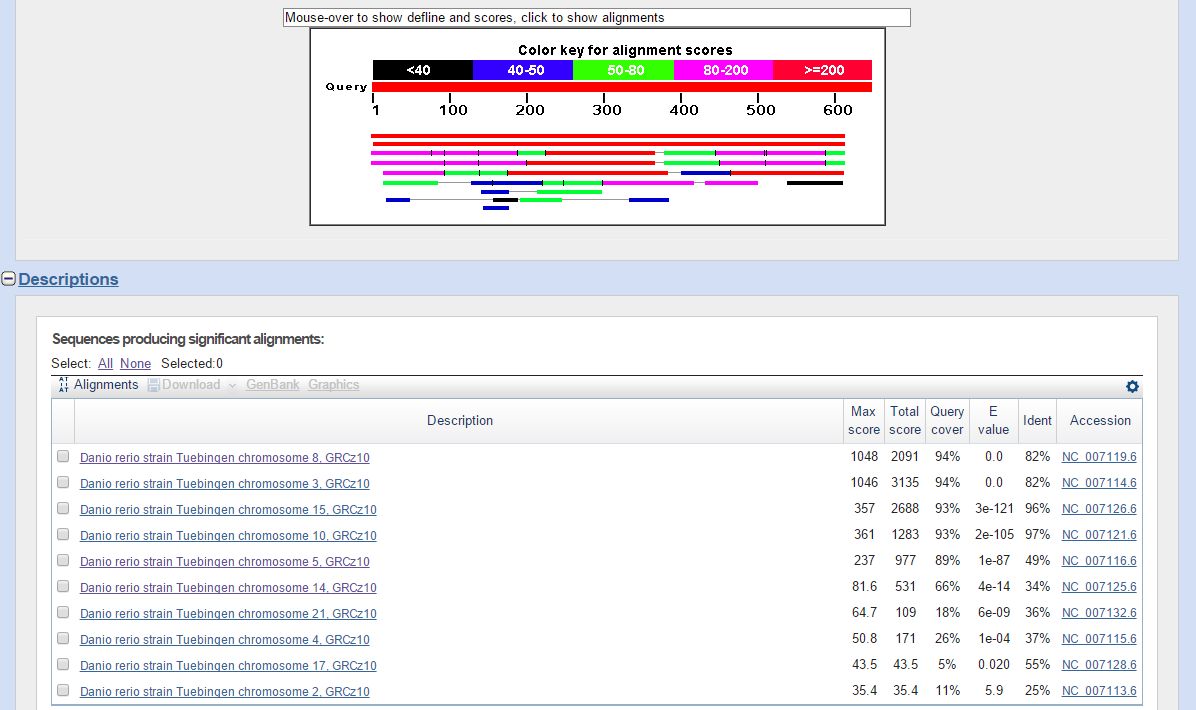
| HSP7C_HUMAN | 5/10 | Danio rerio strain Tuebingen chromosome 8, GRCz10; координаты: 24949043 - 24950878, 4742485 - 4744305 | 82% | 94% | 0.0 |
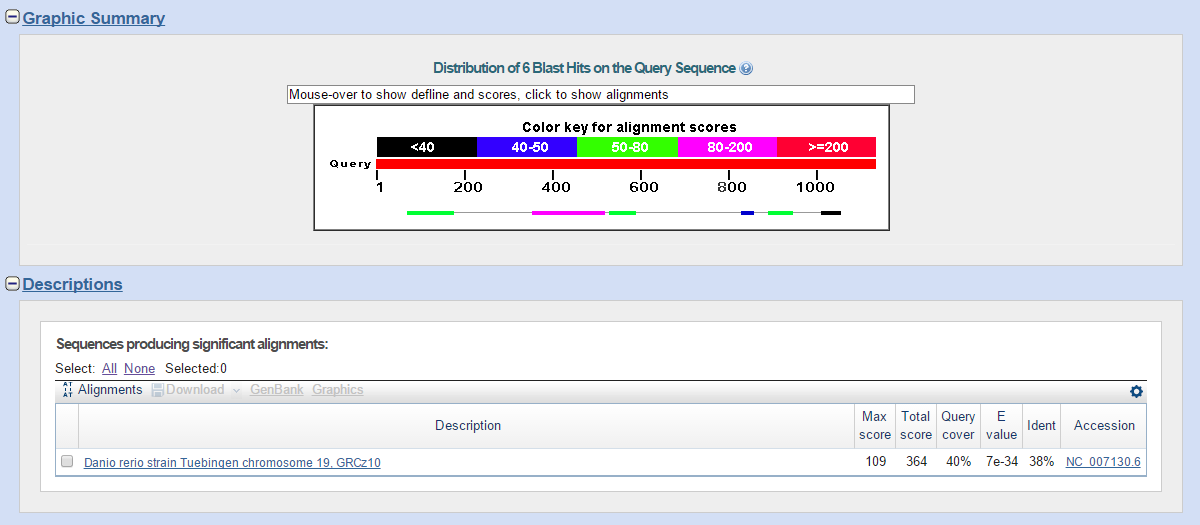
| TERT_HUMAN | 1/1 | Danio rerio strain Tuebingen chromosome 19, GRCz10; координаты: 630351 - 630770, 630880 - 631065, 636583 - 636753, 629478 - 629780, 635562 - 635645, 638725 - 638859 | 38% | 40% | 7e-34 |
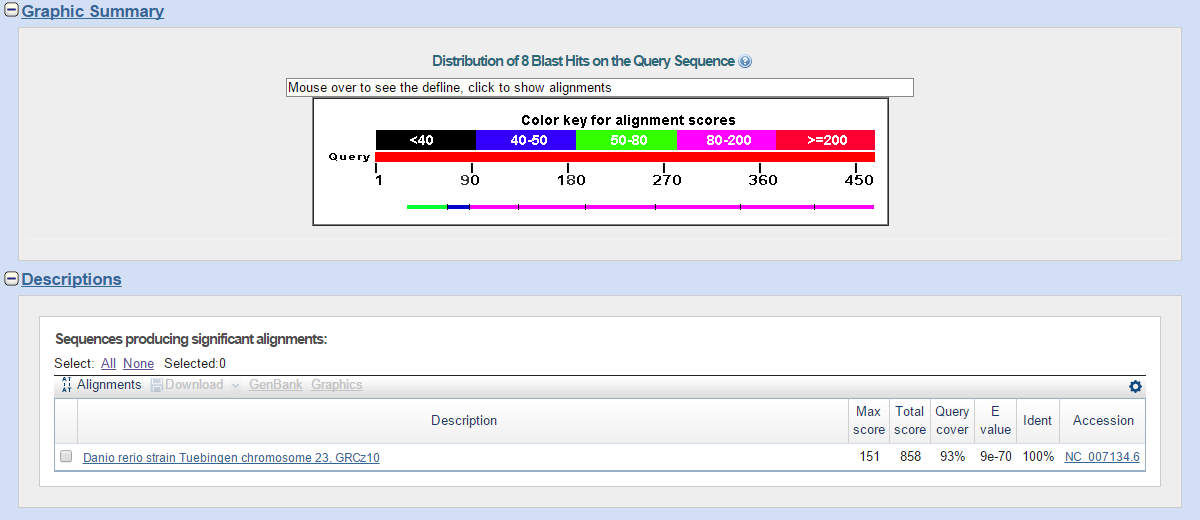
| CISY_HUMAN | 1/1 | Danio rerio strain Tuebingen chromosome 23, GRCz10; координаты: 33997569 - 33997778, 33997150 - 33997491, 33993709 - 33993906, 33991518 - 33991712, 33990663 - 33990797, 33988588 - 33988698, 33990099 - 33990164 | 100% | 93% | 9e-70 |
| RPB1_HUMAN | 1/2 | Danio rerio strain Tuebingen chromosome 12, GRCz10; координаты: 22446298 - 22447125, 22447194 - 22447292, 22448234 - 22448620, 22447948 - 22448154, 22447680 - 22447865, 22443593 - 22444171, 22444453 - 22444671, 22444269 - 22444367, 22442517 - 22442741, 22442798 - 22442962, 22443039 - 22443164, 22441150 - 22441446, 22441535 - 22441702, 22440389 - 22440811, 22444830 - 22445054, 22443364 - 22443576, 22445132 - 22445377, 22445578 - 22445763, 22445862 - 22445969, 22440120 - 22440254, 47781841 - 47782005, 47770395 - 47770529, 47742680 - 47742820, 47754117 - 47754242, 47741809 - 47742084, 47751054 - 47751194, 22447415 - 22447492, 47769749 - 47769880, 22439165 - 22439251, 47732517 - 47732681, 47773321 - 47773449, 22448963 - 22449022 | 78% | 78% | 2e-133 |
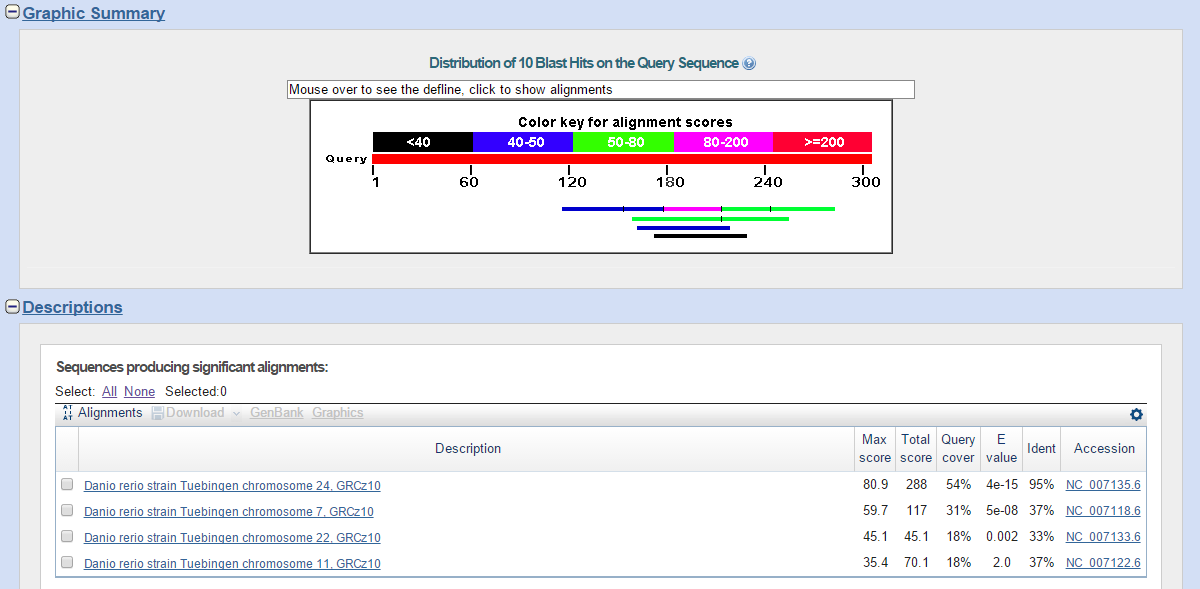
| PABP2_HUMAN | 1/4 | Danio rerio strain Tuebingen chromosome 24, GRCz10; координаты: 14429530 - 14429420, 14428082 - 14427975, 14423934 - 14423809, 14429739 - 14429665, 14431283 - 14431173 | 95% | 54% | 4e-15 |