Структуры нуклеиновых кислот и их комплексов с белками
Предсказание вторичной структуры тРНК
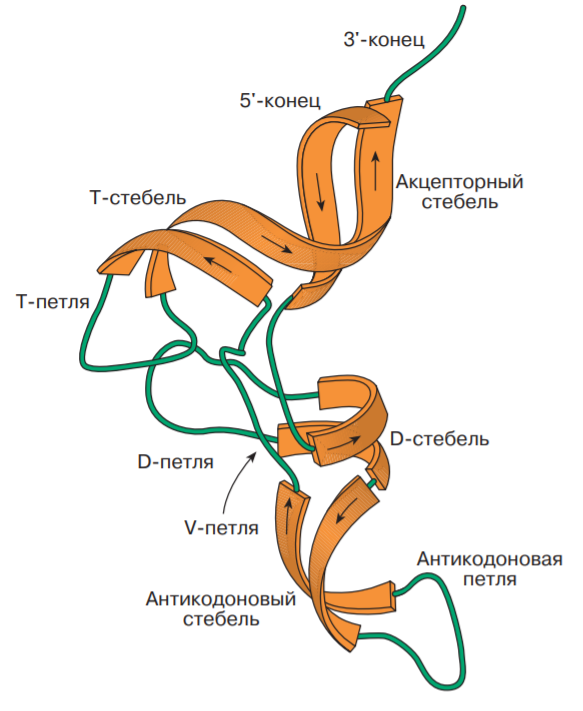
В данной работе продолжилось изучение структуры тРНК (PDB - 2fmt), которая рассматривалась в предыдущем практикуме. С помощью программы einverted из пакета EMBOSS был проанализирован файл 2fmt.fasta, содержащий последовательность тРНК в fasta-формате. Результатом работы программы являлся файл, в котором были указаны инвертированные участки в нуклеотидных последовательностях, а также получено изображение возможных комплементарных участков в последовательности тРНК, при параметре minimum score threshold: 1 и стандартных остальных параметрах. Результаты данного анализа представлены в Таблице 1, для сравнения приведена информация о структуре данной тРНК, полученная с помощью программ find_pair и analyze из пакета EMBOSS (подробную информацию см. в предыдущем практикуме). Ниже на Рис. 1 представлена структура тРНК, которая была покрашена в рамках задач прошлого практикума, и структура тРНК ни Рис. 2, приведенная в статье О. О. Фаворовой на Рис. 3, которая наглядно показывет, расположение различных стеблей. Так, на Рис. 1 оранжевым показан акцепторный стебель, желтым - T-стебель, зелёным - D-стебель, синим - антикодоновый стебель (номера оснований, входящих в тот или иной стебель можно уточнить в предыдущем практикуме).
 |
 |
Помимо этого, предсказание вторичной структуры тРНК было выполнено по алгоритму Зукера с помощью программы RNAfold из пакета Viena RNA Package. На Рис. 3 представлен результат работы данной программы, причем цветом показана вероятность возникновения комплементарных пар по градуировочной шкале, приведённой на Рис. 3, где фиолетовый - наименее вероятная пара, а красный - наиболее. Чтобы понять положение стеблей в тРНК, можно воспользоваться Рис. 2, приведенным в статье О. О. Фаворовой.
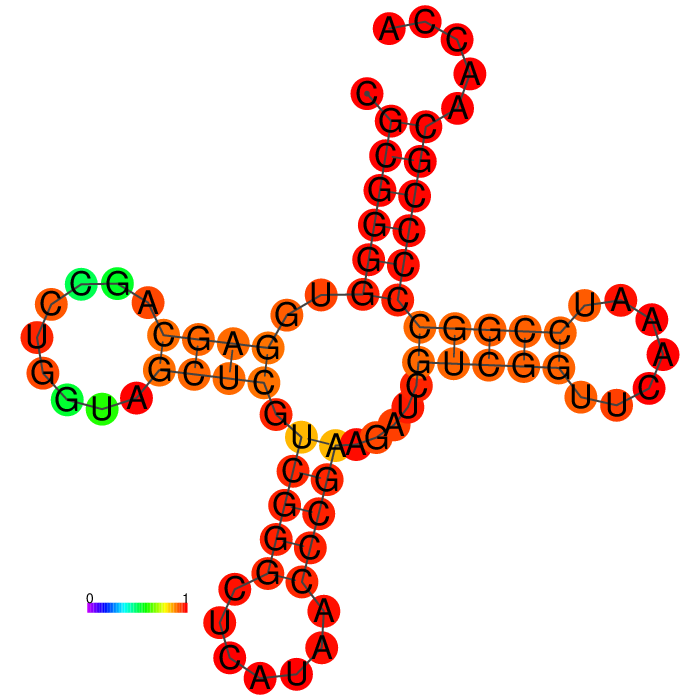 |
Затем Таблица 1 была дополнена информацией о комплементарных парах, полученных с помощью предсказания их алгоритмом Зукера. Различие в количестве канонических пар обусловлено тем, что в результате работы программ find_pair и analyze в полученном файле был отражен модифицировнный цитозин (c[OMC]:32), а с помощью алгоритма Зукера этого не было отражено.
Поиск ДНК-белковых контактов в заданной структуре
Для изучения контактов между молекулой ДНК и белком был рассмотрен комплекс рестрикционного фермента BsobI с молекулой ДНК (PDB = 1dc1), который изучался уже в предыдущем практикуме. В программе JMol была визуализировна молекула ДНК, а также следующие её компоненты:
множество атомов кислорода 2'-дезоксирибозы
множество атомов кислорода в остатке фосфорной кислоты
множество атомов азота в азотистых основаниях
Результат этой работы можно посмотреть на апплете ниже: для включения скрипта нажмите Start script, для переключения между сценариями - Resume. Под апплетом можно ознакомиться со скриптом, по которому воспроизводиться визуализация.
Помимо этого, в Таблицу 2 были сведены данные о количестве некоторых ДНК-белковых контактов. Расмматривались полярные и неполярные контакты между молекулой нуклеиновой кислоты и белка, причем полярными считались атомы кислорода и азота, а неполярными - фосфора, углерода и серы. Под "полярным контактом" подразумевалась ситуация, когда полярные атомы ДНК и полярные же атомы белка находились на расстоянии 3.5 (Å) и менее. Под "неполярным контактом" - расположение неполярных атомов нуклеиновой кислоты и неполярных атомов белка на расстоянии 4.5 (Å) и менее. Однако в результате анализа было выявлено, что атомы фосфора присутствуют только в молекуле ДНК, поэтому в сравнении расстояния до них от ДНК не было необходимости. Для выполнения этого задания был написан скрипт, доступный по этой ссылке. Координаты атомов аденина, тимина и гуанина, принадлежащих малой и большой бороздок были взяты из практикумов однокурсников, доступных по ссылкам: аденин, гуанин, тимин - а принадлежность атомов цитозина малой и большой бороздкам взяты из предыдущего практикума.
Как видно из Таблицы 2, наибольшее количество контактов наблюдается между остатками 2'-дезоксирибозы (90) и на 19 меньше демонстрируют остатки фосфорной кислоты, в то время как остатки азотитых оснований малой и большой бороздок вместе образуют только 42 контакта. Из этого следует, что наибольшему взаимодействию с молекулой белка подвергается именно сахаро-форсфатный остов. В большинстве случаев, число неполярных контактов превалирует, однако для остатков азотистых оснований в большой бороздке ситуация прямо противоположная и чило полярных остатков больше на 2, чем неполярных.
Схемы ДНК-белковых контактов
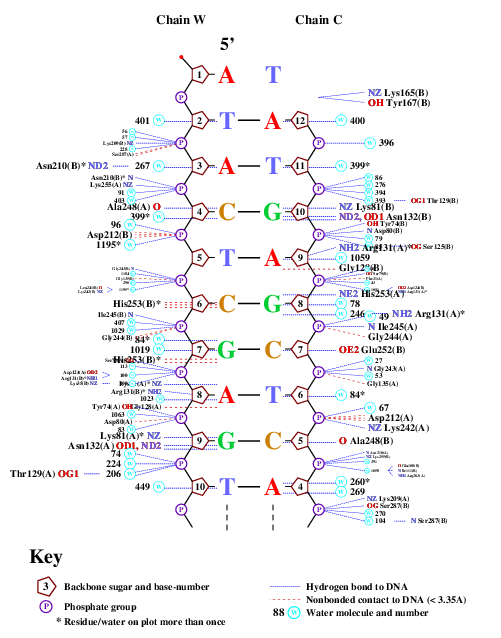
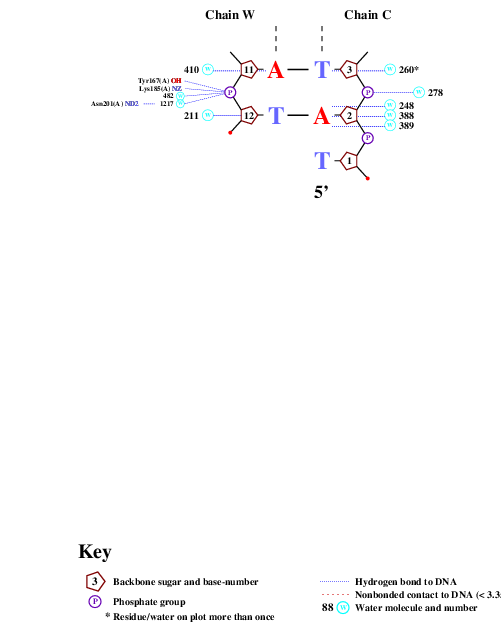
Для получения популярной схемы ДНК-белковых контактов была использована праграмма nucplot из пакета EMBOSS, выдачей данной программы стал файл в ps-формате, конвертированный с помощью специального сайта. На Рис. 4 и 5 представлены изображения ДНК-белковых контактов, на рисунках обозначены цепи ДНК, номера нуклеотидов и аминокислотных остатков, входящи в состав белка, а также синими кругами показаны молекулы воды.
 |
 |
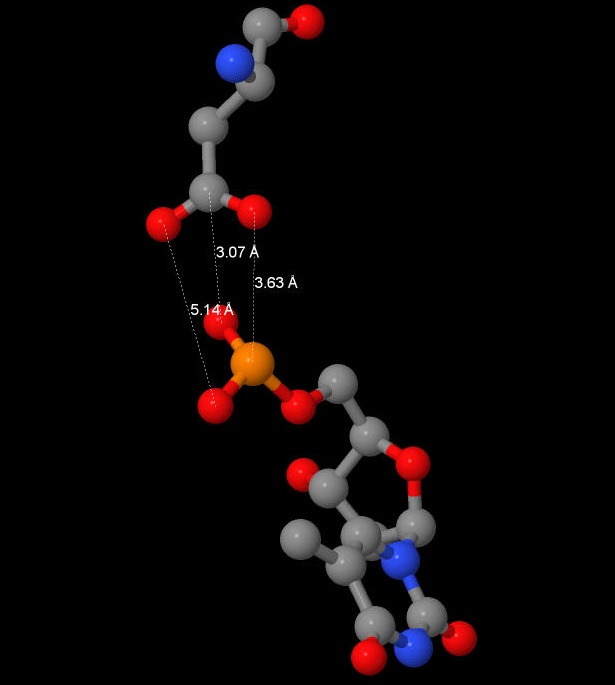
На данной схеме можно увидеть изображения контактов между конкретными аминокислотами и нуклеотидами, как видно из неё, наибольшее количество контактов возникает между молекулой Asp212 и остатком фосфорной кислоты 6 нуклеотида. Данный контакт был визуализирован с помощью программы JMol и изображен на Рис. 6, также на нём показаны наименьшие расстояния между остатком аминокислоты и нуклеиновой кислотой.
 |
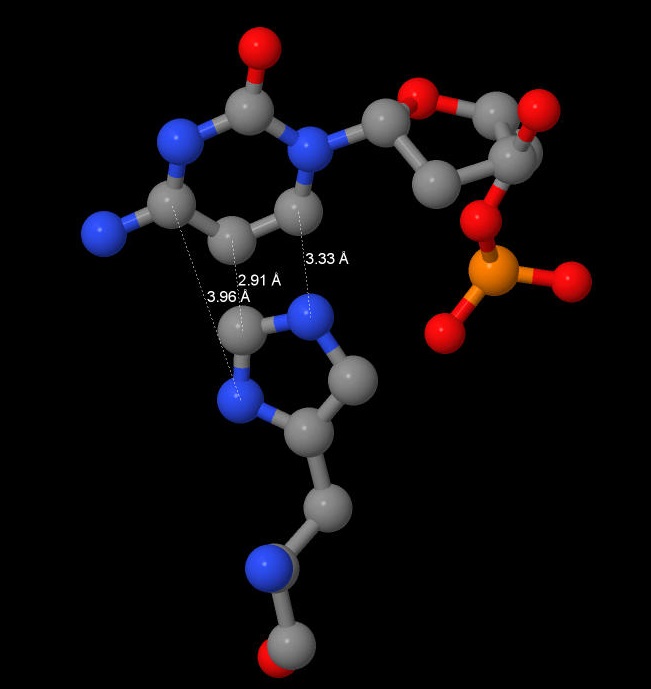
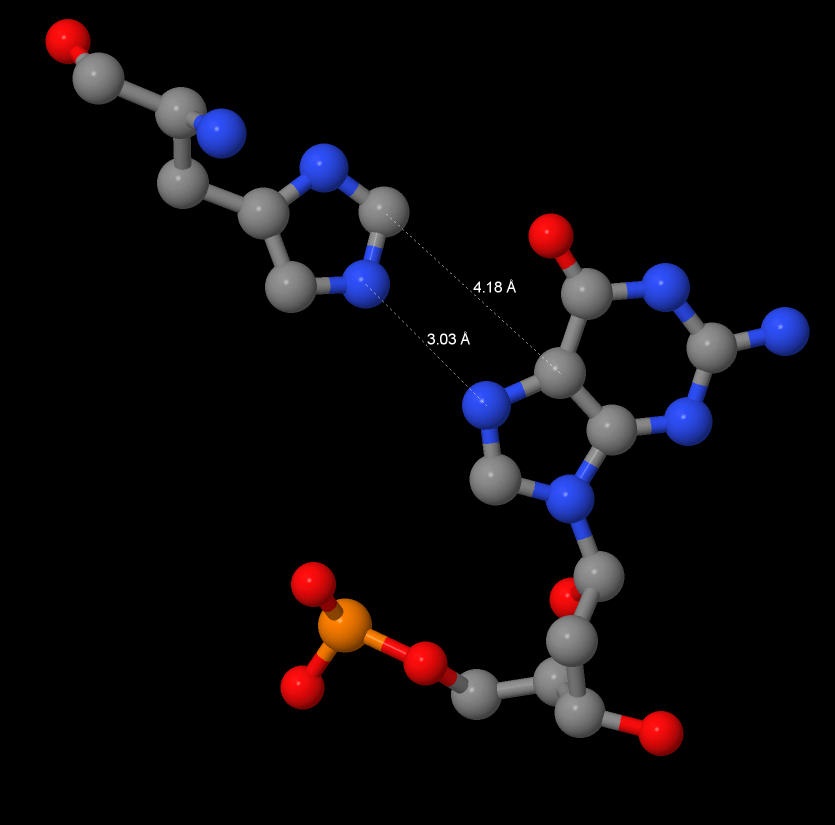
Однако самым важным контактом для данного комплекса является контакт между His253 и нуклеотидами 6 и 7, именно между этими нуклеотидами происходит гидролиз фосфодиэфирной связи, который катализируется эндонуклеазой рестрикции, действующей по сайту 5'-CTCGAG-3' и разрезающей молекулу ДНК в данном месте. На Рис. 7 и 8 показано взаимное расположение His253 с цитозином и с гуанином, также цифрами показано расстояние между атомами.
 |
 |
Источники:
| Статья О. О. Фаворовой |