Учебный сайт Полины Байкузиной | |||
| Главная | Семестры | О себе | Ссылки |
Предсказание генов прокариотЗадание 1. В данном задании требовалось аннотировать последовательность и сравнить с аннотацией генов в записи GenBank. Для работы я выбрала архею Desulfurococcus kamchatkensis 1221n.Таксономия организма представлена на рис.1. Геном археи представлен одной кольцевой молекулой ДНК (CP001140.1). Длина 1365223 п.н. Нуклеотидная последовательность приведена в формате fasta. 
Для получения аннотации последовательности использовался сервер RAST. При помощи RAST была получена исходная таблица аннотаций. Также из NCBI Gene была скачана уже существующая таблица аннотаций. Таблица аннотаций, полученная из NCBI Gene. Всего аннотировано генов в GenBank 1471, в RAST 1563:
Таблица с результатами сравнения аннотаций (желтым выделены аннотации, полученные из GenBank; голубым - при помощи RAST). Примеры генов с несовпадающими аннотациями. Для выбранных генов нужно было проверить аннотацию с помощью blast. 1. Ген уридинмонофосфат-киназы: несовпадение старт-кодонов в аннотациях RAST и GenBank. 
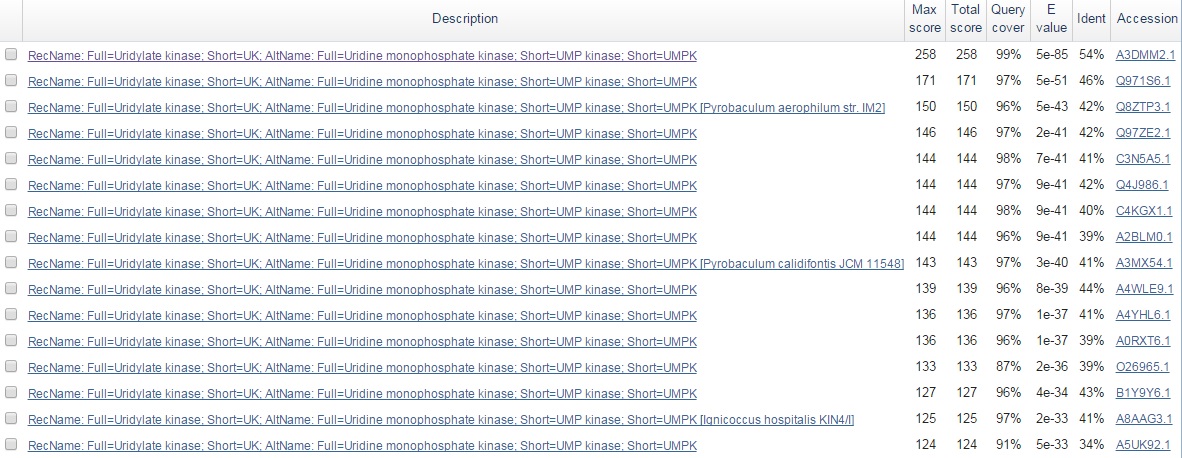
На рис.4 изображено выравнивание последовательности из RAST (лучшая находка), полученное при помощи алгоритма blastp (поиск по Swiss-Prot). Как видно из изображения, старт-кодоны совпадают. Эти данные получены исходя из гомологии, экспериментально подтверждены не были. При запуске blastp для последовательности из GenBank результаты получились хуже. Поэтому можно судить, что аннотация RAST наиболее верна, но все же нельзя говорить об этом с полной уверенностью. 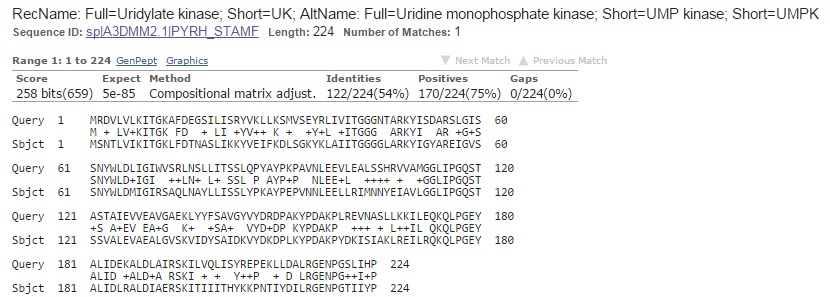
2. Ген фактора инициации трансляции 2: несовпадение старт-кодонов в аннотациях RAST и Genbank. 
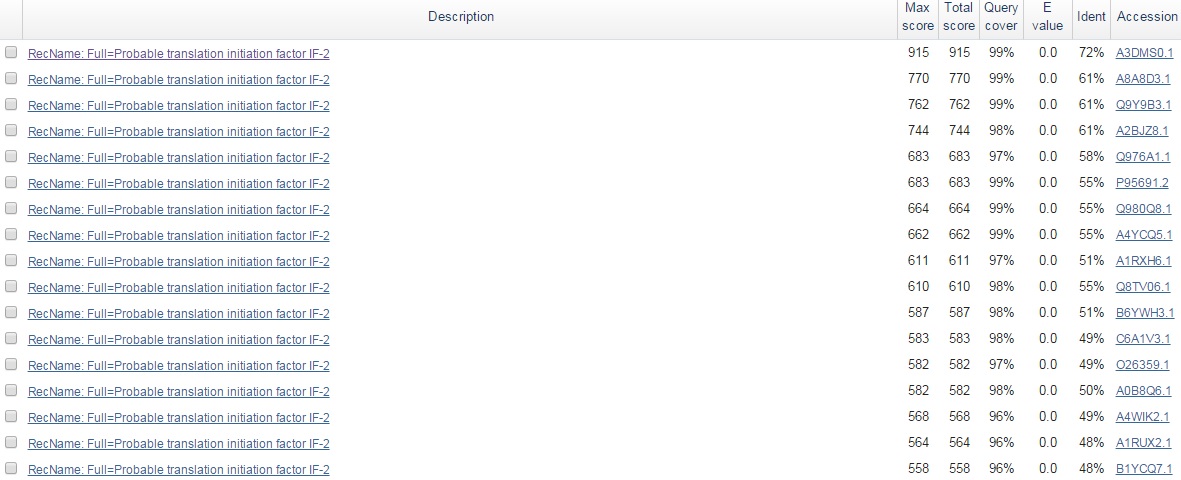
На рис.7 приведено выравнивание последовательности из RAST (лучшая находка), полученное при помощи blastp (поиск по Swiss-Prot). Старт-кодоны совпадают. Данные так же получены исходя из гомологии, экспериментально подтверждены не были. При проверке для последовательности из GenBank результаты получились хуже. Заключить с уверенностью, что аннотация RAST наиболее верна, нельзя. 
3. Расхождение в аннотациях белка. 
Как видно из рис. продукт последовательности с одинаковыми старт- и стоп-кодонами аннотирован по-разному. Для проверки я запустила blastp. Из результатов выдачи алгоритма видно, что лучшие находки являются дезоксирибонуклеазой. Поиск с помощью blast свидетельствует в пользу большей правильности аннотации GenBank. 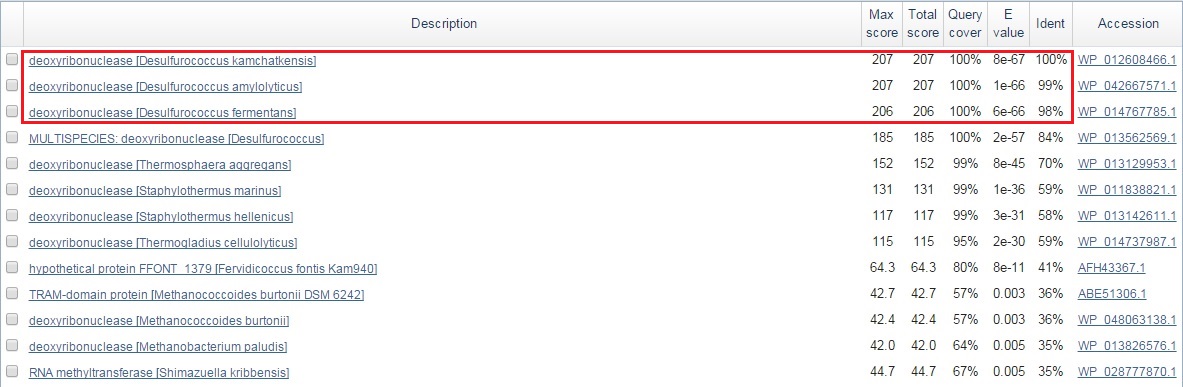
|