Учебный сайт Полины Байкузиной | |||
| Главная | Семестры | О себе | Ссылки |
Предсказание генов эукариотЗадание 1. Предсказание генов X5 с помощью AUGUSTUS. Материал: контиг сборки X5 unplaced-950 (длина: 41249 п.н.). Для предсказания генов был использован сайт AUGUSTUS (AUGUSTUS prediction submission). Затем нужно было выбрать самого близкого родственника из списка организмов, для которых уже было произведено обучение сервиса. Для этого я запустила blastx, который ищет формальные трансляции нашей нуклеотидной последовательности в 6 рамках против базы белков. 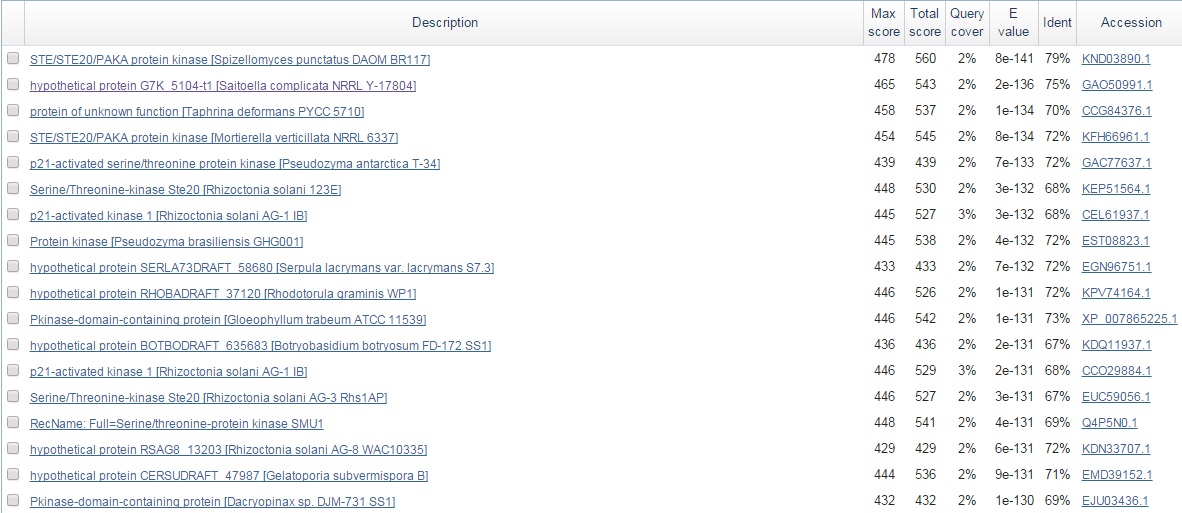
Из результатов выдачи blastx я выбрала организм Saitoella complicata NRRL Y-17804 (Eukaryota; Fungi; Dikarya; Ascomycota; Taphrinomycotina; Taphrinomycotina incertae sedis; Saitoella), т.к. для организма Spizellomyces punctatus DAOM BR117 (Eukaryota; Fungi; Chytridiomycota; Chytridiomycetes; Spizellomycetales; Spizellomycetaceae; Spizellomyces) не был найден близкий родственник в списке организмов. В качестве родственного организма я выбрала Schizosaccharomyces pombe из подотдела Taphrinomycotina. Остальные параметры были оставлены по умолчанию. Адрес страницы с описанием статуса задания: http://bioinf.uni-greifswald.de/webaugustus/prediction/show/0db58f8a5125334801512ff4596d0029 Результат предсказания - архив predictions.tar.gz. В архиве представлены файлы:
Проверка предсказания с помощью BLAST. Программа предсказала 22 белок-кодирующих гена. Список предсказаных генов (таблица). Для проверки экзон-интронной структуры я выбрала 5 генов: g9, g12, g13, g19, g20. Данные о предсказанной экзон-интронной структуре были взяты из файла augustus.gff. Данные для 5 выбранных белков представлены в табл.1. 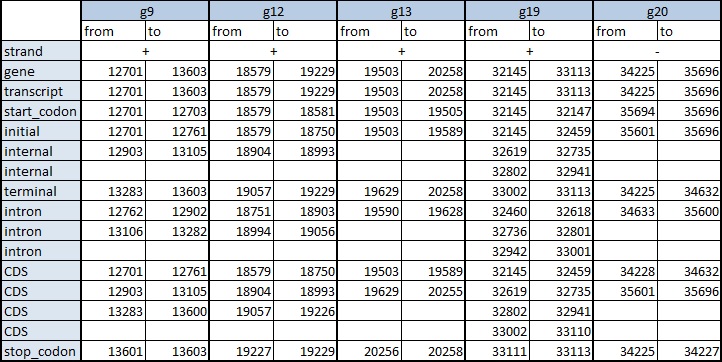
Чтобы проверить экзон-интронную структуру, я использовала blastp (ищет входную последовательность белка в банке белков). Аминокислотные последовательности белков были взяты из файла augustus.aa. g9 Для последовательности g9 я запустила blastp по банку Swiss-Prot, ограничив область поиска таксоном Fungi. 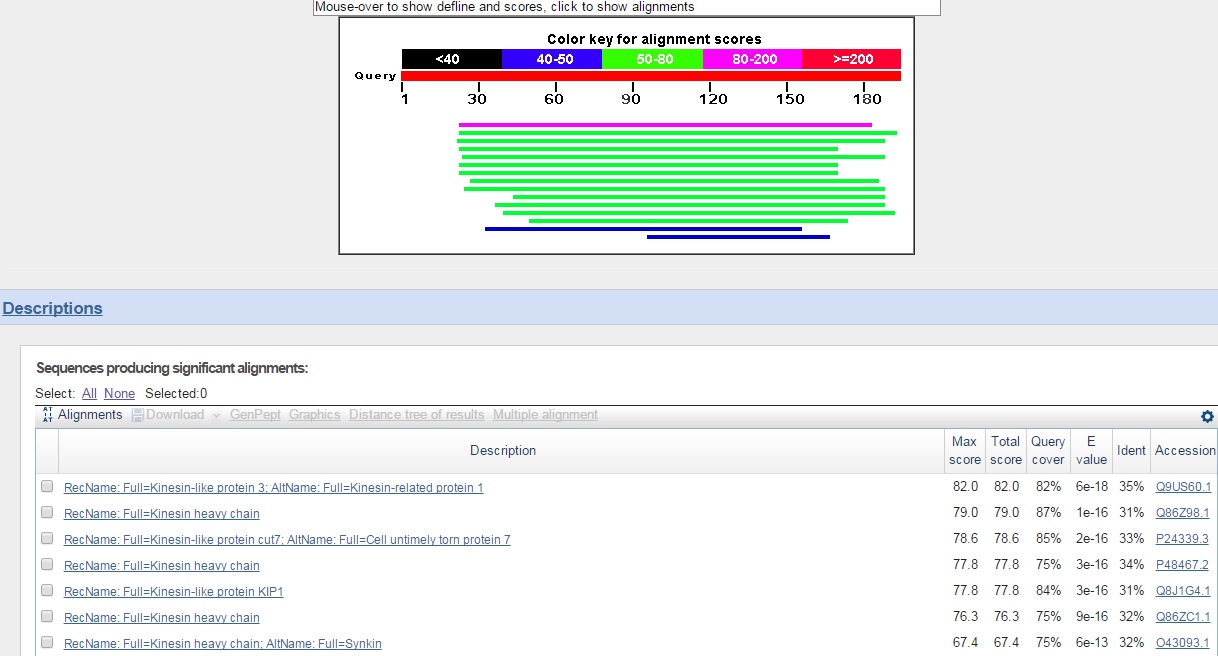
Лучшая находка принадлежит Schizosaccharomyces pombe 972h- (из подотдела Taphrinomycotina, что и исходный организм). У данного организма продукт подтвержден на уровне транскрипта. Также определена функция белка (цитоплазматический мотор, который может играть роль в рециркуляции мембраны аппарата Гольджи) (рис.3). 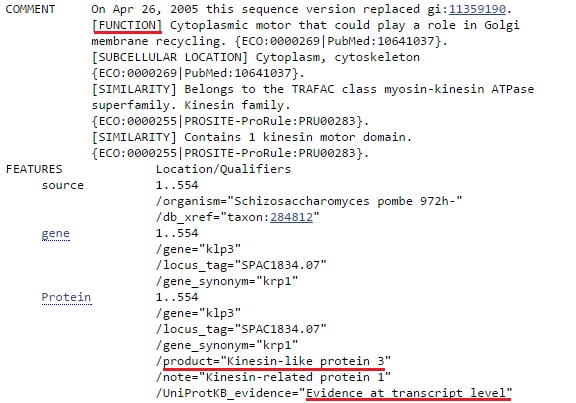
В выравнивании находки (рис.4) с входной последовательностью предсказанного белка есть гэп. Почти в том же месте (с небольшим разбросом) был гэп и у других находок. Это может говорить о том, что AUGUSTUS неправильно определил границы экзона. Возможно, часть интрона была рассмотрена в качестве экзона и транслирована, поэтому с ней нет сходства во всех последовательностях. 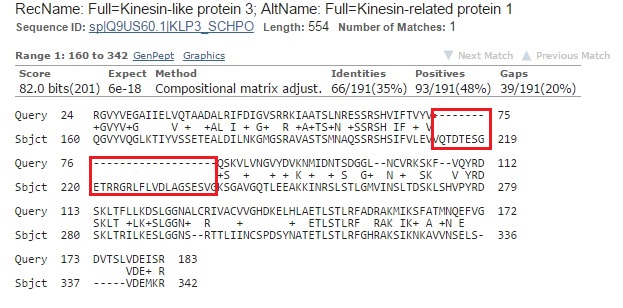
g12 Для последовательности g12 я также запустила blastp по банку Swiss-Prot и ограничила область поиска таксоном Fungi. 
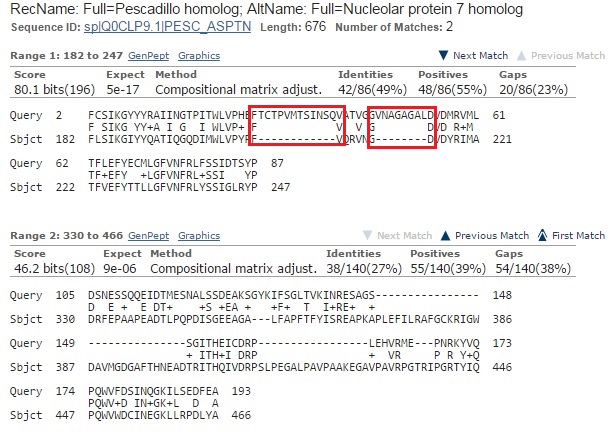
Продукт лучшей находки подтвержден на уровне белка, и определена функция данного белка (рис.6). 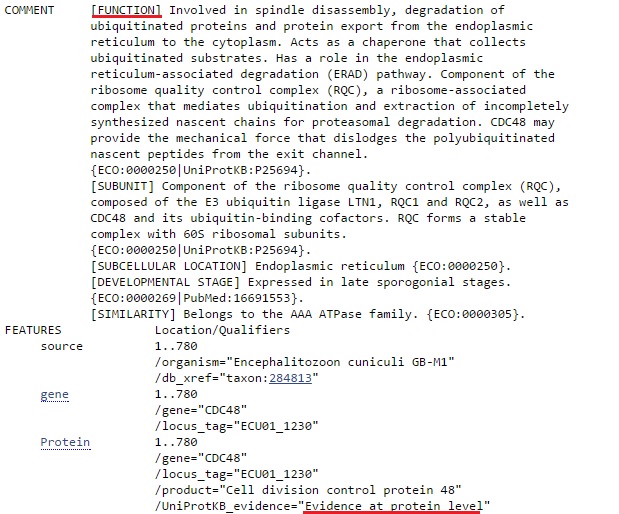
Белок, контролирующий деление клеток, 48 участвует в разборке веретена деления, в деградации убиквитинированных белков и в экспорте белков из эндоплазматического ретикулума в цитоплазму. Действует как шаперон, который собирает убиквитинированные субстраты. Играет роль в расщеплении белка, связанное с эндоплазматическим ретикуломом. Компонент комплекса, контролирующего качество рибосом (RQC). В выравнивании находки есть 2 крупных участка (рис.7). В обоих участках встречаются гэпы. У других находок гэпы встречаются в тех же позициях. Это говорит о том, что границы экзонов определены неправильно. 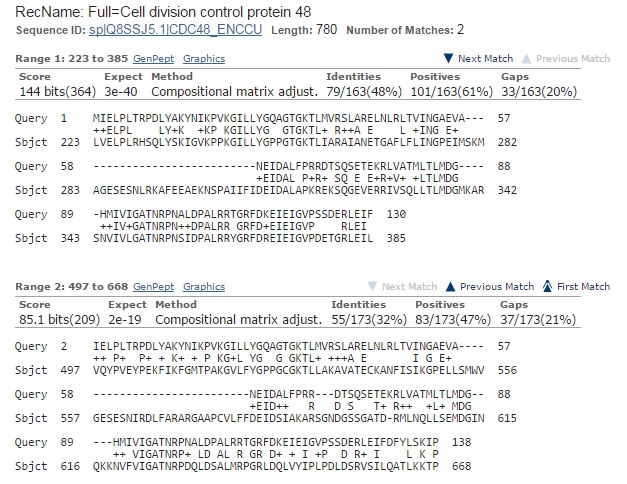
g13 Поиск был произведен по Swiss-Prot (Fungi). Лучшая находка имеет Query over 97%. Продукт предсказан по гомологии. Функция белка не определена. 
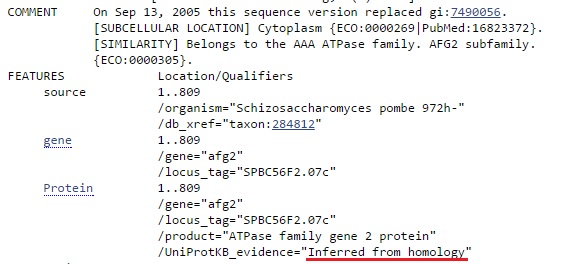
В выравнивании находки есть 2 участка (рис.10), в которых также встречаются гэпы (процент гэпов меньше, чем в выравниваниях предыдущих генов), но у других находок гэпы встречаются примерно в тех же позициях. Из этого можно сделать вывод, что границы экзонов определены неправильно. 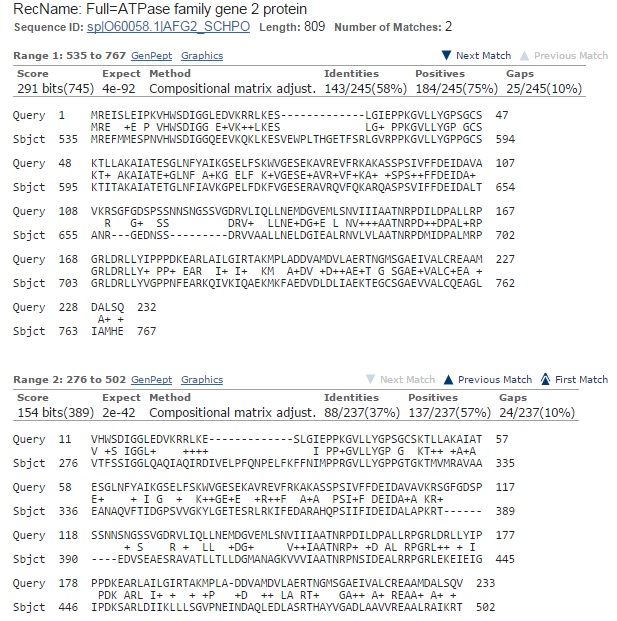
g19 Для данной последовательности я также запустила blastp (поиск производился по банку Swiss-Prot). На рис.11 виден консервативный домен, который встречается почти во всех находках (длина и локализация в белке совпадают). 
Продукт данной находки предсказан по гомологии. Опрделена функция белка: компонент комплекса NOP7, который необходим для созревания 25S и 5.8S рРНК и формирования рибосом 60S. 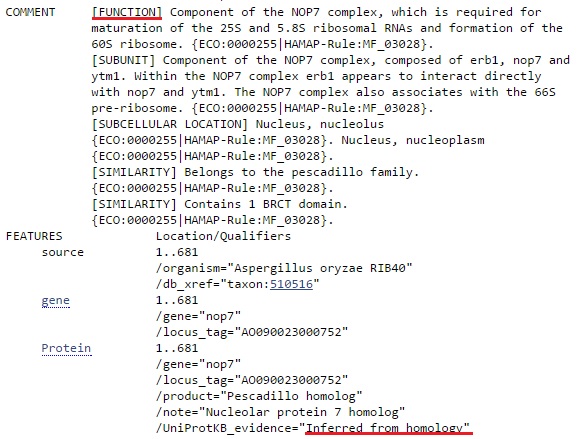
Выравнивание лучшей находки представлено на рис.13. На нем показан один крупный участок. В выравниваниях других находок (рис.14) можно увидеть 2 участка (первый соответствует консервативному домену). Гэпы, найденные в 1 участке, у других находок встречаются в тех же позициях. Во 2 участке также есть гэпы. Это говорит о неправильном определении границ экзонов сервером AUGUSTUS. 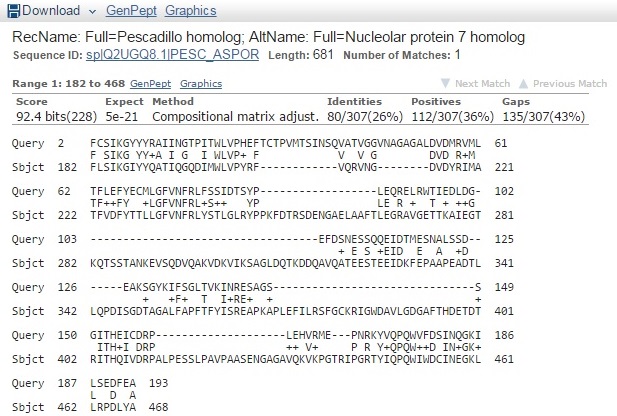
g20 Для последовательности g20 также был запущен blastp (Swiss-Prot, Fungi). В результате было получено всего 3 находки (рис.15). Данные находки довольно плохие. 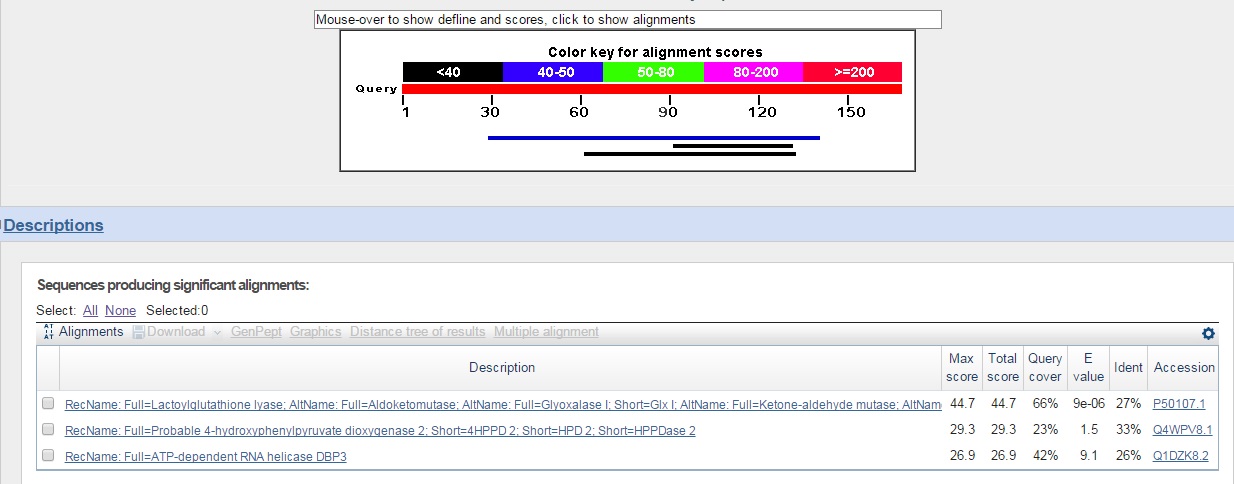
При запуске blastp по nr было получено довольно много находок (рис.16). 
Из этих результатов видно что все находки представляют белок метилмалонил-CoA эпимераза. Достоверной информации о гомологии нет. Из выравнивания (рис.17) видно, что встречаются гэпы. Гэпы есть во всех находках, что говорит о неверном определении границ экзона. 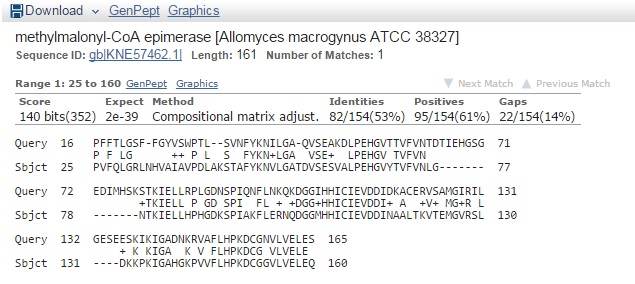
Задание 2. Сравнение аннотации Refseq и AUGUSTUS одного гена человека. Для выполнения данного задания был выбран ген peptidylprolyl isomerase E (cyclophilin E) (PPIE, ID: 10450), который кодирует белок из семейства пептидилпролил-цис-транс-изомераз (PPIase). Эти белки катализируют цис-транс-изомеризацию пептидных связей олигопептидах и ускоряют фолдинг белков. Данный белок содержит высоко консервативный циклофилиновый домен, а также РНК-связывающий домен. Координаты гена: хромосома 1, от 39738845 до 39763914 п.н. (в UCSC от 39738882 до 39753707), прямая цепь, длина: 25070 п.н. (в UCSC 14826 п.н.). Для выполнения данного задания использовался UCSC Genome Browser. Далее я выбрала последнюю сборку генома человека (hg38) и нашла интересующий ген с помощью Genome Browser. Отрегулировав нижнее меню, оставила три трэка: base position, Refseq и AUGUSTUS. 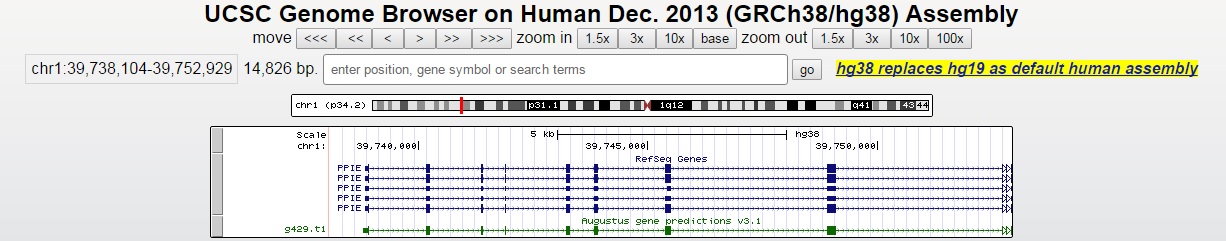
Затем были получены предсказания экзон-интронной структуры, выполненные RefSeq и AUGUSTUS, в виде таблиц. RefSeq выдал предсказания для 5 изоформ. Все они представлены (а так же результаты предсказания AUGUSTUS) в таблице в формате Excel. 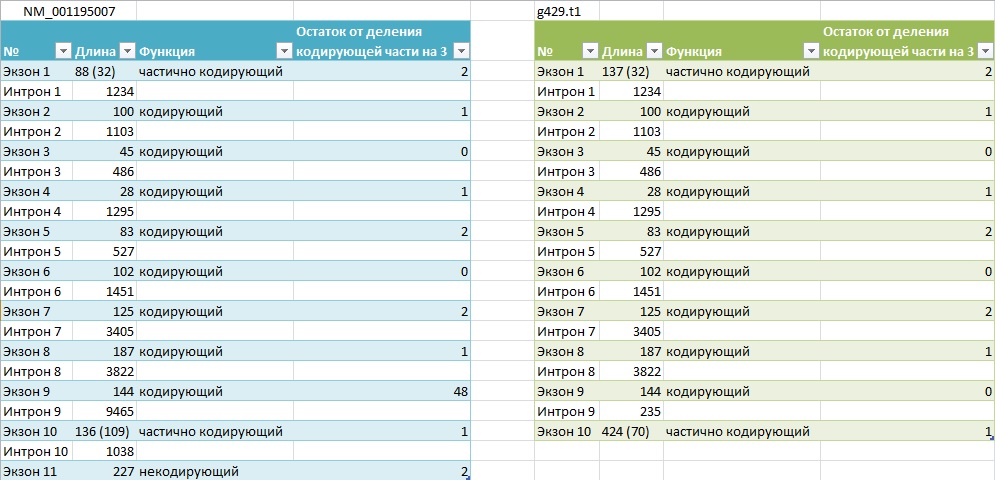
На рис.19 представлены сводные таблицы интрон-экзонной структуры гена самой полной изоформы (изоформы - белки, полученные альтернативным сплайсингом) для двух аннотаций. Как видно из рис.18 и 19, аннотации RefSeq и AUGUSTUS расходятся в описании некодирующих регионов первого экзона. Также можно заметить, что в аннотации Refseq есть некодирующий экзон, который входит в нетранслируемую область. Возможно, в связи с этим описания последних экзонов расходятся. Остальные данные идентичны. |