Учебный сайт Полины Байкузиной | |||
| Главная | Семестры | О себе | Ссылки |
A- и В- формы ДНК. Структура РНКЗадание 1. В этом упражнении требовалось построить модели структур A-, B- и Z-формы ДНК с помощью программы fiber пакета 3DNA. Пакет 3DNA один из популярных пакетов программ для анализа и простейшего моделирования структур нуклеиновых кислот. Были построены два дуплекса ДНК, последовательность одной из нитей которых представляет собой 5 раз повторенную последовательность "gatc". Z-форма представлена 5 раз повторенной парой G-C (программа fiber позволяет строить только такую Z-форму ДНК). Структуры дуплексов сохранены в файлах A-форма, B-форма, Z-форма. Задание 2. Упражнение 1.
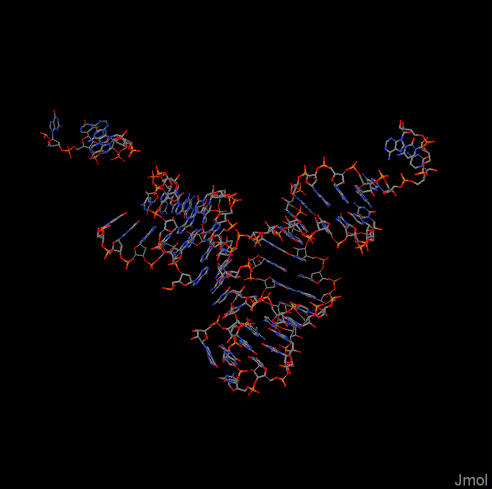
Упражнение 2. В данном упражнении требовалось получить структуры тРНК и ДНК-белкового комплекса в формате PDB с сайта PDB. Были получены файлы со структурами тройного комплекса дрожжевой тирозил-трнк синтетазы и engrailed homeodomain-DNA. Упражнение 3. В данном упражнении требовалось проверить полученные структуры РНК и ДНК на наличие разрывов. На рис.1 изображена структура тРНК. В структуре есть разрывы, которые делят нуклеиновую кислоту на 6 фрагментов. Возможно, что структура на самом деле содержит несколько фрагментов. На рис.2 для более удобного изображения показана ленточная модель тРНК.
На рис.3 изображена структура ДНК. В молекуле разрывов не обнаружено. 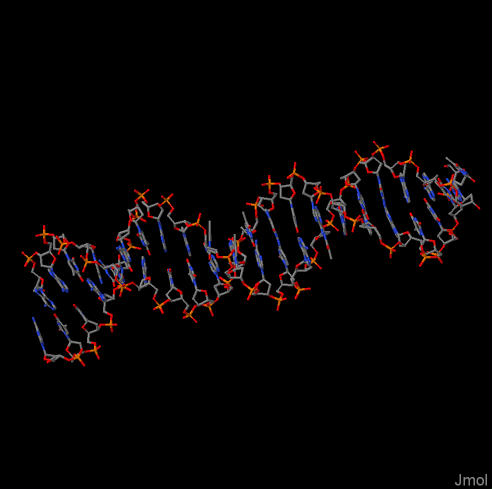
Сохраненные координаты атомов ДНК и РНК. Задание 3. Упражнение 1. В данном упражнении необходимо было определить большую и малую бороздки ДНК. Для этого была взята структура B-формы ДНК. На рис.4 изображена молекула ДНК с отмеченными большой и малой бороздками. 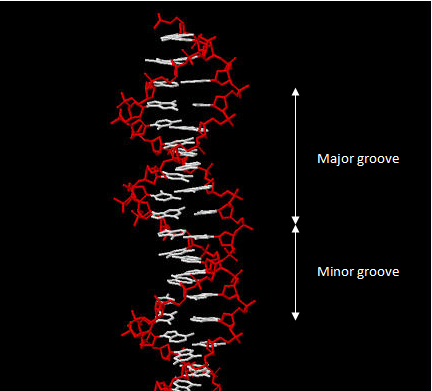
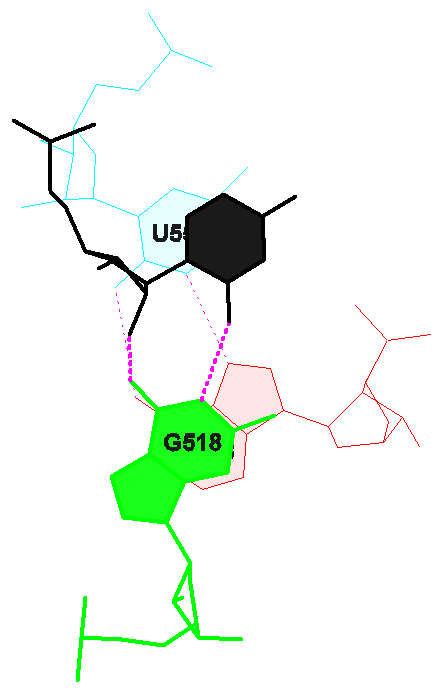
Был выбран остаток тиминового нуклеотида под номером 11. При помощи программы Jmol нужно было определить какие атомы основания явно обращены в сторону большой бороздки, а какие в сторону малой. С помощью программы ChemSketch было получено изображение тимина (рис.5). Красным цветом отмечены атомы, обращенные в сторону большой бороздки, синим - в сторону малой бороздки. 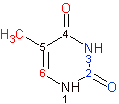
Упражнение 2. Сравнение основных спиральных параметров разных форм ДНК.
Как видно из таблицы, в А-форме ДНК большая бороздка по ширине меньше, чем малая, но большая бороздка глубже. Упражнение 3. Сравнение торсионных углов в структурах А- и В-форм.
Как видно из таблицы, значения углов A- и В-форм не совпадают со значениями, приведенными в презентации. Задание 4. Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA. Т.к.пакет 3DNA работает только со старым форматом PDB, для перевода файлов в старый формат была использована программа remediator, установленная на kodomo. Для анализа структур нуклеиновых кислот были использованы программы find_pair и analyze. Программа find_pair определяет спаренные основания и положения спиралей в структуре. В результате работы программ был создан ряд файлов с описанием разных параметров структуры, в файлах 1HDD_old.out и 2DLC_old.out можно найти описание водородных связей, значения всех торсионных углов, ширину малой и большой бороздок и т.д. Упражнение 1. Определение торсионных углов нуклеотидов. В данном упражнении требовалось определить торсионные углы в заданной структуре ДНК и тРНК; с помощью Excel определить среднее значение каждого из торсионных углов (краевые нуклеотиды не рассматривались). Таблица с значениями торсионных углов представлена здесь. На данной таблице представлены средние значения каждого из торсионных углов.
Наиболее "деформированным" нуклеотидом в молекуле ДНК является Т10. Значения торсионных углов α, β, ζ отклоняются от средних значений. В молекуле тРНК - G6. Углы α, β, γ наиболее сильно отклоняются от средних значений. Упражнение 2. Определение структуры водородных связей.
Упражнение 3. Поиск возможных стекинг-взаимодействий. В файле 2DLC.out были найдены данные о величине площади
"перекрвывания" двух последовательных пар азотистых оснований. Для пары с наибольшим значением step13
(uP/ga) было получено стандартное изображение стекинг-взаимодействия с помощью программ
ex_str и stack2img пакета 3DNA. Результат представлен на рис.6.
|