Нуклеотидный blast
Задание 1 представлено в 4 задании практикума №7.
Задание 2. Сравнение списков находок нуклеотидной последовательности 3-я разными алгоритмами blast.
В данном задании нужно было для последовательности из практикума 7 запустить 3 разных
алгоритма blast (blastn, megablast, discontiguous megablast), а затем сравнить результаты работы.
Для работы были установлены следующие параметры: поиск производился по роду Ophiopholis (при выборе конкретного вида получалось слишком мало находок),
максимальное количество находок - 1000, остальные параметры были оставлены по умолчанию. Также сходство находок должно быть заметно разным: находки имеют
диапазон сходства от 80% до 100%.
Таблица 1. Результаты работы алгоритмов.
| Алгоритм |
blastn |
megablast |
discontiguous megablast |
| Число находок |
50 |
44 |
50 |
| E-value худшей находки |
6e-133 |
1e-155 |
6e-133 |
| Сходство худшей находки |
80% |
82% |
80% |
| Query cover |
68% |
86% |
68% |
 Рис.1. Часть находок алгоритма blastn. Красным выделены находки, не найденные с помощью алгоритма megablast.
Рис.1. Часть находок алгоритма blastn. Красным выделены находки, не найденные с помощью алгоритма megablast.
Результаты blastn и discontiguous blastn оказались одинаковыми (рис.2 и 3). Оба алгоритма выдали находки, которые можно считать достоверными
(E-value = 0.0, за исключением худшей находки, приведенной в таблице 1).
 Рис.2. Часть находок алгоритма blastn.
Рис.2. Часть находок алгоритма blastn.
 Рис.3. Часть находок алгоритма discontiguous megablast.
Рис.3. Часть находок алгоритма discontiguous megablast.
Как видно из таблицы 1, в megablast не могут найтись последовательности с низким query cover, потому что паттерн для поиска этим алгоритмом
составляет 28 букв (для остальных - 11 букв). По числу находок и по значениям E-value видно, что megablast можно использовать для поиска только
гомологичных последовательностей.
Алгоритм blastn используется для поиска любых сходных последовательностей; discontiguous megablast ищет сходные последовательности, являющиеся
гомологами. Т.к. результаты получились идентичными, можно предположить, что все найденные находки с помощью алгоритма blastn являются гомологами.
Задание 3.2. Проверка наличия гомологов пяти белков в геноме одного из организмов.
Для этого задания был взят организм X5 (Amoboaphelidium). Далее были выбраны 5 белков, которые должны быть почти у всех эукариот:
- TF3A_HUMAN (AC: Q92664) - транскрипционный фактор TFIIIA. Необходим для правильной инициации транскрипции генов 5S РНК
с участием РНК-полимеразы III. Может инициировать транскрипцию гена 5S-рибосомной РНК и поддерживать стабильность транскрипции других генов.
- H2A1_HUMAN (AC: P0C0S8) - гистон H2A 1 типа. Основной компонент нуклеосомы. Нуклеосомы
упаковывают ДНК в хроматин. Гистоны играют цетральную роль в регуляции транскрипции, репарации ДНК, репликации ДНК и хромосомной стабильности.
Доступность ДНК регулируется сложным комплексом пост-трансляционных модификаций гистонов, также называемых гистоновым кодом, и ремоделированием нуклеосом.
- DPOLB_HUMAN (AC: P06746) - ДНК-полимераза β. Играет ключевую роль в репарации ДНК.
Имеет 5'-дезоксирибо-5'-фосфатлиазной актисвностью (dRp-лиазной), которая удаляет фосфат на 5'-конце, и также действует как ДНК-полимераза, добавляя по
1 нуклеотиду к 3'-концу возникающих однонуклеотидных разрывов. В отличие от других ДНК-полимераз проводит синтез ДНК ступенчато, а не процессивно.
- ACTB_HUMAN (AC: P60709) - актин, цитоплазматический 1. Актины - высоко консервантивные белки,
которые участвуют в различных видах клеточной подвижности и повсеместно экспрессируются во всех эукариотических клетках.
- HS105_HUMAN (AC: Q92598) - белок теплового шока Hsp105. Препятствует агрегации
денатурированных белков в клетках при сильном стрессе, при котором уровень АТФ заметно сокращается. Ингибирует активность HSPA8/HSC70 АТФ-аз и шаперонов.
Последовательности всех белков
Для работы алгоритма tblastn нужно было создать базу данных из последовательности генома организма Amoboaphelidium (команда: makeblastdb -in X5.fasta -dbtype nucl).
Далее для поиска гомологов запустила tblastn (команда: tblastn -query proteins.fasta -db X5.fasta -out Proteins.out -outfmt 7).
В геноме Amoboaphelidium были найдены все вышеперечисленные белки. Результаты работы представлены в табл.2 и
в файле. За хорошие находки я считала находки с E-value меньше 0.1.
Таблица 2. Результаты поиска пяти белков в геноме Amoboaphelidium (сборка генома) с помощью tblastn.
| Белок |
Число находок (хорошие/все) |
Параметры лучшей находки |
| Запись |
Процент идентичности |
Query cover |
E-value |
| TF3A_HUMAN |
57/113 |
scaffold-110 |
42.17% |
22,7% |
2e-15 |
| H2A1_HUMAN |
6/8 |
scaffold-57 |
90.00% |
77,5% |
1e-53 |
| DPOLB_HUMAN |
2/5 |
scaffold-22 |
25.87% |
94,6% |
3e-18 |
| ACTB_HUMAN |
18/22 |
scaffold-444
scaffold-17 |
93.87% |
100% |
0.0 |
| HS105_HUMAN |
16/23 |
unplaced-959 |
41.55% |
85,6% |
3e-143 |
Результаты выдачи tblastn:
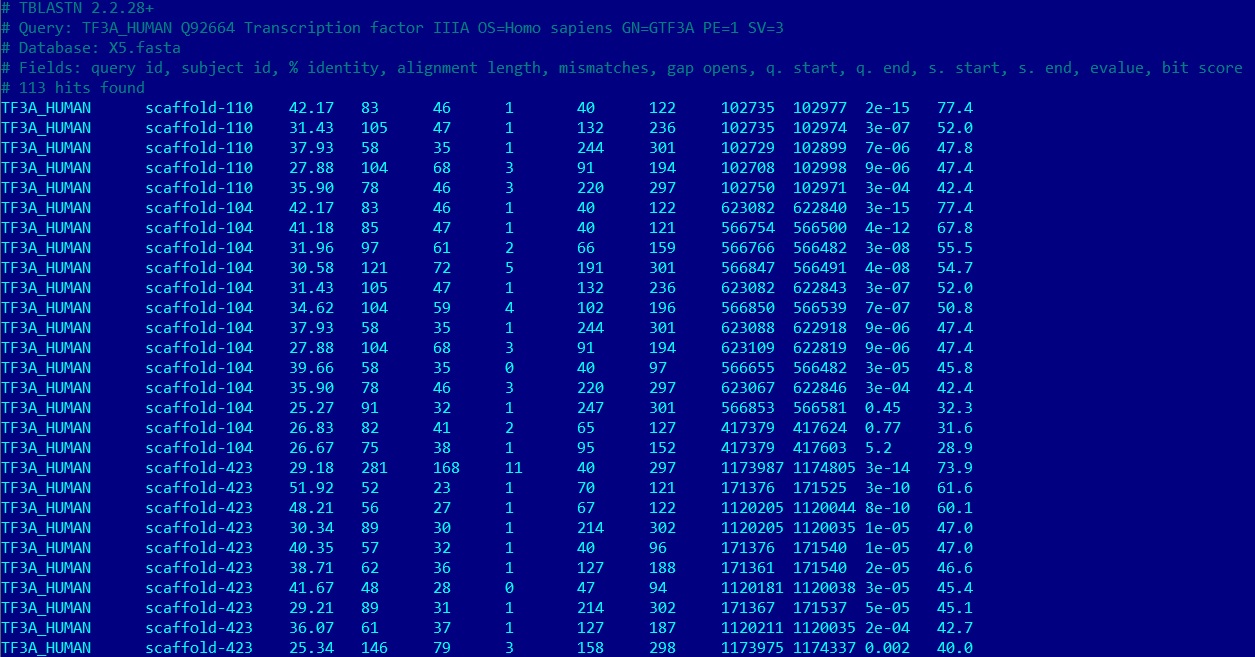 Рис.4. Часть результатов, полученных с помощью алгоритма tblastn для белка TF3A_HUMAN.
Рис.4. Часть результатов, полученных с помощью алгоритма tblastn для белка TF3A_HUMAN.
 Рис.5. Результаты, полученные с помощью алгоритма tblastn для белка H2A1_HUMAN.
Рис.5. Результаты, полученные с помощью алгоритма tblastn для белка H2A1_HUMAN.
- Для данного белка найдены гомологичные участки из одного скэффолда. Параметры, как видно из рис.5, абсолютно одинаковы. Это позволяет
предположить, что эти находки представляют собой повторы.
 Рис.6. Результаты, полученные с помощью алгоритма tblastn для белка DPOLB_HUMAN.
Рис.6. Результаты, полученные с помощью алгоритма tblastn для белка DPOLB_HUMAN.
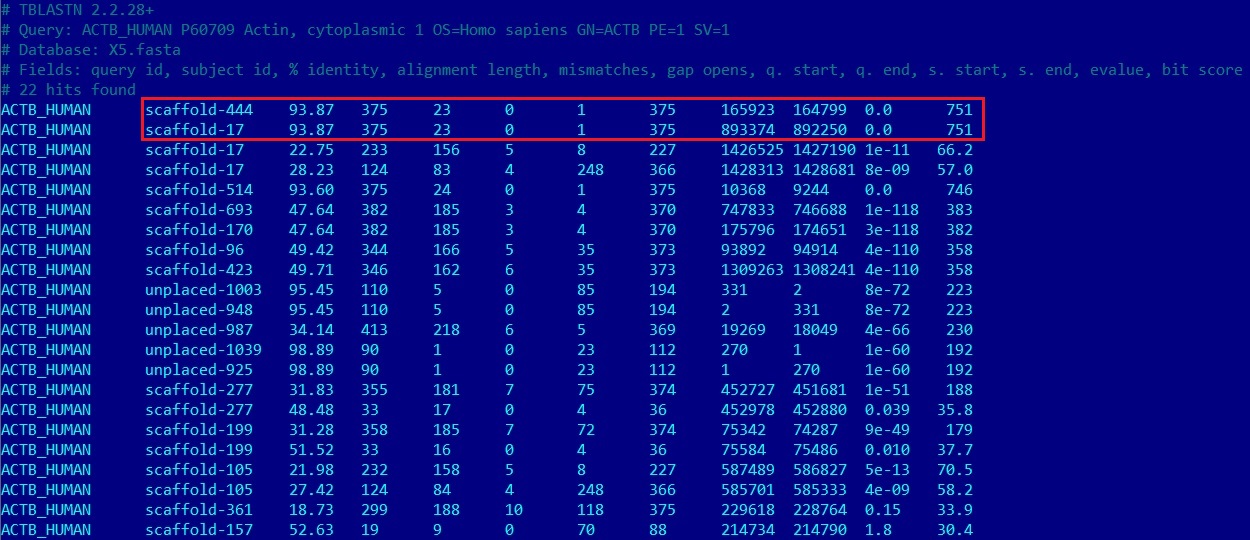 Рис.7. Результаты, полученные с помощью алгоритма tblastn для белка ACTB_HUMAN.
Рис.7. Результаты, полученные с помощью алгоритма tblastn для белка ACTB_HUMAN.
- Из рис.7 видно, что лучшие находки принадлежат записям scaffold-444 и scaffold-17. Они имеют одинаковые параметры. E-value обеих находок равно
0.0, что говорит о их гомологичности.
 Рис.8. Результаты, полученные с помощью алгоритма tblastn для белка HS105_HUMAN.
Рис.8. Результаты, полученные с помощью алгоритма tblastn для белка HS105_HUMAN.
|







