Создание репрезентативной выборки гомологов белка YojM_BACSU
Для поиска гомологов был осуществлён алгоритм BLASTP по базе данных RefSeq, результатом которого оказались белки с наиболее совпадающими аминокислотными последовательностями. Гомологами считались белки с E-value < 10-5 (в этом случае степень покрытия выравнивания составляла ~55-75%). Дополнительные параметры указаны в таблице 1.
| Таблица 1. Параметры поиска BLAST | |||||
|---|---|---|---|---|---|
| Поиск | Алгоритм BLAST | Название базы данных | Ограничения по таксонам | Порог e-value | Максимальное количество хитов |
| По прокариотам | BlastP | Reference proteins | не Firmicutes и не Eukaryota |
1 | 1000 |
| По эукариотам | BlastP | Reference proteins | только Eukaryota | 1 | 250 |
Поиск по прокариотам
Для поиска по прокариотам из всех живых организмов были исключены Eukaryota и Firmicutes - филум, которому принадлежит Bacillus subtilis (требовалось по условию). Было получено 1000 хитов алгоритма BLAST, из которых по первым 824 хитам была получена сводка GenPept базы данных GenBank. После этого как будто случайным образом было выбрано 25 белков прокариот из разных филумов. Однако текущие параметры BLAST не исключают вирусов, которые для полноты картины также были добавлены в выборку, отображённую в таблице 2 (бактерия с исходным белком выделена цветом).
Поиск по эукариотам
Для этого поиска BLAST проводился только по доминиону Eukaryota, с ограничением в 250 хитов. После чего ход действий ничем не отличался, за исключением проверки белка на происхождение из генома хлоропластов или митохондрий. В итоге, нашлось 2 необычные записи:
PREDICTED: superoxide dismutase [Cu-Zn], chloroplastic-like [Cucumis sativus]
PREDICTED: superoxide dismutase [Cu-Zn], chloroplastic-like [Bombus impatiens]
Насколько я могу понять, то эти белки по происхождению хлоропластные. Но самое интересное то, что 1 запись принадлежит огурцу обыкновенному, а вторая - шмелю Bombus impatiens. Надеюсь, что это ошибка, ведь обе записи предсказаны автоматически компьютерным анализом методом GNOMON. Отчёт по выборке также представлен в таблице 2.
| Таблица 2. Встречаемость белков в различных таксонах | |||
|---|---|---|---|
| Домен | Филум/Царство | Название организма | Количество белков |
| Archaea | Methanocellales | Methanocella arvoryzae MRE50 | 1 |
| Methanosarcinales | Methanosarcina barkeri str. Fusaro | 1 | |
| Bacteria | Acidobacteriales | Granulicella mallensis MP5ACTX8 | 2 |
| Granulicella tundricola MP5ACTX9 | |||
| Actinomycetales | Rhodococcus triatomae BKS 15-14 | 2 | |
| Streptosporangium roseum DSM 43021 | |||
| Aquificales | Aquifex aeolicus VF5 | 2 | |
| Thermocrinis albus DSM 14484 | |||
| CFB group bacteria | Formosa sp. AK20 | 2 | |
| Fulvivirga imtechensis AK7 | |||
| Chlamydias | Candidatus Protochlamydia amoebophila UWE25 | 2 | |
| Parachlamydia acanthamoebae UV-7 | |||
| Cyanobacteria | Lyngbya sp. PCC 8106 | 3 | |
| Pseudanabaena biceps PCC 7429 | |||
| Synechococcus sp. CC9605 | |||
| Deinococcales | Deinococcus gobiensis I-0 | 1 | |
| Firmicutes | Bacillus subtilis strain 168 | 1 | |
| Planctomycetes | Planctomyces maris DSM 8797 | 2 | |
| Rhodopirellula sallentina SM41 | |||
| Proteobacteria | Azoarcus sp. KH32C | 6 | |
| Campylobacter showae CSUNSWCD | |||
| Cystobacter fuscus DSM 2262 | |||
| Salmonella enterica subsp. enterica serovar Typhimurium str. LT2 | |||
| Sphingomonas sp. MM-1 | |||
| Vibrio cholerae O1 biovar El Tor str. N16961 | |||
| Solibacterales | Candidatus Solibacter usitatus Ellin6076 | 1 | |
| Eukaryotes | Acanthamoeba | Acanthamoeba castellanii str. Neff | 1 |
| Animals | Acyrthosiphon pisum | 14 | |
| Anolis carolinensis | |||
| Aplysia californica | |||
| Bombus impatiens | |||
| Branchiostoma floridae | |||
| Caenorhabditis elegans | |||
| Culex quinquefasciatus | |||
| Drosophila melanogaster | |||
| Ixodes scapularis | |||
| Otolemur garnettii | |||
| Rattus norvegicus | |||
| Salmo salar | |||
| Sarcophilus harrisii | |||
| Xenopus laevis | |||
| Capsaspora | Capsaspora owczarzaki ATCC 30864 | 1 | |
| Cellular slime molds | Dictyostelium discoideum AX4 | 1 | |
| Ciliates | Tetrahymena thermophila | 1 | |
| Green plants | Cucumis sativus | 3 | |
| Physcomitrella patens subsp. patens | |||
| Selaginella moellendorffii | |||
| Fungi | Cryptococcus gattii WM276 | 2 | |
| Penicillium chrysogenum Wisconsin 54-1255 | |||
| Perkinsida | Perkinsus marinus ATCC 50983 | 1 | |
| Schizopyrenida | Naegleria gruberi strain NEG-M | 1 | |
| Viruses | Baculoviridae | Mamestra configurata nucleopolyhedrovirus B | 2 |
| Pieris rapae granulovirus | |||
| Phycodnaviridae | Paramecium bursaria Chlorella virus 1 | 2 | |
| Paramecium bursaria Chlorella virus FR483] | |||
| Poxviridae | Amsacta moorei entomopoxvirus 'L' | 1 | |
Последовательности аминокислот гомологов YojM_BACSU из представленных организмов содержатся в файлах Prokaryota.fasta и Eukaryota.fasta
и Eukaryota.fasta . Для редактирования этих последовательность был использован скрипт
. Для редактирования этих последовательность был использован скрипт , в котором вручную менялись имена файлов.
, в котором вручную менялись имена файлов.
Множественное выравнивание гомологов белка YojM_BACSU
На сервере Европейского Биоинформатического Института программой Muscle было проведено множественное выравнивание гомологов белка YojM_BACSU одновременно из эукариот и прокариот, найденнх в предыдущем разделе. Полная картина полученного выравнивания изображена на рисунке 1.
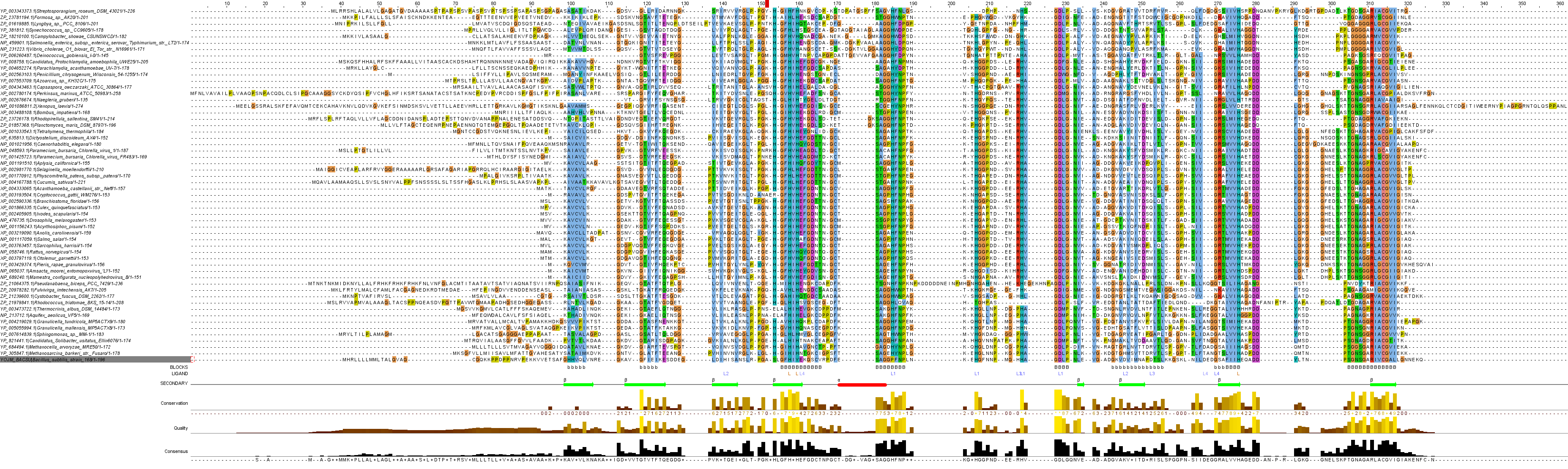 Рисунок 1. Множественное выравнивание гомологов белка YojM_BACSU.
Рисунок 1. Множественное выравнивание гомологов белка YojM_BACSU. Использована стандартная окраска аминокислотных остатков ClustalX без ограничения на степень консервативности. Строка с исходным белком выделена серым.
К выравниванию было добавлено 3 новые строки аннотации SECONDARY, LIGAND и BLOCKS:
- SECONDARY. Здесь наглядно отображена вторичная структура белков, основываясь на информации из банка данных PDB по строению белка 1XTM бактерии Bacillus subtilis.
- LIGAND. В этом поле указаны лиганды напротив соответствующих аминокислот. Серым цветом обозначены ионы цинка, бурым - меди. Цифры соответсвют номеру лиганда: L - Cu500, L1 - Zn501, L2 - Zn502, L3 - Zn505, L4 - Zn506.
- BLOCKS. В этой строке буквами "B" указаны участки белка, которые точно можно отнести к "блоку" (см. базу данных BLOCKS), а буквами "b" - участки, которые лишь похожи на "блоки" (либо они недостаточно консервативны, либо содержат гэпы)
Результаты анализа множественного выравнивания гомологов белка YojM_BACSU
В целом, аминокислотная последовательность гомологов оказалась почти консервативна.
Наихудшим образом выровнены концевые участки белков. Это связано с тем, что они не учавствуют в формировании вторичной структуры белка и являются петлями, которые могут наращиваться, сокращаться, или в них могут могут возникать никому не нужные ни на что не влияющие мутации.
Как и стоило ожидать, участки консервативности напрямую связаны с элементами вторичной структуры белка: они почти полностью покрывают каждый из таких участков. Но не все участки вторичной структуры являются стого консервативными. К тому же у ряда организмов в пределах спирали (отмеченной красным) возникает инсерция, и не ясно, что эта спираль из себя представляет в этой группе организмов *.
На элементы вторичной структуры белка почти не приходится колонок-гэпов (имеется 2-3 + описанный выше случай со спиралью), в отличие от других участков белка. Выбранные мною блоки строго соотвествуют участкам вторичной структуры и аминокислотам, связывающих лиганды. Блоки выбирались согласно критерию, приведённому в подсказаках: "всегда под 'блоками' подразумевают как-то выровненные по всей толщине (не обязательно сильно консервативные) участки выравнивания, противопоставляя им вообще не выровненные петли".
Как выяснилось, в структуре изучаемого белка имеются как предельно консервативные аминокислоты, связывающие лиганды (ионы металлов Zn и Cu), так и совсем не консервативные. Так, ионы Cu500, Zn501, Zn506 удерживаются 3 аминокислотами, 2 из которых более чем консервативны (см. рис. 2 - цветность означает консервативность, лиганд-связывающие аминокислоты не подписаны, ввиду громоздкости рисунка в противном случае - эти данные имеются в рис.1).
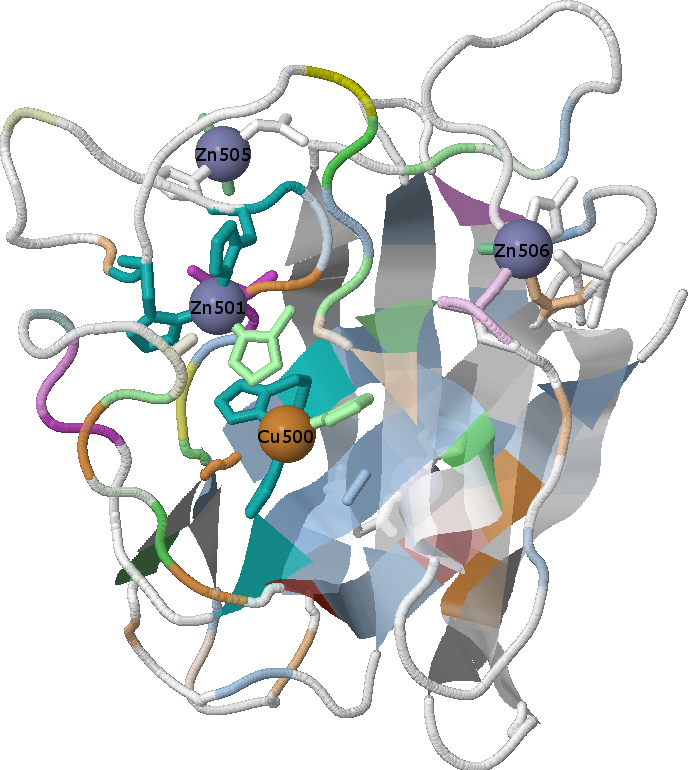
Рисунок 2. Консервативность лиганд-связывающих аминокислот, таковые обозначены цветом.
На периферии белка находятся 2 иона цинка Zn502 и Zn505, которые скорее всего могут удерживаться лишь в ряде найденных гомологов. Согласно полученному выравниванию Zn502 (L2) связывается абсолютно неконсервативными аминокислотами (см. рис.3), и в большинстве случаев заменяется на незаряженную; поэтому вполне возможно, что у большинства этот ион не будет удерживается. Zn505 (L3) с одном стороны у половины организмов связывается либо гистидином, либо аргинином (в остальных случаях пролином или чем-то другим), а с обратной стороны наблюдается огромная изменчивость аминокислотного остатка (ион координирует вокруг себя молекулы воды - связи изображены салатовым).
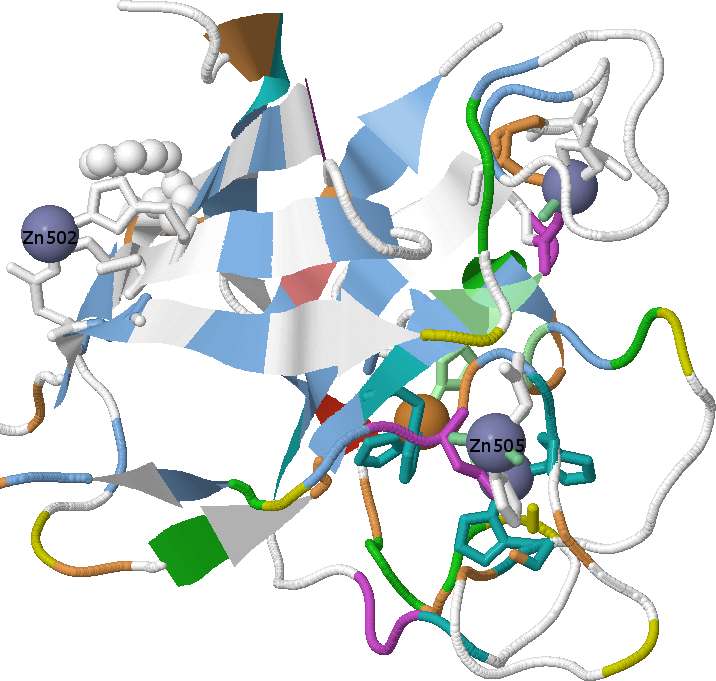
Рисунок 3. Неконсервативное связывание Zn502 и Zn 505.
Выравнивание гомологов из Eukaryota и Prokaryota по раздельности
Ради интереса на том же сервере той же программой при таких же параметрах было проведено множественное выравнивание гомологов белка из эукариот отдельно от выравнивания по прокариотам - приведено на рис.4 и рис.5 соответсвенно.
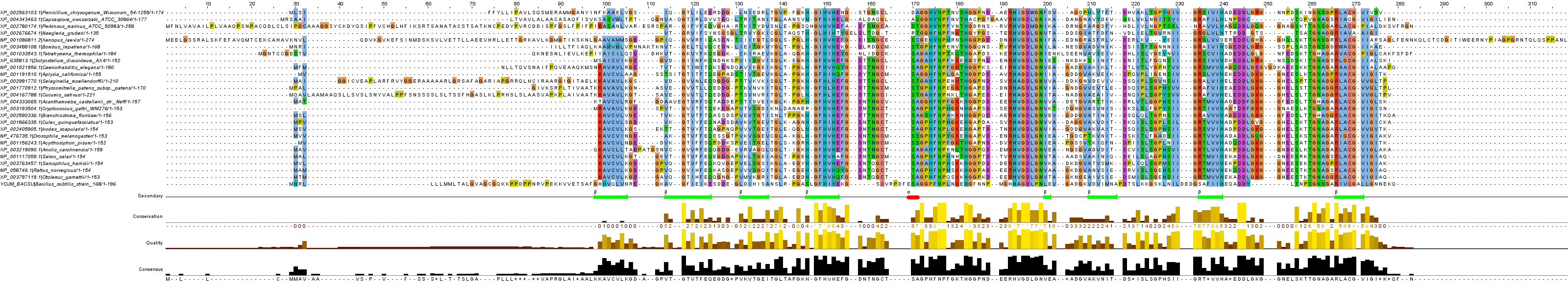 Рисунок 4. Выравнивание белков из Eukaryota
Рисунок 4. Выравнивание белков из Eukaryota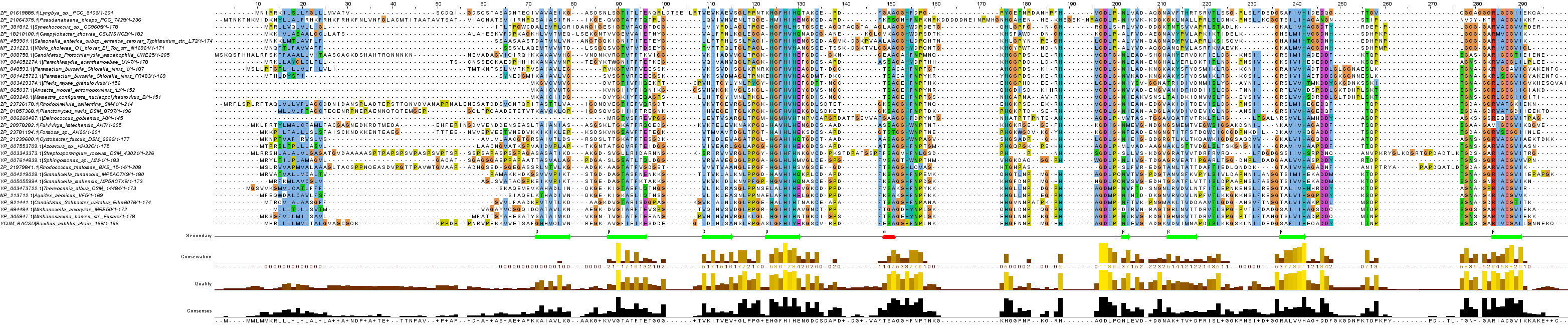 Рисунок 5. Выравнивание белков из Prokaryota
Рисунок 5. Выравнивание белков из Prokaryota- В спирали нет огромного гэпа! * А у эукариот итого хуже: от спирали остаётся всего 2 аминокислотных остатка. Видимо, эта спираль формируется абсолютно слючайно и никакой роли для структуры всего белка не играет.
- Выравнивание по эукариотам в целом лучше, чем про прокариотам. В первом случае основная часть выравнивания почти полностью залита цветом с редкими точечными мутациями или небольшими неконсервативными полосками. А в случае с прокариотами мы имеем положение дел похуже: большое количество неконсервативных столбцов, гэпы в консервативных полосах (колонки-гэпы получились из-за 2-3 бактерий, их не учитываем: это простая случайная инсерция). И что ещё забавно, среди прокариот довольно неплохо выровнялся начальный участок (петля по сути).