Поиск ДНК-белковых контактов в 1RH6
Определение множеств в JMol
В скрипте 1  последовательно заданы множества JMol:
последовательно заданы множества JMol:
- атомов кислорода 2'-дезоксирибозы
- атомов кислорода в остатке фосфорной кислоты
- атомов азота в азотистых основаниях
Cкрипт 2  последовательно даёт изображение:
последовательно даёт изображение:
- всей структуры с PDB-ID 1RH6
- только ДНК в проволочной модели
- той же модели, но с выделенными шариками указанным ранее множеством атомов
ДНК-белковые контакты в 1RH6
| Контакты атомов белка с | Полярные | Неполярные | Всего |
|---|---|---|---|
| остатками 2'-дезоксирибозы | 7 | 26 | 33 |
| остатками фосфорной кислоты | 11 | 44 | 55 |
| остатками азотистых оснований со стороны большой бороздки | 3 | 5 | 8 |
| остатками азотистых оснований со стороны малой бороздки | 1 | 5 | 6 |
define polar oxygen or nitrogen define nonpolar carbon or phosphorus or sulfur select polar and within(3.5, *.??') and protein select nonpolar and within(4.5, *.??') and protein select polar and within(3.5, *.P or *.OP?) and protein select nonpolar and within(4.5, *.P or *.OP?) and protein define major *.c6 or *.n6 or *.o6 or *.c5 or (*.c4 and (c or t)) or *.n7 or *.n4 or *.o4 or *.c5 or *.c8 or *.c5m select polar and within(3.5, major) and protein select nonpolar and within(4.5, major) and protein define minor *.c2 or *.o2 or *.n1 or *.n2 or *. n3 or (*.c4 and (a or g)) or *.n9 or *.c6 select polar and within(3.5, minor) and protein select nonpolar and within(4.5, minor) and protein
Как видно из полученной таблицы, в структуре комплекса преобладают неполярные взаимодействия. Белок имеет почти вдвое больше контактов с остатками фосфорной кислоты, чем с остатками сахара. Согласно таблице, число контактов белка с большой бороздкой и с малой почти равно, т.е. он лежит как в большой бороздке, так и в малой (если говорить, опираясь на 3D-структуру).
Получение схемы ДНК-белковых контактов
С помощью программы nucplot на сервере kodomo была построена схема ДНК-белковых взаимодействий (представлена на рисунке 1, а также в файле nucplot.ps  )
)
 Рис. 1 Схема ДНК-белковых взаимодействий
Рис. 1 Схема ДНК-белковых взаимодействийАнализ полученной схемы
Как видно из схемы, наибольшее количество контактов с белком имеет фосфор 21-ого нуклеотида (хоть и через воду). Однако не понятно, каким местом взаимодействует с ДНК валин-25 цепи А белка (см. рисунок 2). Скорее всего на схеме должен быть указан Val25(B)
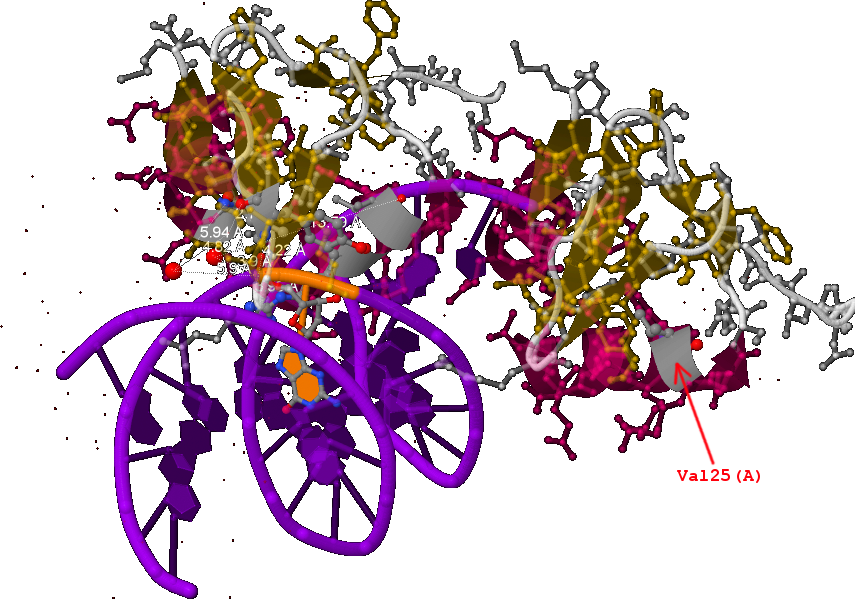 Рис. 2 Пространственное представление ДНК-белковых взаимодействий
Рис. 2 Пространственное представление ДНК-белковых взаимодействийСогласно схеме, наибольшее число контактов с ДНК имеет тирозин с порядковым номером 41 цепи B: он взаимодействует с фосфором 22-ого и 21-ого нуклеотида напрямую и через молекулы воды с 21-ым (см. рисунок 3).
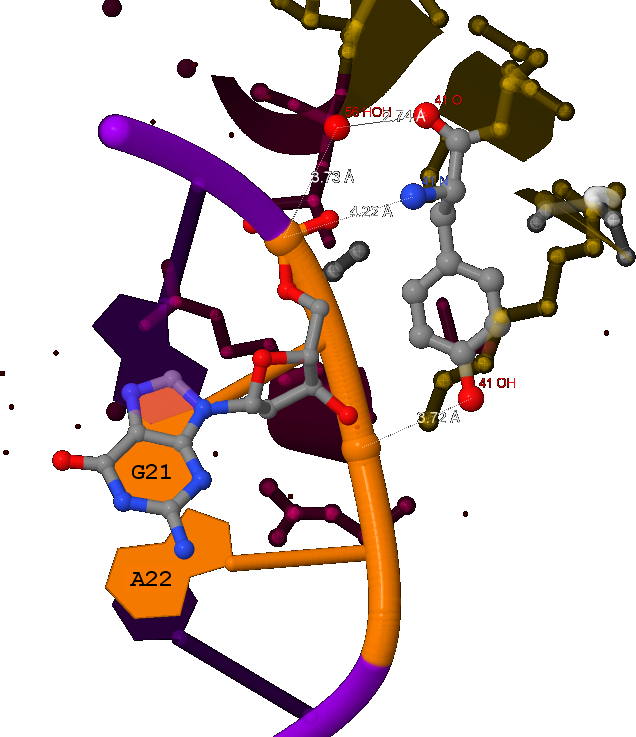
Рис. 3 Изображение контактов Tyr41 с ДНК. Взаимодействующие атомы подписаны (кроме фосфоров), пунктиром обозначены связи. На рисунке также показано окружение Tyr41, G21 и A22 в радиусе 5Å.
Также в схеме имеется единственый случай, когда боковой радикал аминокислоты напрямую взаимодействует (без участия воды) с азотистым основанием ДНК, причём с двумя сразу. Это контакты аминогрупп Аргинина23(B) с кислородами аминокислот Tимин6 и Гуанин5 (см. рисунок 4)
Рис. 4 Изображение контактов Arg23 с ДНК. Взаимодействующие атомы подписаны (кроме кислородов), пунктиром обозначены водородные связи. На рисунке также показано окружение Arg23, T6 и G5 в радиусе 10Å.
Предсказание вторичной структуры тРНК из 1GTR
Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
Программа einverted из пакета EMBOSS находит инвертированные участки в нуклеотидных последовательностях. Но на мой взгляд, она почти непригодна для предсказания вторичной структуры тРНК, т.к. она делит последовательность всего на 2 части и выравнивает их друг против друга. А в тРНК имеются комплементарные участки, идущие сразу друг за другом (T- и D- стебли), которые einverted врядли определит. Тем не менее, были подобраны параметры, при которых полностью находятся акцепторный и антикодоновый стебли:
1GTR_RNA: Score 176: 24/28 ( 85%) matches, 12 gaps
1 ggggtatc---g---ccaa--gcggtaaggcaccggattc 32 |
|||||| | | | || ||| || | ||||| || | предсказание einverted
69 ccccat-gctcctaagcttggagcc-ttac--ggccttag 34 |
ggggta--------------------a-ggcaccggatt |реальная
ccccat-gctc--------------------cggcctta |ситуация
| | | |
акцепторный антикодоновый
стебель стебель
Параметры: -gap 4 -threshold 50 -match 10 -mismatch -4
Результаты занесены в таблицу 2.
Предсказание вторичной структуры тРНК по алгоритму Зукера
На сервере Mobyle@Pasteur проведено предсказание вторичной структуры тРНК из 1GTR по алгоритму Зукера при 37 ℃. Из предложенных вариантов выбран тот, что почти точь-в-точь повторяет реальную структуру (структура изображена на рис. 5)
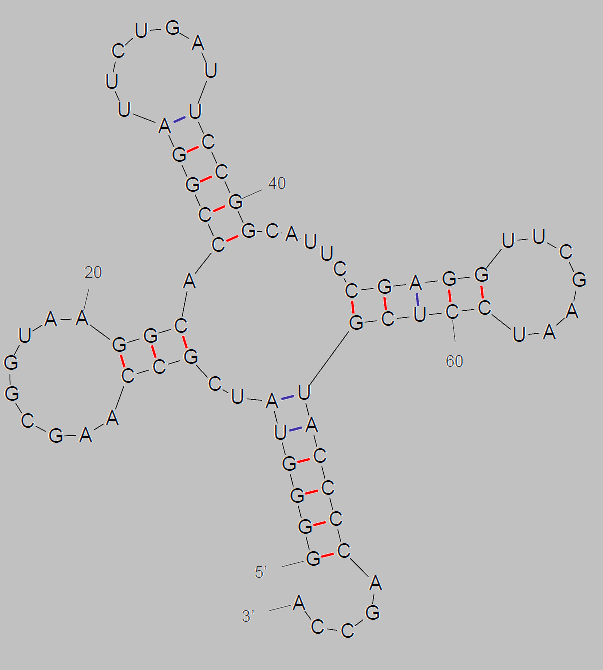
Рис. 5 Предсказанная по алгоритму Зукера вторичная структура тРНК из 1GTR
| Участок структуры | Позиции в структуре (по результатам find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
|---|---|---|---|
| Акцепторный стебель | 5'- 2‒7 -3' 3'- 71‒66 -5' всего 6 пар | 6 из 6 | 6 из 6 |
| D-стебель | 10‒12, 13, 14, 15 25‒23, 45, 21, 48 всего 6 пар | ∅ | 3 из 6 |
| T-стебель | 5'- 49‒53, 54, 55 3'- 65‒61, 58, 18 всего 7 пар | ∅ | 5 из 7 |
| Антикодоновый стебель | 5'- 37‒44 -3' 3'- 33‒26 -5' всего 8 пар | 7 из 8 | 5 из 8 |
| Общее число канонических пар нуклеотидов | 19 (+8 не У-К пар) | 12 (+1 не У-К пара) | 19 |