GO
Белок H8M323
H8M323 - Sec-независимый белок транслоказы TatB патогенной Грам-отрицательной аэробной бактерии Salmonella enterica (subsp. enterica serovar Typhimurium) [штамм 798]. Существование белка предположено по гомологии. На странице UniProt имеется информация о том, какие термины GO (Gene onthology)относятся к данному белку (отображены в табл. 1).
| Онтология | ID | Название | Определение |
|---|---|---|---|
| Биологический процесс | GO:0009306 | Секреция белков | Контролируемое выведение белка из клетки |
| Биологический процесс | GO:0043953 | Транспорт белков с помощью комплекса Tat | Процесс, в котором свернувшиеся белки транспортируются через плазмалемму бактерии или мембраны хлоропластов и митохондрий с помощью комплекса Tat |
| Клеточный компонент | GO:0033281 | TAT-комплекс транспорта белков | Комплекс 3 белков, интегрированных в плазмалемму бактерий или мембрану хлоропластов и митохондрий, вовлечён в мембранный транспорт свернувшихся белков. |
| Клеточный компонент | GO:0005887 | Интегральный компонент плазмалеммы | Компонент плазмалеммы, состоящий из продуктов генов и комплексов белков, некая часть которых входит как минимум в 1 слой мембранного бислоя. Этот компонент включает в себя продукты гена, которые погружены в бислой так, что не подвергаются воздействию снаружи мембраны. |
| Молукулярная функция | GO:0008320 | Активность трансмембранного транспортёра белков | Катализирует транспорт белков с одной стороны мембраны на другую |
Полученная и информация на странице UniProt позволяют понять, что белок является частью TAT-системы секреции (twin-arginine translocation).
Tat-система секреции
Tat - путь транспорта белков, существующий у архей, бактерий и растителных хлоропластов. У бактерий таким образом экспортируются белки через клеточную мембрану, что важно для многих процессов, включая образование клеточной оболочки, образование бактериальных биоплёнок, устойчивость к тяжёлым металлам и др. Эта система примечательна тем, что позволяет транспортировать большие белки, и даже димеры в нативном (свёрнутом) состоянии. К тому же такой транспорт не требует энергии АТФ и основан только на движущей силе, создаваемой протонами.
Tat-комплекс формируют 3 субъединицы: TatA, TatB и TatC, которые кодируются одним опероном tatABC. TatB и TatC формируют связывающий комплекс, а TatA расположена отдельно. Сигнальный пептид для этого комплекса имеет особую структуру (см. рис. 1) и очень консервативный SRRxFLK мотив (содержит 2 аргинина подряд), из-за чего комплекс и получил своё название [1].
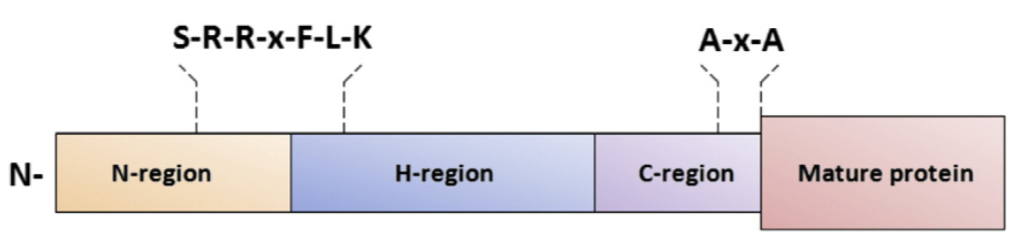
Рис.1 Структура сигнального пептида Tat-комплекса. На N-конце субстратов имеются следующие домены: полярный, богатый амино-группами N-регион, гидрофобное ядро (H-регион) и полярный карбоксильный С-регион. С-регион имеет A-x-A мотив, узнаваемый сигнальными пептидазами [1].
Механизм работы комплекса до конца не изучен, но имеются экспериментальные доказательства модели, изображённой на рис. 2 [2]. Насколько я понимаю, сигнальный пептид не зря имеет гидрофобный домен, т.к. он "просачивается" сквозь мембрану, пока субстрат проходит через комплекс белков TatA.

Рис.2 Модель работы Tat-комплекса у E.Coli и хлоропластов. PMF - proton-motive force, движущая сила, создаваемая протонами [2].
Функциональная роль H8M323 в системе Tat
Белок H8M323 является субчастицей TatB комплеса TatBC системы секреции Tat. TatB и TatC формируют стабильный комплекс, узнающий и связывающий сигнальный пептид (см. рис. 3).
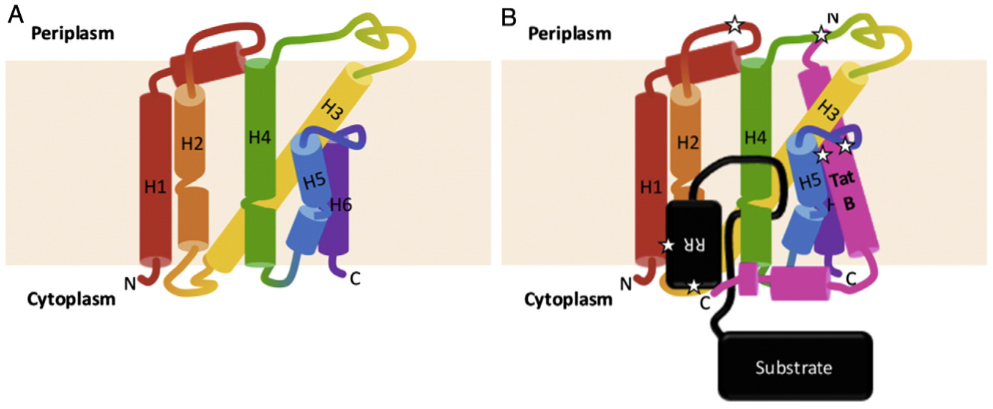
Рис.3 TatBC-комплекс. А: TatA состоит из 6 трансмембранных субъединиц и способна связывать сигнальный пептид. В: TatB способен связываться с TatA в 3 местах, обозначенных звёздочкой ★ [1].
SEED
На сервере SEED (FIG) был найден ген tatB в бактерии Salmonella enterica subsp. enterica serovar Typhimurium, после чего были найдены его 15 ортологов из разных организмов (нет одинаковых видов). На рис. 4 приведены карты окрестностей гена tatB у найденных ортологов.
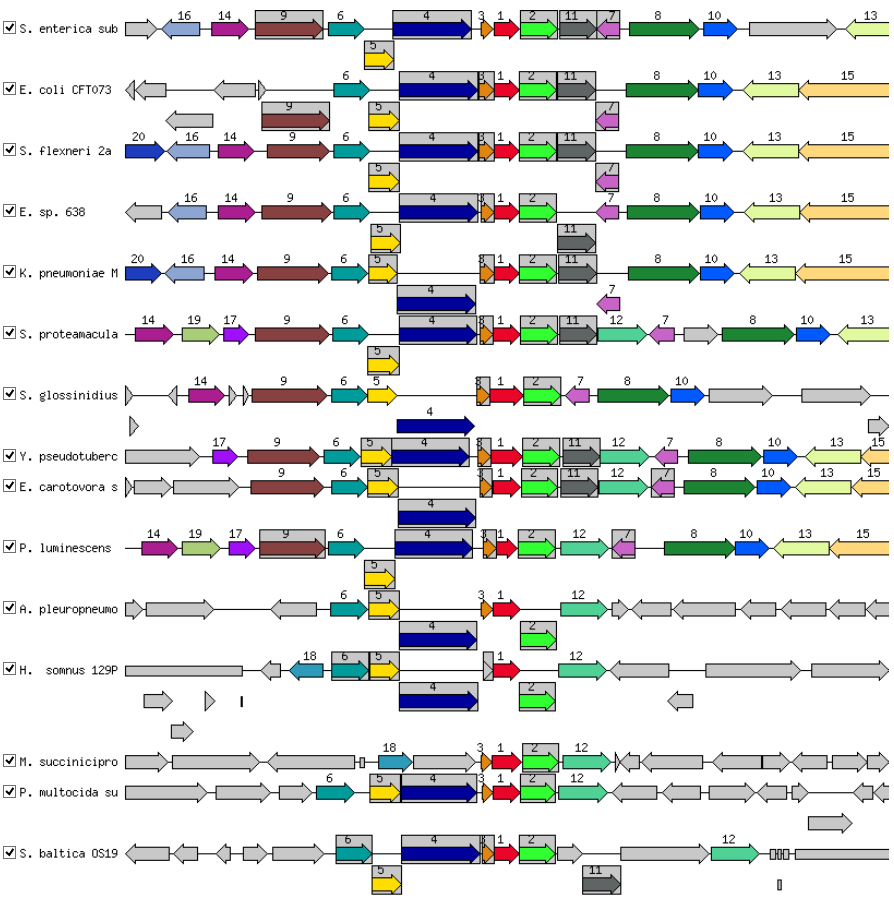
Рис.4 Карты окрестностей гена tatB (обозначен как 1). Рядом, как и положено, находятся гены tatA (3) и tatC (2).
На сервере SEED также было построено выравнивание и сохранено в clustalW-файл, которое также представлено в проекте Jalview и на рис. 5.

Рис.5 Выравнивание последовательностей белка H8M323 с его ортологами.
Также с сервера SEED загружена таблица всех генов окрестностей, по которой построена сводная таблица Excel, отражающая, какие гены (точнее их номера) встречаются в каких геномах (1-ый лист), а также отражает встречаемость некоторых подсистем (2-ой лист).
Выводы
- Согласно системе SEED, для всех 15 ортологов гена tatB верно, что он идёт только в группе с tatA и tatC (образуют секреторный комплекс), причём в одном и том же порядке (что совсем не удивительно). Интересно, что для некоторых карт в группе с этой тройкой идёт ген tatD (11) - дезоксирибонуклеаза, которая относится к той же подсистеме согласно SEED. Однако про эту субчастицу нигде больше не говорится.
- Примечательно, что с этой тройкой почти всегда (14 случаев из 15) ко-локализуются гены белка Ubiquinone biosynthesis monooxygenase UbiB и белка YigP (связанный с генами биосинтеза убихинона). Я не смог найти причин такой колокализации. Однако могу предположить следующее: Tat-комплекс для своей работы использует движущую силу, создаваемую протонами. Убихинон же может иметь восстановленную форму (Co QH2), протоны которой могут быть использованы для полимеризации TatA.
- Ортологичность генов на удалённых от tatB областях сохраняется только для близкородственных организмов (первые 11 хитов запроса на картинке), а потом резко сходит на нет.
Колокализация генов Tat-системы
Попытки найти организм, в котором tatB не был бы колокализован с tatA и tatC, увенчались неуспехом. Как следствие, Tat-комплекс может работать только при участии всех 3 субъединиц. Однако при E-value cut off = 1*E-6 для исследуемого гена и выборке в 1000 организмов нашёлся такой (Syntrophomonas wofei subsp. wolfei str. Goettingen), в котором ген tatA расположен без tatB и tatC - ссылка.
Источники:
[1] "Protein transport by the bacterial Tat pathway"; Patel R, Smith SM, Robinson C; Biochim Biophys Acta. 2014 Feb 26
[2] "The twin-arginine translocation (Tat) protein export pathway"; Palmer T, Berks BC; Nat Rev Microbiol. 2012 Jun 11