Трансмембранные структуры
Трансмембранными доменами белков могут служить α-спирали и β-баррели. В таблице 1 приведены некоторые параметры таких структур. Толщина гидрофобных частей и номера аминокислотных остатков, находящихся в мембране, взяты с сервера Orientations of Proteins in Membranes (OPM). Рассчёты медиан приведены в таблице Excel.
| PDB код | Тип мембраны | Мембрана | Толщина гидрофобной части (Å) | Медиана длины трансмембранного участка (аа) |
|---|---|---|---|---|
| 1BHA | Спираль | Мембрана археи Halobacterium salinarum (бактериородопсин) | 29.6 ± 2.2 | 21 |
| 4PXZ | Спираль | Периплазм E. coli | 36.0 ± 1.9 | 27 |
| 4P79 | Cпираль | Мембрана клеток мыши Mus musculus | 31.8 ± 0.7 | 24.5 |
| 1BXW | Баррель | Внешняя мембрана Грам-отрицательной бактерии (Escherichia coli) | 25.4 ± 1.9 | 9.5 |
| 2FGQ | Баррель | Внешняя мембрана Грам-отрицательной бактерии (Delftia acidovorans) | 25.0 ± 0.9 | 9 |
| 7AHL | Баррель | Секретируется через мембрану Грам-положительной бактерии (Staphylococcus aureus) | 23.5 ± 0.9 | 8.5 |
Невозможно не заметить, что трансмембранный участок β-баррелей более чем в 2 раза короче аналогичного среди α-спиральных белков. Это связано со структурой доменов: α-спирали упакованы более плотно, т.к. аминокислотная цепь белка закручена по спирали, а β-тяжи β-баррелей линейны.
Отбор гомологов
Для отбора гомологов белка с PDBID 3L1L использовался сервис Blastp на NCBI. 3L1L (UniProt: ADIC_ECO57) - аргинин/агматин антипортер бактерии E. coli; главный компонент системы устойчивости к кислотам, позволяющий кишечным патогенам выживать в кислой среде желудка. Он обменивает внеклеточный аргинин на внутриклетоный продукт его декарбоксилирования - агматин, таким образом вытесняя протоны внутри клетки. При выборе гомологов использовались следующие критерии:
- База данных - RefSeq
- E-value ≤ 1E-30 (лучший был около 1E-160)
- Организмы должны быть из разных филумов
- Покрытие выравнивания должно составлять не менее 90%
- AC преимущественно должен начинаться на 'NP_' (найдено 6)
E.coli относится к γ-протеобактериям, однако, т.к. филум протеобактерий довольно обширный, а выборка должна быть в меру однородной, то среди прочих была также выбрана α-протеобактерия Sinorhizobium meliloti. Также был проведён поиск только по эукариотам, в результате чего найдены 2 гомолога (правда, со сноской PREDICTED): муха Ceratitis capitata (E-value 2e-98, покрытие 97%) и огурец обыкновенный (E-value 9e-60, покрытие 86%). Может быть, это и ошибка секвенирования, но я их включил в рассмотрение. Все последовательности записаны в fasta-файл.
Анализ структуры белка 3L1L
На сервере OPM был найден белок 3L1L, откуда о нём собрана информация и помещена в таблицу 2. На сервере TCBD белка не оказалось, поэтому на OPM был взят самый близкий из TC-кодов.
| PDB ID | Организм | Тип мембраны | TC-код | Угол наклона спиралей к нормали | Количество трансмембранных спиралей |
|---|---|---|---|---|---|
| 3L1L | Escherichia coli | Внутренняя мембрана Грам-отрицательной бактерии | 2.A.3 | 2 ± 2° | 24 (в обеих субъединицах) |
Расшифровка полей TC:
- 2: транспортёры, движимые электрохимическим потенциалом
- 2.А: транспортёры, осуществляющие симпорт, антипорт или унипорт
- 2.А.3: надсемейство амино APC (Acid-Polyamine-Organocation). Сложно сказать по-русски, поэтому "органические катионы-полиамины с кислотной группой"
Анализ множественного выравнивания трансмембранных белков
На сервере EBI программой muscle было произведено множественное выравнивание последовательностей найденных гомологов - представлено в fasta-файле а также на рис. 1. Затем к выравниванию была добавлена строка аннотации "TM_REAL", в которой отмечены все α-спирали (по данным pdb-файла). Однако при текущем выравнивании 2 спирали попадали на гэп, от чего казались больше нужного. Из-за этого выравнивание было изменено средствами Jalview - перенесены начала спиралей на конец гэпов (см. рис. 2)
Один из гомологов, а именно белок бактерии Sinorhizobium meliloti был проанализирован программой TMHMM (на последнем выравнивании второй сверху). Результат представлен на рис. 3. Найденные трансмембранные участки были отражены в аннотации выравнивания "TM_PREDICTED". В аннотации "TM_REAL" оставлены только трансмембранные участки спиралей для возможности сравнения работы программы. Также к названиям строк последовательнотсей были добавлены названия организмов. Полученное выравнивание представлено на рис. 4 и в проекте Jalview.
Выдача программы TMHMM (click me to view):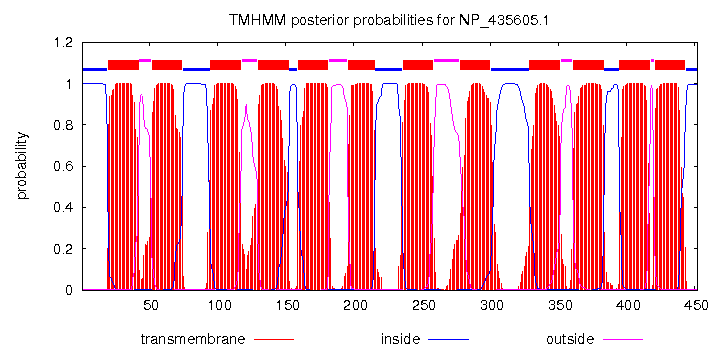
Рис. 3 Результат анализа последовательности белка Q92ZU0_RHIME программой TMHMM. Красным выделены трансмембранные участки, синим - обращённые внутрь клетки, magenta - направлены из клетки.

Рис. 4 Аннотированное выравнивание с окраской по степени гидрофобности: градиент от красного к синему соответсвует переходу от наиболее гидрофобного к наиболее гидрофильному. Порог консервативности равен 15%.
В соотвествии с раскраской выравнивания на рис. 4 приведена пространственная структура белка ADIC_ECO57 (см. рис. 5).
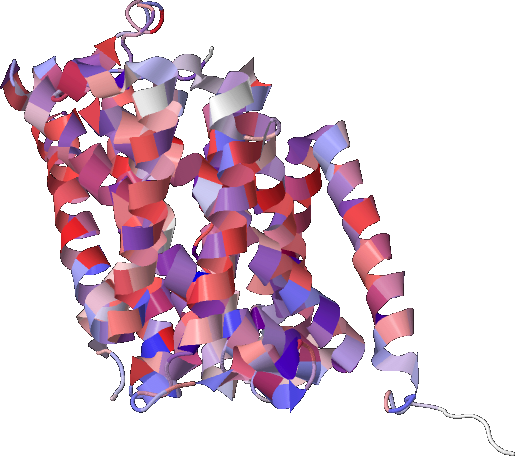
Рис. 5 Пространственная структура белка ADIC_ECO57 с раскраской, аналогичной таковой на рис. 4.
Обсуждение
Согласно рисунку 1, трансмембранные спирали обладают высокой консервативностью, в отличие от петель между ними. N- и C-концы белков варьируют наиболее сильно как в длине, так и в аминокислотной последовательности. В последовательностях α-спиралей преобладают такие неполярные аминокислоты, как V, I, L, A. Однако в них встречаются очень консервативные позиции гидрофильных аминокислот: Tyr239, Tyr365, Tyr424 (по E.coli). Они могут быть нужны для транспорта положительно заряженного аргинина через мембрану. Петли, тем не менее тоже обладают очень консервативными позициями, обычно пролина или глицина, т.к. эти аминокислоты позволяют аминокислотной цепочке совершить резкий поворот - Gly71, Gly72, Pro261 (по E.coli). Это предположение хорошо согласуется с тем, что рассматриваемый белок содержит очень много спиралей и коротких петель между ними.
По рисунку 4 можно сделать вывод о работе программы TMHMM. C рассматриваемыми белками программа справилась на приемлемом уровне. Она предсказала все трансмембранные участки. Также программой не сделано лишних и неверных предсказаний. На сколько я понимаю, случай с рассматриваемым белком довольно сложный для правильной работы программы: в структуре белка ADIC_ECO57 имеются такие структуры, как 2 α-спирали, идущие подряд по прямой или под углом; или же случай когда маленькая 310-спираль встраивается между двумя длинными α-спиралями наверняка должен мешать определению концов спирали.
Клетка размером с дыню
Допустим гипотетический эксперимент: увеличим клетку до размеров дыни. Размеры бактерий в среднем составляют 0,5—5 мкм, т.е. в среднем 2 мкм. Размер дыни возьмём равным 20 см. Толщина клеточный мембраны составлет 40 Å. Тогда бактерия, увеличенная до размеров дыни будет иметь плазмалемму толщиной:
$$\frac{20\cdot10^{-2}\cdot40\cdot10^{-10}}{2\cdot10^{-6}} = 4\cdot10^{-4}\;м = 0.4 \text{ мм}$$
Таким образом, мембрану клетки размером с дыню по толщине можно сравнить с половиной толщины её кожицы.
Positive inside
На полученной выборке гомологов была произведена попытка проверить правило "positive inside": положительные аминокислоты (K,R,H) были раскрашены в красный, отрицательные (D,E) - в синий. Результат представлен на рис. 6.

Рис. 6 Раскраска выравнивания по зараядам. В красные прямоугольники выделены области, обращённые внутрь клетки, в синие - наружу.
В общих чертах правило выполняется: внутрь клетки как правило обращены положительные аминокислоты. В связи со структурой белка, из клетки почти не выходят аминокислотные петли, поэтому в половине случаев в синих прямоугольниках действительно больше отрицательных аминокислот, а в некоторых - поровну. Но в оригинале правила про внешнюю сторону ничего и не говорится.

