Создание поверхностей
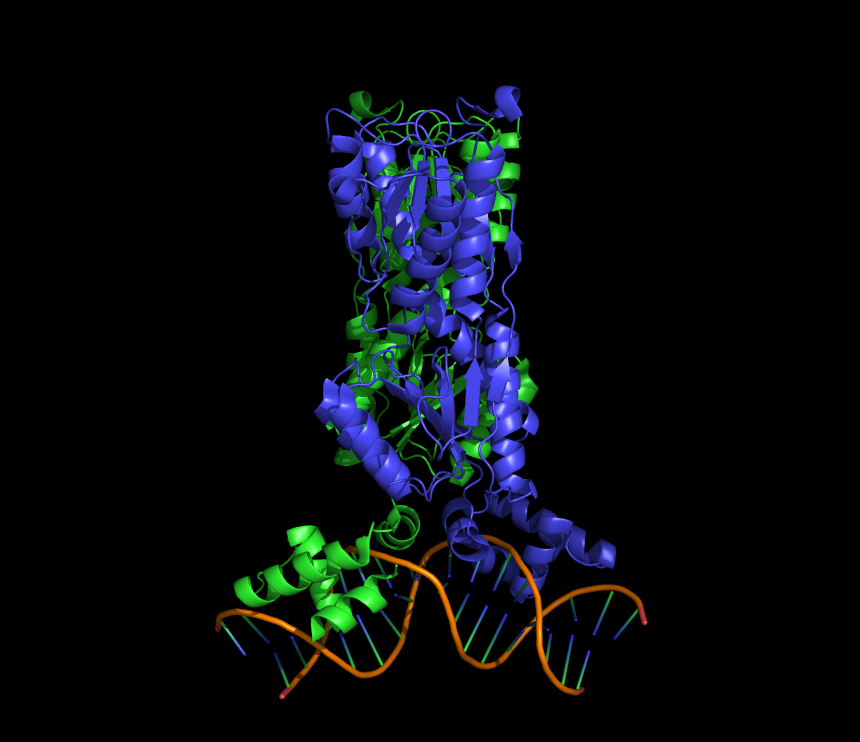
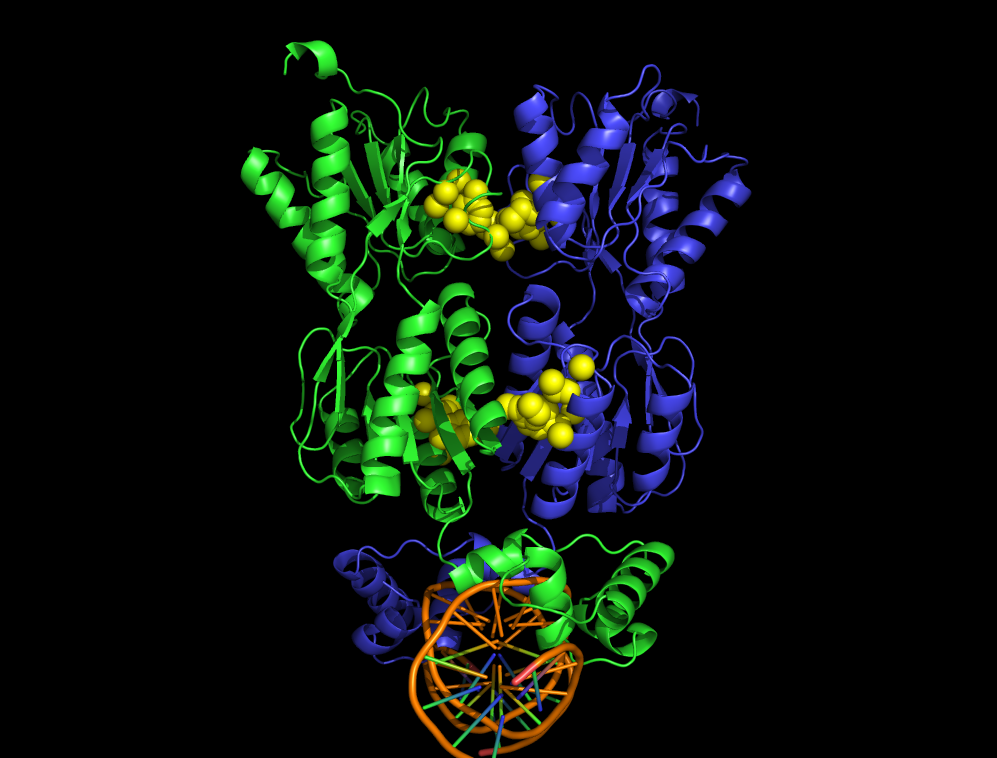
Для анализа был взят комплекс пуринового репрессора с ДНК 1QPZ. Данная структура была расширена до димера с помощью команды PyMol: symexp sym, 1qpz, all, 2.5 (см. рис. 1).
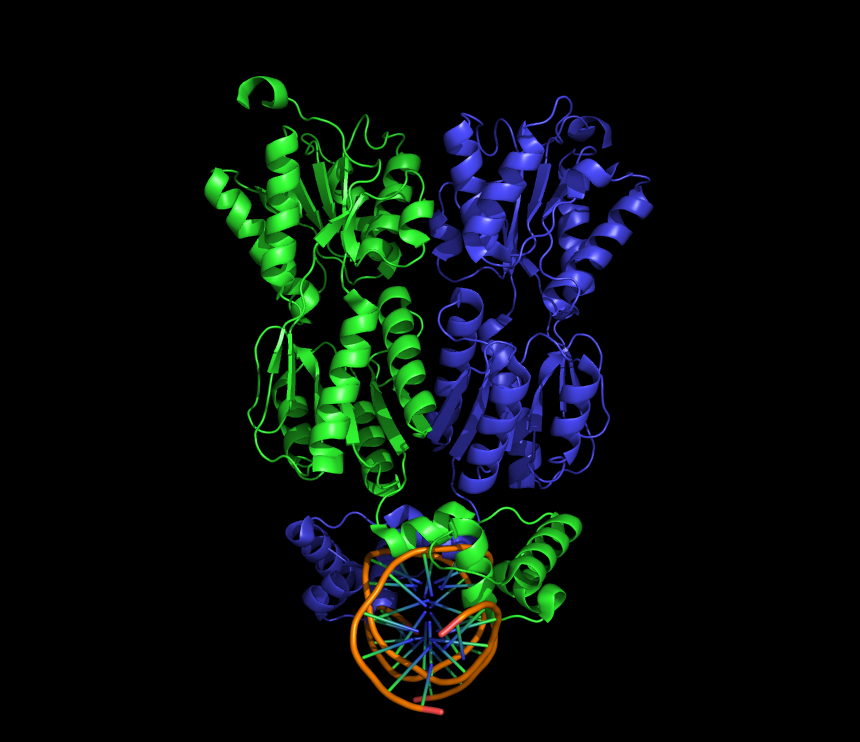
Поверхность контакта мономера с мономером
Для адекватной прорисоки поверхностей необходимо каждое из выделений держать в виде отдельного объекта PyMol
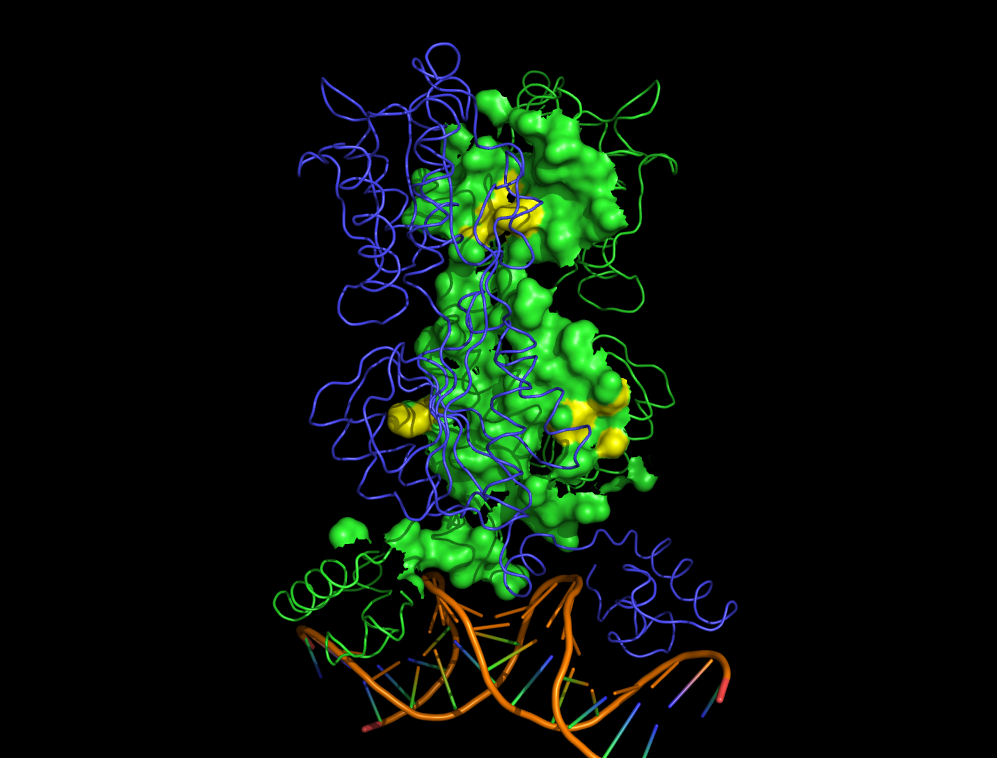
Для выделения контактов мономера белка с симметричным мономером на расстоянии 5Å использовалась команда PyMol: select contact1, /1qpz//A near_to 5 of /sym02000000//A. Полученное выделение представлено в виде поверхности (см. рис. 2).
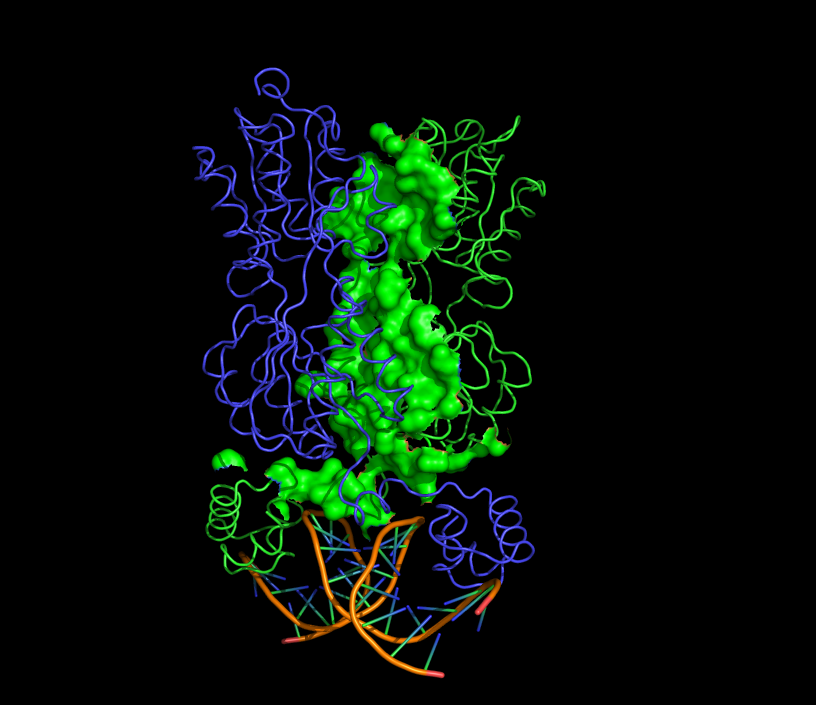
Поверхность контакта димера с ДНК
Аналогично были выделены контакты димера белков с двойной спиралью ДНК на расстоянии 5Å с помощью PyMol. Полученное выделение представлено в виде поверхности (см. рис. 3).
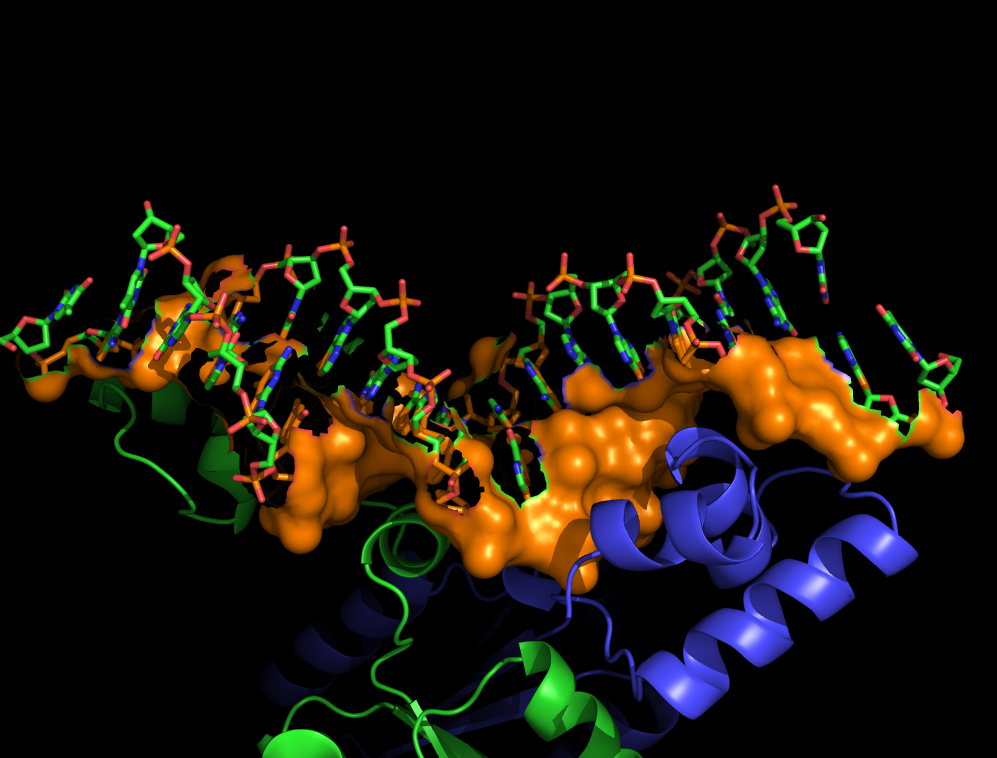
Поверхность контакта ДНК с димером
Также были выделены контакты ДНК с димером белков на расстоянии 5Å с помощью PyMol. Полученное выделение представлено в виде поверхности (см. рис. 4).
Создание поверхности с гидрофобными кластерами
С помощью сервиса CluD с порогом расстояния 4.5Å были найдены гидрофобные кластеры на интерфейсе мономеров белка - RasMol script  (см. рис. 5).
(см. рис. 5).
Были выделены контакты мономера белка с симметричным мономером на расстоянии 5Å с помощью PyMol. Полученное выделение представлено в виде поверхности (см. рис. 6).