Выбор белка
Для анализа электронной плотности был выбран белок из первого семестра: супероксид-дисимутаза-подобный белок из Bacilus subtilis, PDB-код 1XTM (вернее его мутант Y88H-P104H).
R-Value Work - насколько хорошо дифракция модели совпадает с экспериментальными данными. В идеале - 0, обычно ~ 0.2, для случайного набора атомов - 0.63
R-Value Free - менее смещённая мера; перед уточнением модели изымаем 10% атомов, а после - смотрим, на сколько хорошо уточнённая модель описывает те 10%. Обычно - 0.26
Экспериментальные данные:
- Разрешение: 1.6 Å
- R-Value Work: 0.253
- R-Value Free: 0.266
- Space Group: P 21 21 2 (кристаллографическая группа)
По результатам PDBeFold для данного белка имеется большое количество (553) структурных гомологов с RMSD 0.8-3.0 (оценка сходства) и значением N_align 58-152 (число сопоставленных остатков).
Изображение электронной плотности
Визуализация электронной плотности проводилась с помощью PyMol. Загрузка файла omap осуществлялась командой:
load /pathway/1xtm.omap, 1xtm_map
вокруг полипептидной цепи
Изображение электронной плотности вокруг остова цепи А структуры 1XTM проводилось с 2 уровнями подрезки: 1 (рис. 1.1) и 0.5 (рис. 1.2) с помощью команд:
isomesh new_map, 1xtm_map, 1, chain A and (name ca+c+n+o) and !het, carve=2isomesh new_map, 1xtm_map, 0.5, chain A and (name ca+c+n+o) and !het, carve=2
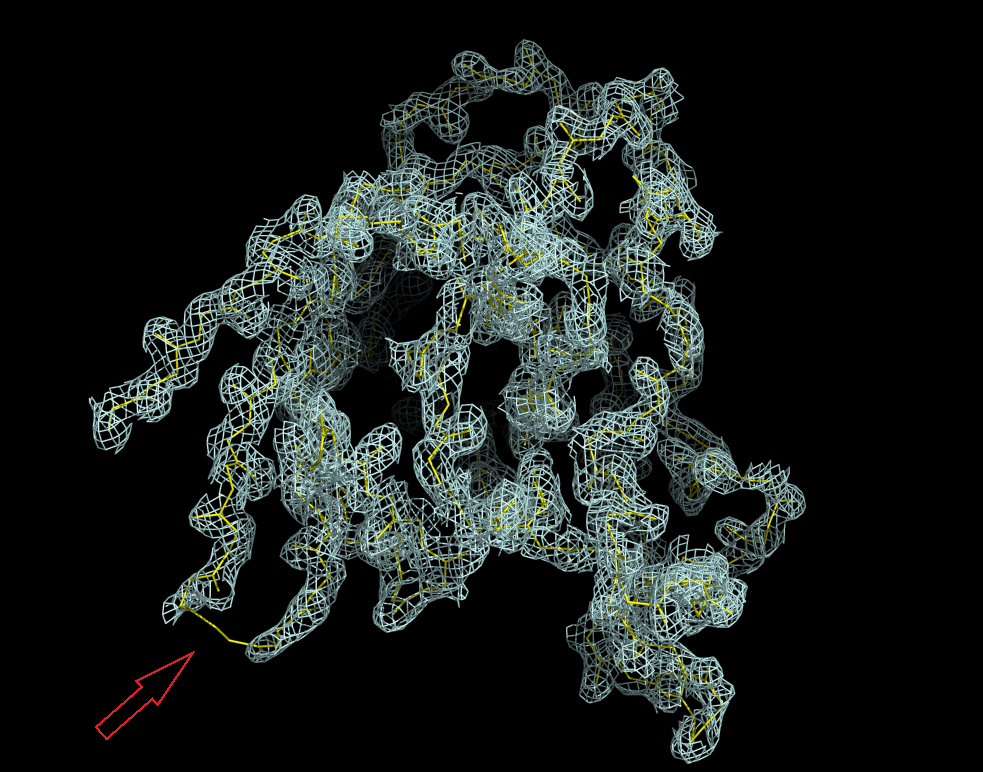 Рисунок 1.1. Изображение электронной плотности (σ=1) вокруг полипептидной цепи без боковых радикалов. Красной стрелкой указан участок, электронную плотность которого не видно (2 аспартата А:64-65).
Рисунок 1.1. Изображение электронной плотности (σ=1) вокруг полипептидной цепи без боковых радикалов. Красной стрелкой указан участок, электронную плотность которого не видно (2 аспартата А:64-65).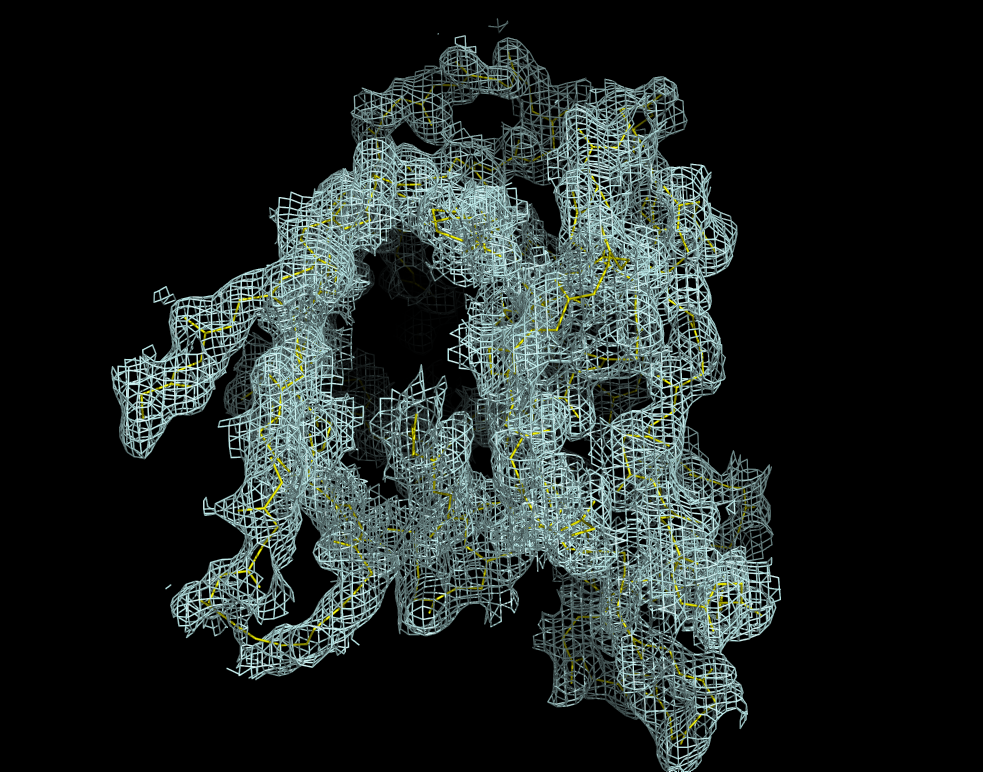 Рисунок 1.2. Изображение электронной плотности (σ=0.5) вокруг полипептидной цепи без боковых радикалов.
Рисунок 1.2. Изображение электронной плотности (σ=0.5) вокруг полипептидной цепи без боковых радикалов.Уровень подрезки σ=1 является наиболее удобным для визуализации электронной плотности, однако при таком пороге электронную плотность некоторых фрагментов белка не видно. Уровень подрезки σ=0.5 является более слабым, от чего отображение электронной плотности стало объёмнее, но даже теперь для участка, выделенного на рисунке 1.1 красной стрелкой, электронная плотность отображена не полностью.
Однако, если уменьшить уровень подрезки до σ=0.2, то электронная плотность как раз описывает 2 аспартата (рис. 2). Как бы то ни было, данный участок является слабым местом экспериментальных данных.
вокруг аминокислотных остатков
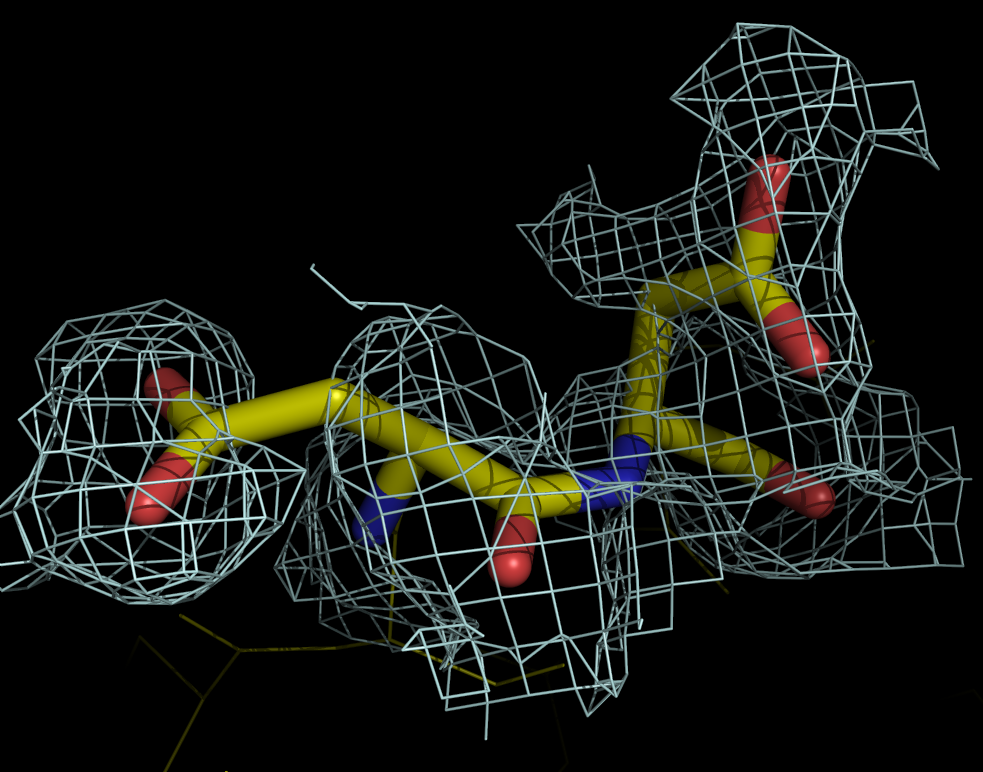
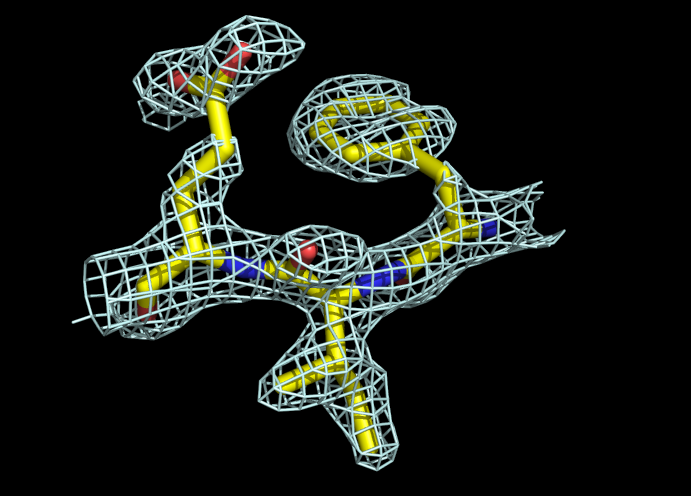
Для визуализации электронной плотности вокруг аминокислотных отстатков был выбран фрагмент Phe57-Ile58-Glu59. На уровне подрезки σ=1.5 (рис. 3.1) хорошо видно, как электронная плотность описывает структуру выбранных аминокислот. Причём даже заметно, что электронная плотность на глутамате смещена от Сγ к карбоксильной группе, а на фенилаланине - от Сβ к бензольному кольцу.
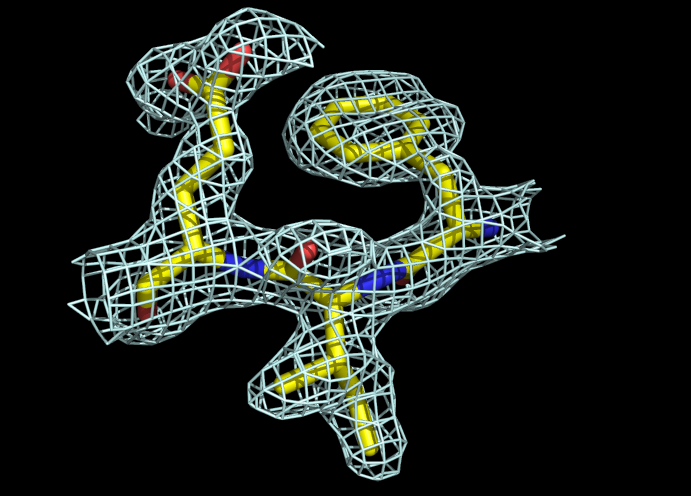
Чтобы убедиться, что атомы Сγ (Glu) и Сβ (Phe) не лишены электронной плотности, уровень подрезки был уменьшен до σ=1 (рис. 3.2). Как и должно быть, электронная плотность присутсвует у каждого атома.
В заключение можно сказать, что координаты атомов данной модели структуры 1XTM соответствуют сгущениям электронной плотности даже в слабых местах (A:64-65). Разрешение структуры, конечно, атомарным не является, но оно более, чем приемлемо.