Задание №1
В задании требовалось построить модели структур A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA.
С помощью программы Рutty перейдем в рабочую директорию:
cd term3/block1/pr2
Используем следующие команды для указания пути к 3DNA:
export PATH=${PATH}:/home/preps/golovin/progs/X3DNA/bin
export X3DNA=/home/preps/golovin/progs/X3DNA
С помощью программы fiber пакета 3DNA получим A-, B- и Z-форму дуплекса ДНК,
последовательность одной из нитей которого представляет собой 5 раз повторенную последовательность "gatc":
fiber -a gatc-a.pdb(А-форма дуплекса в PDB)
fiber -b gatc-b.pdb(B-форма дуплекса в PDB)
fiber -z gatc-z.pdb(Z-форма дуплекса в PDB)
Задание №2
Упражнение №1
В этом упражнении, используя файл А-формы, созданный в задании 1, требуется с помощью JMol выделить цветом или способом отображения:
1. Сахарофосфатный остов ДНК;2. Все нуклеотиды;
3. Все аденины;
4. Атом N7 во всех гуанинах и/или только в первом по последовательности.
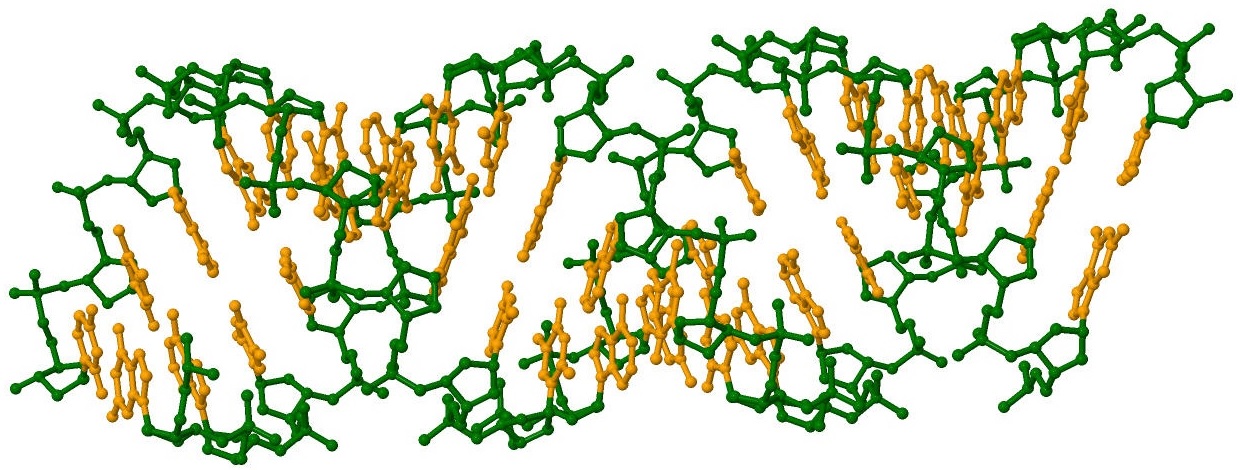 Рисунок 1. Дуплекс А-формы ДНК. Зеленым цветом отмечен
сахарофосфатный остов, а желтым - нуклеотиды.
Рисунок 1. Дуплекс А-формы ДНК. Зеленым цветом отмечен
сахарофосфатный остов, а желтым - нуклеотиды.
На рисунке 2 отмечены все аденины дуплекса ДНК:
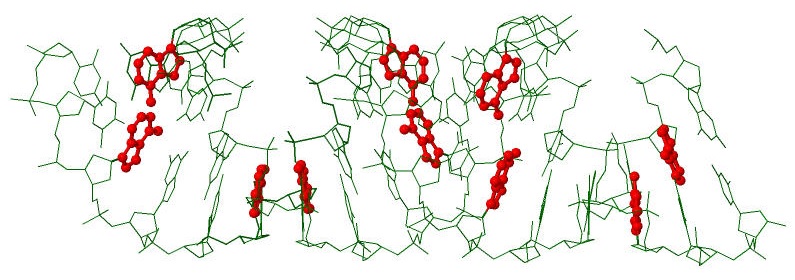 Рисунок 2. Все аденины отмечены красным цветом.
Рисунок 2. Все аденины отмечены красным цветом.
На рисунке 3 отмечены седьмые атомы азота гуанина:
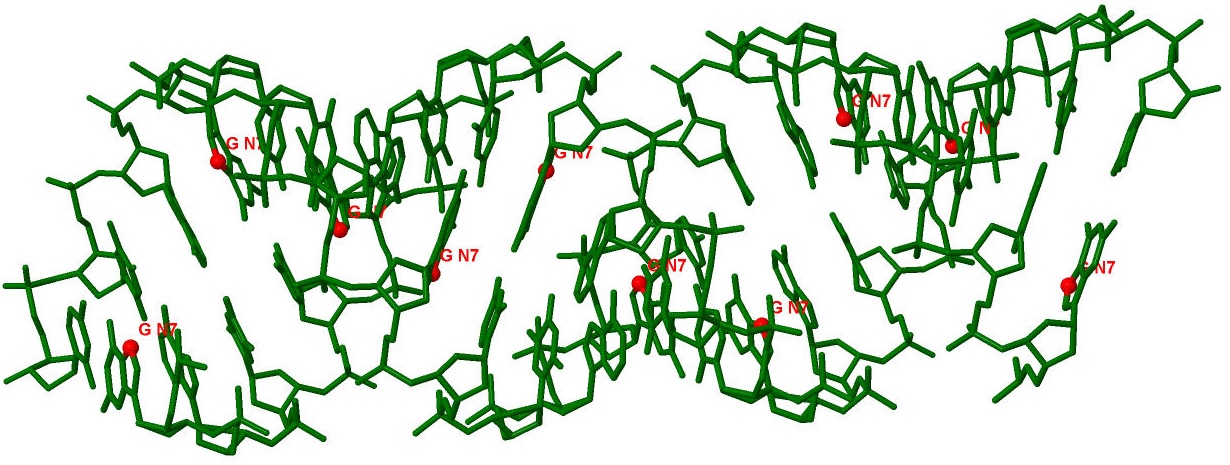 Рисунок 3. Атомы N7 каждого гуанина отмечены красным цветом.
Рисунок 3. Атомы N7 каждого гуанина отмечены красным цветом.
Упражнение №2
В упражнении треовалось найти файлы PDB на сайте www.rcsb.org:
1. 1LQ1 (скачать в формате PDB)2. 1C0A (скачать в формате PDB)
1.1LQ1. DNA Complexed Structure of the Key Transcription Factor Initiating Development in Sporulation Bacteria
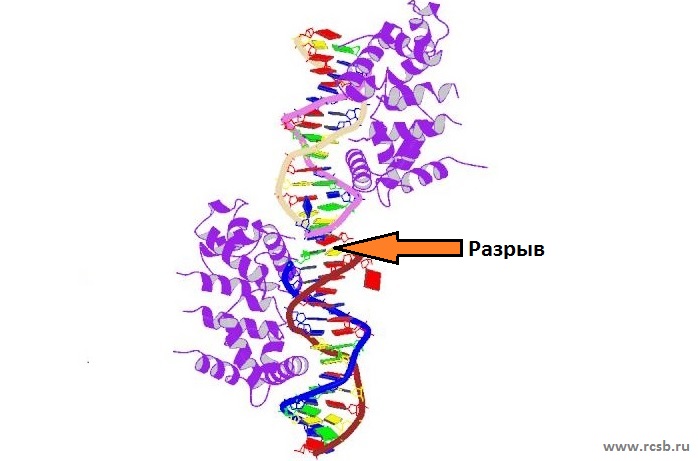 Рисунок 4. Структура ключевого фактора транскрипции, инициирующей развитие
в спорулирующей бактерии в комплексе с ДНК.
Рисунок 4. Структура ключевого фактора транскрипции, инициирующей развитие
в спорулирующей бактерии в комплексе с ДНК.
2.1C0A. Crystal Structure of the E. Coli Aspartyl-tRNA Synthetase : TRNAASP : Aspartyl-Adenylate Complex
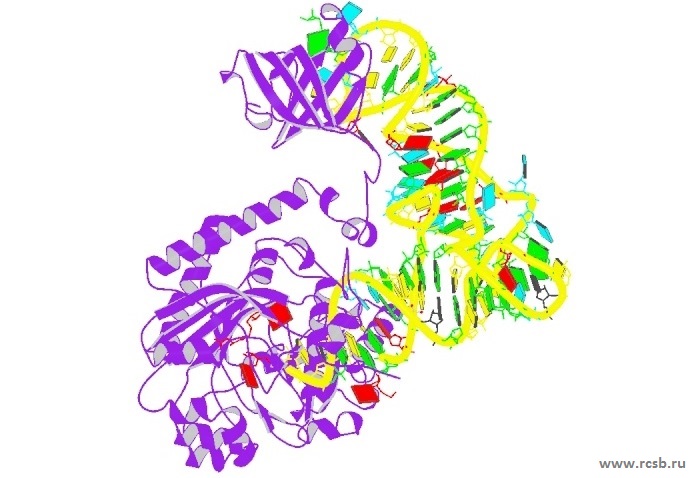 Рисунок 5. Кристальная структура аспартил-тРНК синтетазы E. Coli.
Рисунок 5. Кристальная структура аспартил-тРНК синтетазы E. Coli.
Упражнение №3
Привожу PDB файлы с координатами атомов только ДНК и РНК, соответственно:
1. 1LQ1 (скачать в формате PDB)2. 1C0A (скачать в формате PDB)
На рисунке 6 показан двойной разрыв дуплекса ДНК:
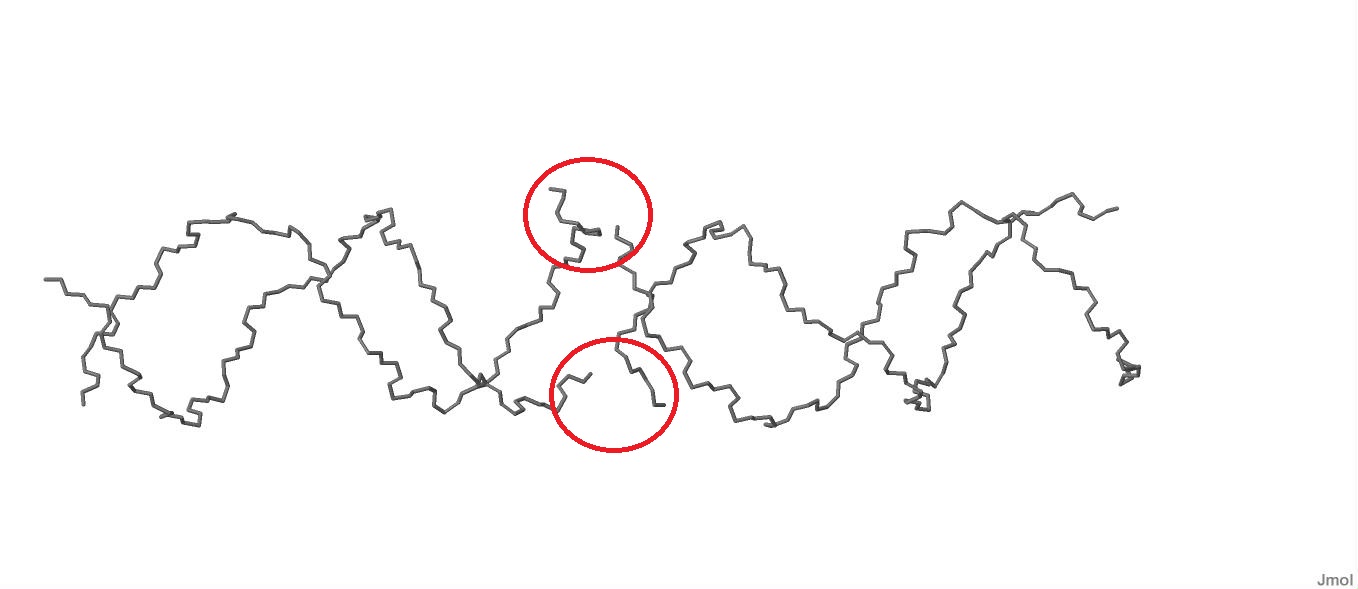 Рисунок 6. Изображение ДНК в проволочной структуре. Имеется двойной разрыв дуплекса.
Рисунок 6. Изображение ДНК в проволочной структуре. Имеется двойной разрыв дуплекса.
В структуре тРНК разрывов не нашлось:
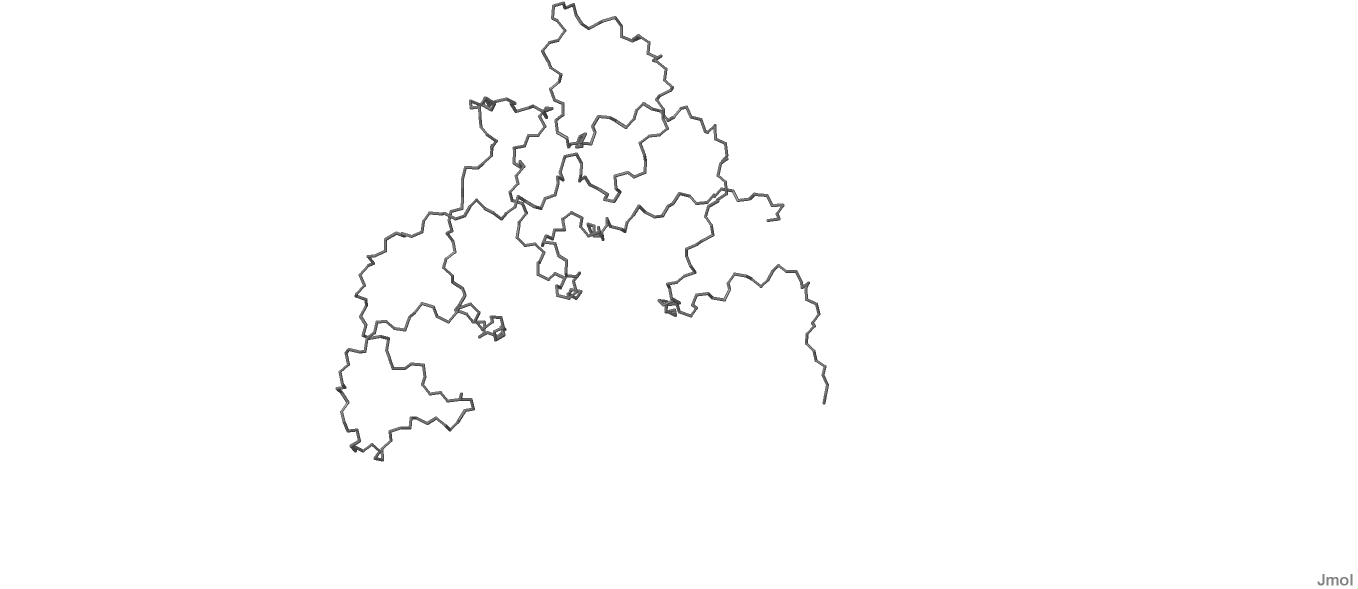 Рисунок 7. Изображение тРНК в проволочной структуре. Разрывов не имеется.
Рисунок 7. Изображение тРНК в проволочной структуре. Разрывов не имеется.
Задание №3
Упражнение №1
Определим, какие атомы цитозина в положении 12 В-формы ДНК, полученного в первом задании, обращены в сторону большой бороздки, а какие в стоону малой:
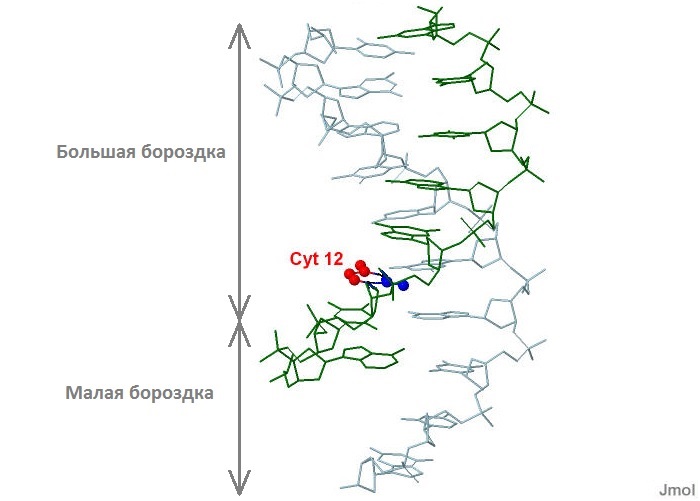 Рисунок 8. Участок дуплекса В-формы ДНК. Красным цветом
выделены атомы, обращенные в сторону большой бороздки, а синим - в сторону малой.
Рисунок 8. Участок дуплекса В-формы ДНК. Красным цветом
выделены атомы, обращенные в сторону большой бороздки, а синим - в сторону малой.
С помощью ChemSketch получим изображение цитозина, выделим красным цветом атомы, смотрящие в сторону большой бороздки, а синим - в сторону малой:
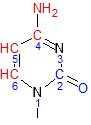 Рисунок 9. Цитозин в положении 12 в В-форме ДНК.
Рисунок 9. Цитозин в положении 12 в В-форме ДНК.
В сторону большой бороздки обращены атомы с12.с4, с12.n4, с12.с5, с12.с6;
В сторону малой бороздки обращены атомы c12.c2, c12.o2;
Остальные атомы основания: c12.n1 соединяется с сахарофосфатным остовом, c12.n3 образует водородную связь с гуанином.
То же самое наблюдается в А-форме:
 Рисунок 10. Цитозин в положении 12 в А-форме ДНК.
Рисунок 10. Цитозин в положении 12 в А-форме ДНК.
Однако в Z-форме все обстоит с точностью да наоборот. Обращенность атомов цитозина Z-формы ДНК в сторону большой и малой бороздок инвертирована по сравнению с двумя предыдущими формами:
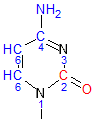 Рисунок 11. Цитозин в положении 12 в Z-форме ДНК.
Рисунок 11. Цитозин в положении 12 в Z-форме ДНК.
Однако следует заметить, что обращенность в сторону одной из бороздок и экспонированность - не одно и то же. К примеру, в А-форме с12.с2, как и с12.о2, обращен в сторону малой бороздки, но экспонирован только атом с12.о2. В большой же бороздке экспонированные атомы образуют специфические комбинации донорно-акцепторных сайтов, узнавемых различными белками.
Упражнение №2
Требуется сравнить основные спиральные параметры разных форм ДНК.
Для сравнения нужно использовать файлы, полученные при выполнении задания 1:
A-форма ДНК
Шаг спирали составляет 28.0Å, на один виток которой приходится 10 пар оснований:
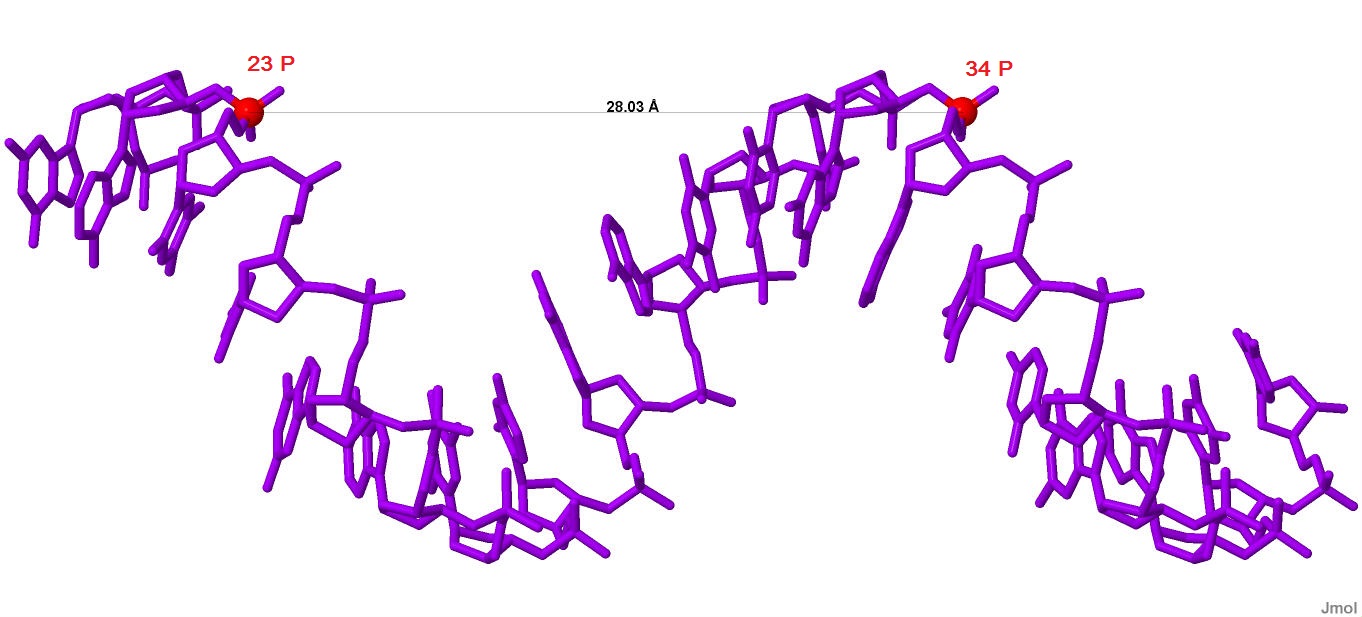 Рисунок 12. Шаг спирали A-ДНК. Красными шарами отмечены атомы фосфора.
Рисунок 12. Шаг спирали A-ДНК. Красными шарами отмечены атомы фосфора.
Ширина большой бороздки составляет 17.0Å, а малой 8.0Å:
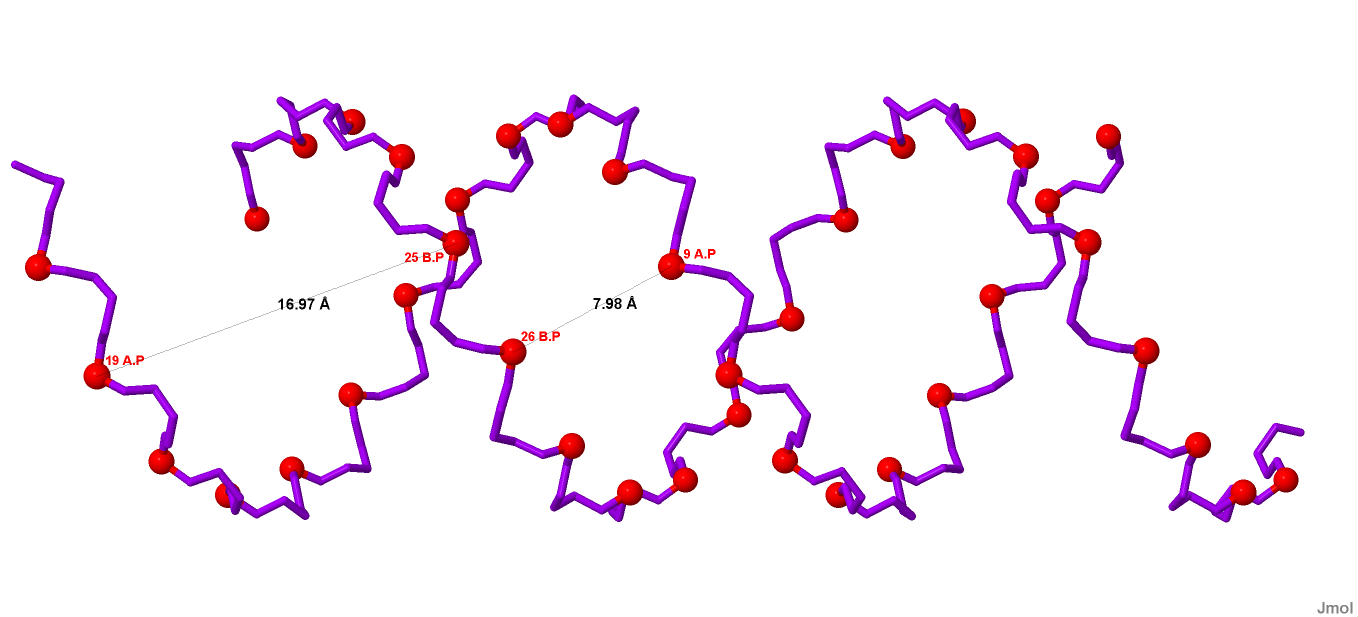 Рисунок 13. Ширина бороздок A-ДНК. Красными шарами отмечены атомы фосфора.
Рисунок 13. Ширина бороздок A-ДНК. Красными шарами отмечены атомы фосфора.
B-форма ДНК
Шаг спирали составляет 33.75Å, на один виток которой приходится 11 пар оснований:
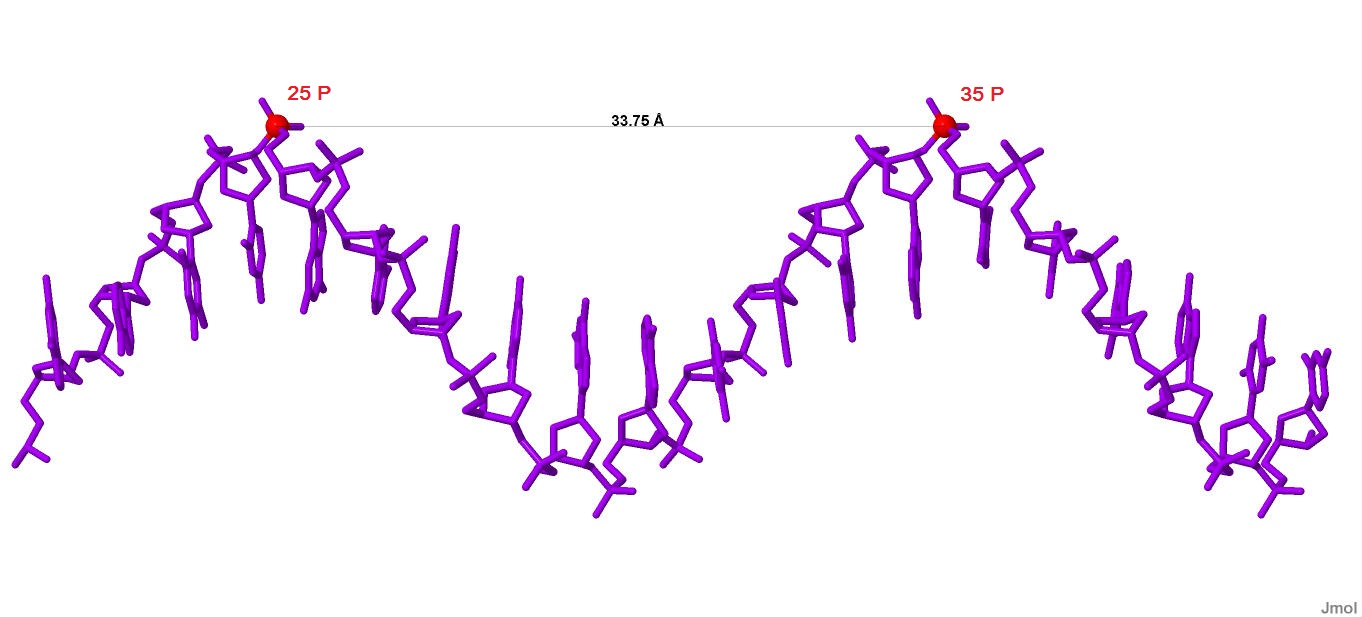 Рисунок 14. Шаг спирали В-ДНК. Красными шарами отмечены атомы фосфора.
Рисунок 14. Шаг спирали В-ДНК. Красными шарами отмечены атомы фосфора.
Ширина большой бороздки составляет 17.2Å, а малой 12.7Å:
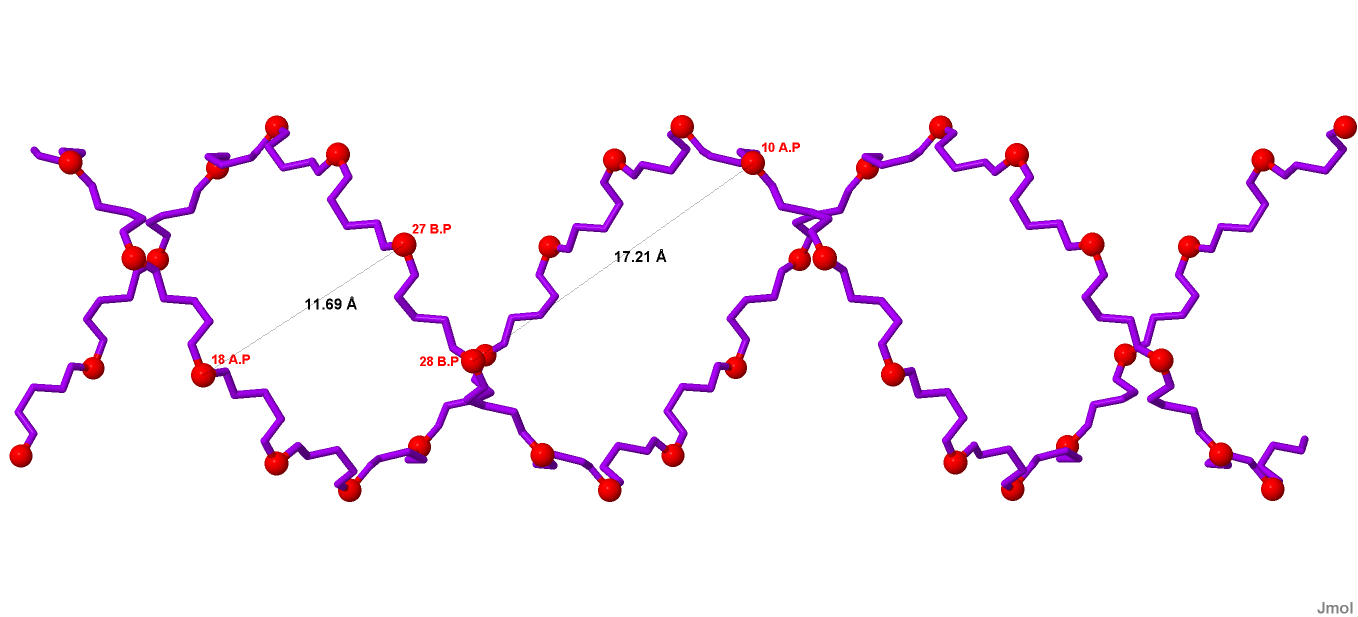 Рисунок 15. Ширина бороздок В-ДНК. Красными шарами отмечены атомы фосфора.
Рисунок 15. Ширина бороздок В-ДНК. Красными шарами отмечены атомы фосфора.
Z-форма ДНК
Z-форма ДНК, полученная в первом задании оказалась недостаточно длинной. Как видно на рисунке 16, виток из 10 пар оснований оказался неполным:
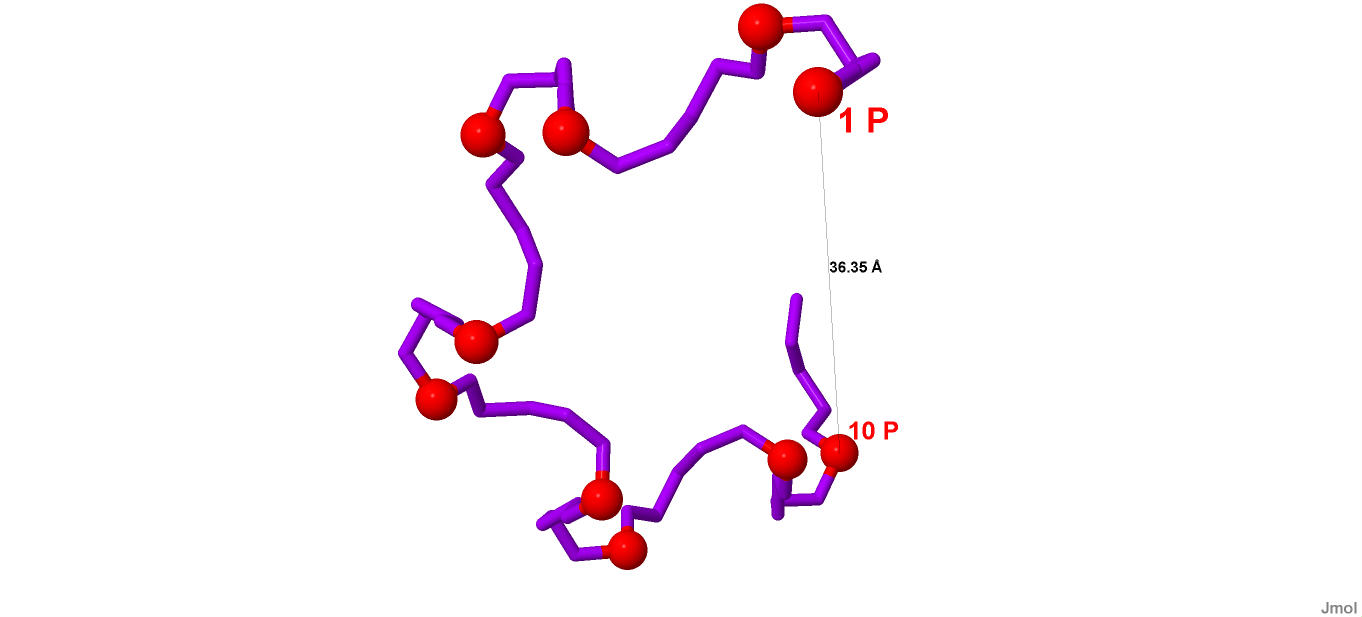 Рисунок 16. Вид сверху Z-формы ДНК, полученной в задании №1. Красными шарами отмечены атомы фосфора.
Рисунок 16. Вид сверху Z-формы ДНК, полученной в задании №1. Красными шарами отмечены атомы фосфора.
Прежде, чем померить шаг спирали с помощью той же программы fiber пакета 3DNA получим новый дуплекс Z-формы ДНК, с полным оборотом витка:
fiber -z gatc-z2.pdb(новая Z-форма дуплекса в PDB):
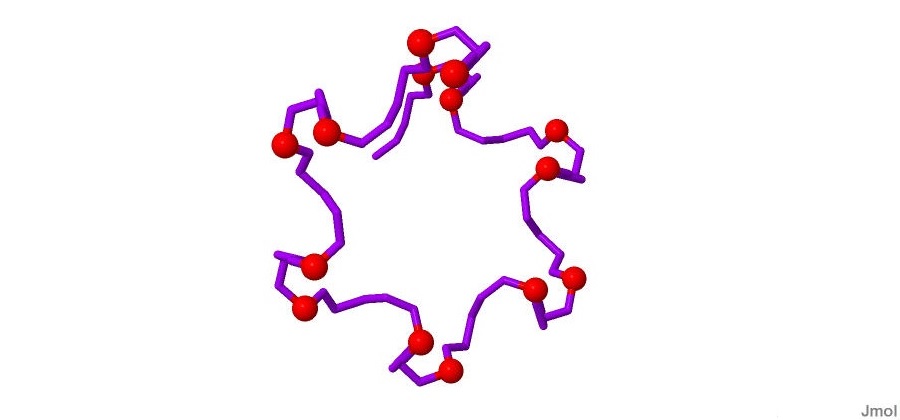 Рисунок 17. Новый дуплекс Z-ДНК с полным оборотом витка. Красными шарами отмечены атомы фосфора.
Рисунок 17. Новый дуплекс Z-ДНК с полным оборотом витка. Красными шарами отмечены атомы фосфора.
На нем и будем измерять нужные параметры. Длина полного оборота витка, образованного 12 парами оснований, составляет 43.5Å:
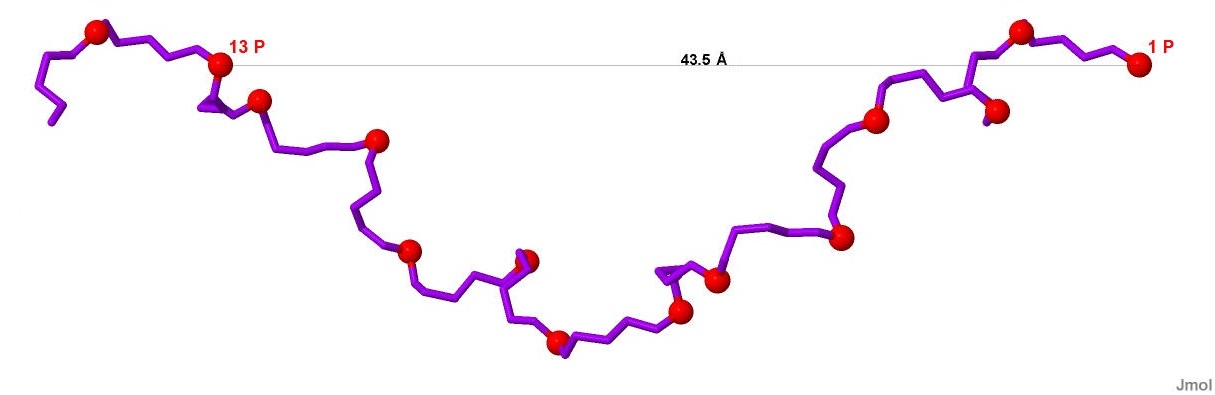 Рисунок 18. Шаг спирали Z-ДНК. Красными шарами отмечены атомы фосфора.
Рисунок 18. Шаг спирали Z-ДНК. Красными шарами отмечены атомы фосфора.
Ширина большой бороздки составляет 18.3Å, а малой 9.9Å:
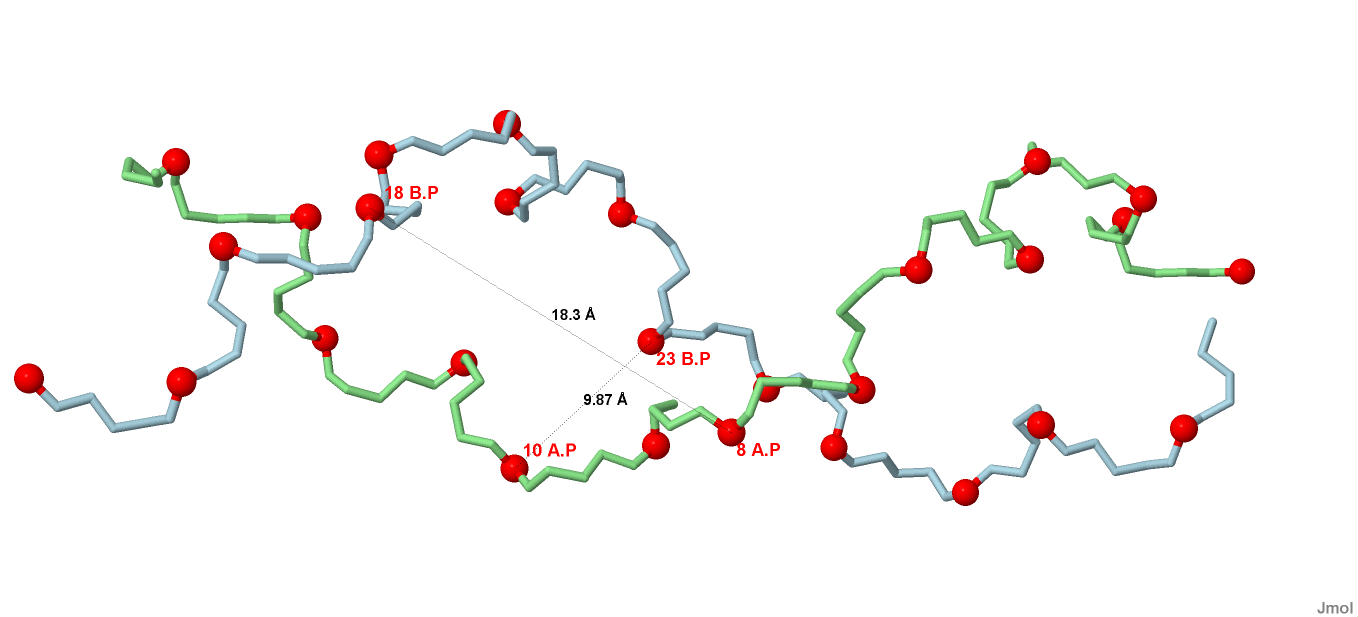 Рисунок 19. Ширина бороздок Z-ДНК. Красными шарами отмечены атомы фосфора.
Рисунок 19. Ширина бороздок Z-ДНК. Красными шарами отмечены атомы фосфора.
Сравнительная таблица геометрических параметров рассмотренных форм ДНК:
| Параметр | A-форма | B-форма | Z-форма |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Å) | 28.0 | 33.8 | 43.5 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) | 17.0 | 17.2 | 18.3 |
| Ширина малой бороздки (Å) | 8.0 | 11.7 | 9.9 |
Упражнение №3
Значения торсионных углов при цитозине в 12 положении A-формы ДНК:
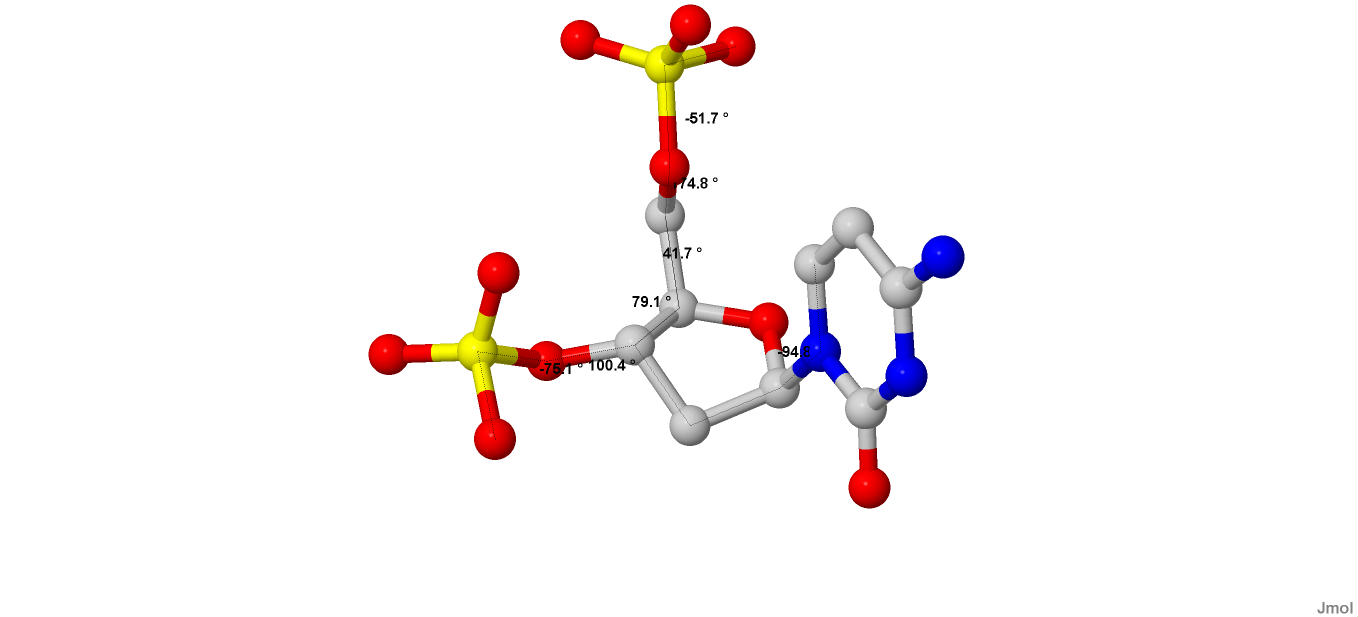 Рисунок 20. Цитозин в 12 положении A-формы ДНК.
Рисунок 20. Цитозин в 12 положении A-формы ДНК.
Значения торсионных углов при цитозине в 12 положении В-формы ДНК:
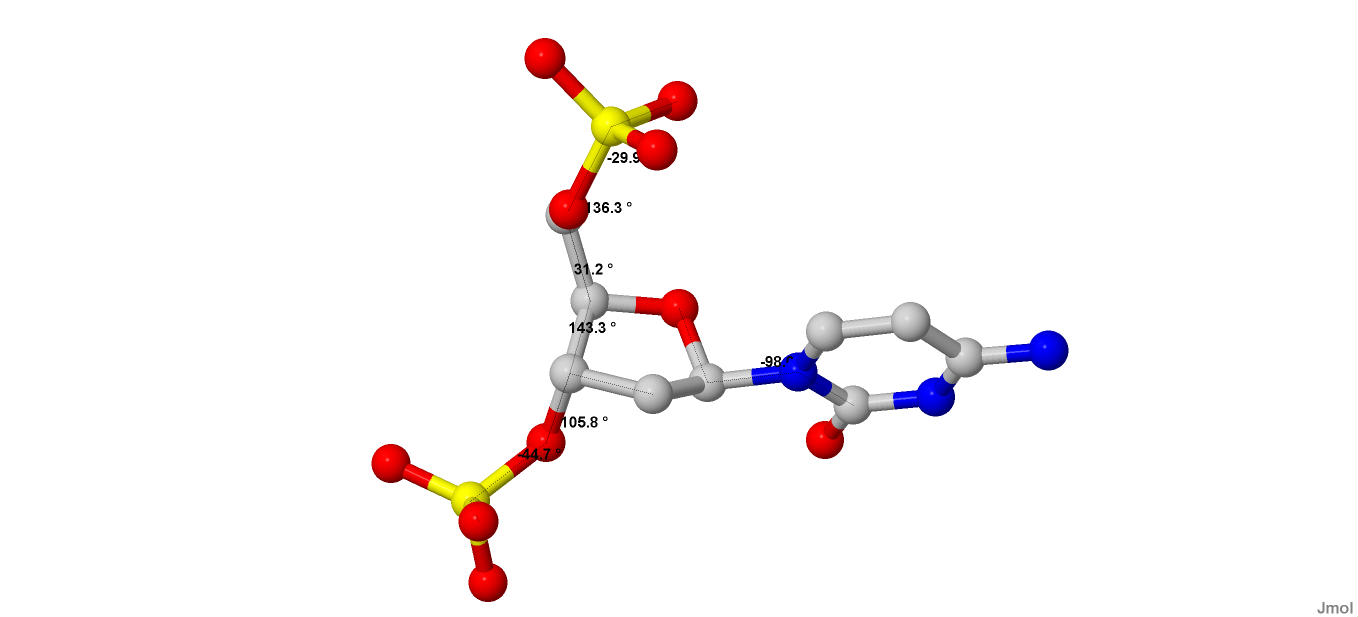 Рисунок 21. Цитозин в 12 положении В-формы ДНК.
Рисунок 21. Цитозин в 12 положении В-формы ДНК.
Сравнительная таблица значений торсионных углов при цитозине в 12 положении, А- и В-формы ДНК:
| Форма | α | β | γ | δ | ε | ζ | χ |
| А-ДНК | -51.7 | 174.8 | 41.7 | 79.1 | 100.4 | -75.1 | -94.8 |
| В-ДНК | -29.9 | 136.3 | 31.2 | 143.3 | 105.8 | -44.7 | -98.0 |
Значения торсионных углов, приведенных в презентации:
| Форма | α | β | γ | δ | ε | ζ | χ |
| А-ДНК | -62 | 173 | 52 | 88 или 3 | 178 | -50 | -160 |
| В-ДНК | -63 | 171 | 54 | 123 или 131 | 155 | -90 | -117 |
Задание №4
Упражнение №1
В упражнении требуется определить торсионные углы в заданной структуре ДНК. Торсионные углы представлены в электронной таблице в формате Excel.
Упражнение №2
Исследуемая транспортная РНК:
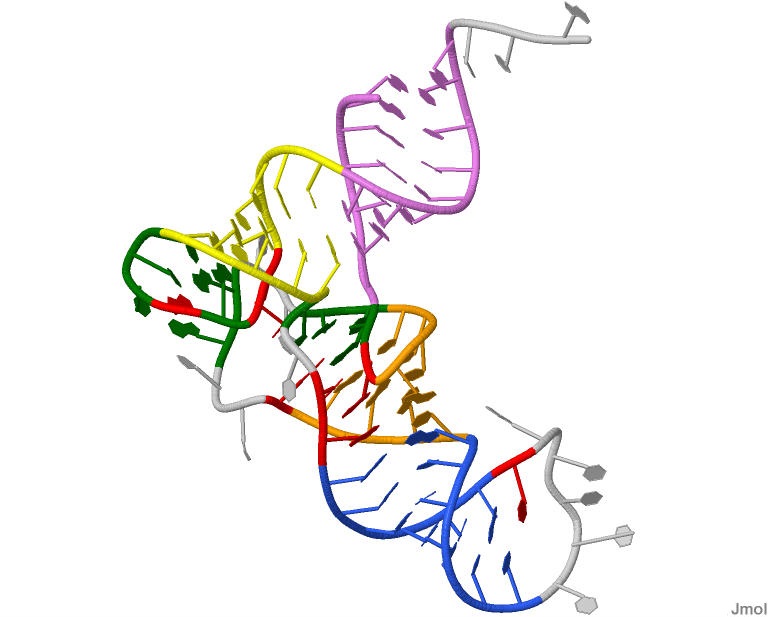 Рисунок 22. Исследумая тРНК. Пары оснований, входящие в
акцепторный стебель выделены фиолетовым, в Т-стебель - желтым, в D-стебель - оранжевым,
в антикодоновый стебель - синим; комплементарные пары, не имеющие отношения к стеблям
отмечены зеленым, а основания, которые между собой образут только стекинг-взаимодействия - красным;
основания, не образующие никаких взаимодействий с другими выделены серым цветом.
Рисунок 22. Исследумая тРНК. Пары оснований, входящие в
акцепторный стебель выделены фиолетовым, в Т-стебель - желтым, в D-стебель - оранжевым,
в антикодоновый стебель - синим; комплементарные пары, не имеющие отношения к стеблям
отмечены зеленым, а основания, которые между собой образут только стекинг-взаимодействия - красным;
основания, не образующие никаких взаимодействий с другими выделены серым цветом.
Акцепторный стебель состоит из участка 601-607 и комплементарного участка 672-666.
D-стебель состоит из участка 610-613 и комплементарного участка 625-622.
T-стебель состоит из участка 649-653 и комплементарного участка 665-661.
Антикодоновый стебель состоит из участка 638-644 и комплементарного участка 632-626.
Пары оснований, не образующие стебли во вторичной структуре заданной тРНК:
604_:[..G]:[..U]:.669
649_:[..G]:[PSU]:.665
654_:[5MU]:[..A]:.658
655_:[PSU]:[..G]:.618
638_:[..C]:[..C]:.632
644_:[..G]:[..A]:.626
610_:[..G]:[..U]:.625
614_:[..A]:[4SU]:.608
615_:[..G]:[..C]:.648
619_:[..G]:[..C]:.656
Дополнительные водородные связи в тРНК, стабилизирующие ее третичную структуру
654_:[5MU]:[..A]:.658
655_:[PSU]:[..G]:.618
614_:[..A]:[4SU]:.608
615_:[..G]:[..C]:.648
619_:[..G]:[..C]:.656
Неканонические пары оснований. В случае канонической пары было бы, например "А-----Т":
604_:[..G]G-*---U[..U]:.669
654_:[5MU]u-**-xA[..A]:.658
655_:[PSU]Px**+xG[..G]:.618
649_:[..G]G-*---P[PSU]:.665
644_:[..G]Gx*---A[..A]:.626
638_:[..C]C-*---C[..C]:.632
610_:[..G]G-*---U[..U]:.625
614_:[..A]A-**-xu[4SU]:.608
615_:[..G]Gx**+xC[..C]:.648
Упражнение №3
Стекинг-взаимодействия:
649_:[..G]G-*---P[PSU]:.665
650_:[..C]C-----G[..G]:.664
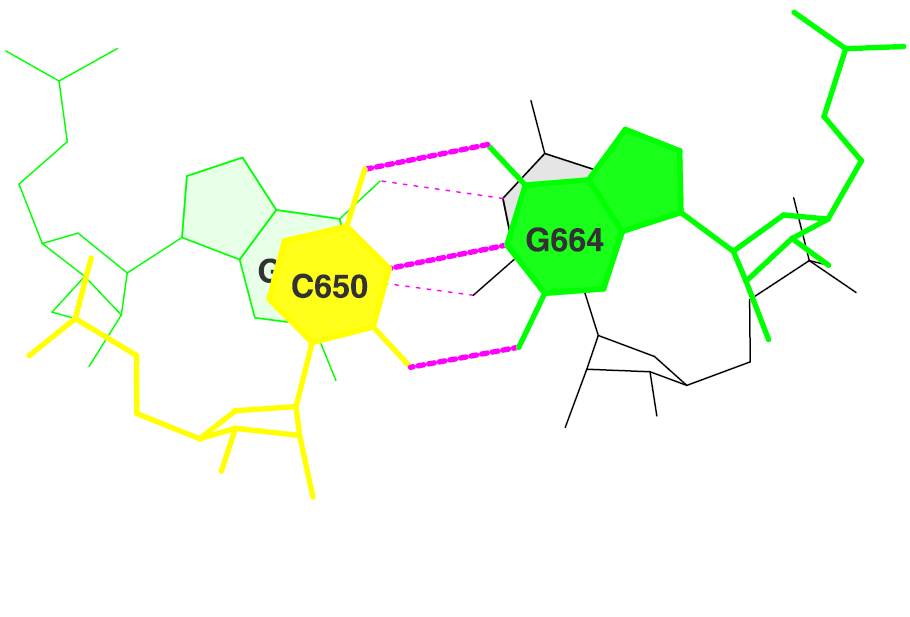 Рисунок 23. Две последовательные пары с наибольшим значением величины площади "перекрывания".
Рисунок 23. Две последовательные пары с наибольшим значением величины площади "перекрывания".