| ФББ МГУ | Корень | Обо мне | Семестры |
Рентгеноструктурный анализ
Изображение и анализ электронной плотности для PDB файла
Текст задания: практикум 1.
Сокращения
ЭП - электронная плотность
Введение
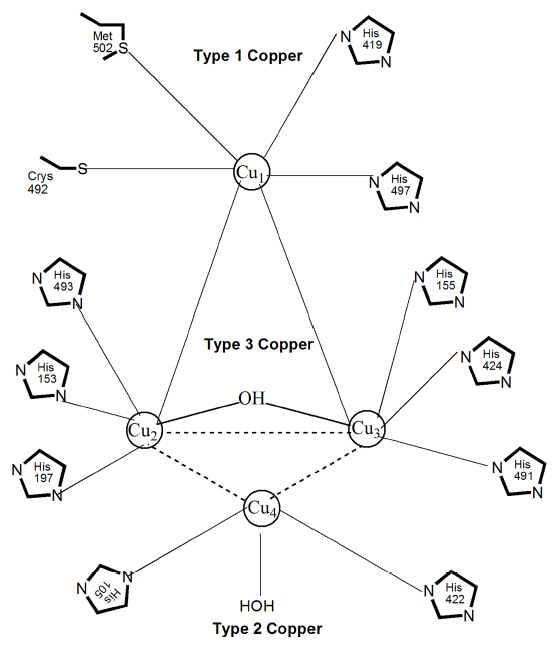
| Я выбрала запись PDB, удовлетворяющую требованиям. Это лакказа гриба Coriolopsis caperata, данный фермент имеет 4 координированных иона меди I, которые образуют сайты связывания субстрата трех типов: Т1, Т2 и Т3 (см рис 1). По каким-то причинам получить кристалл лакказы со всеми 4мя ионами меди сразу не получалось, поэтому был получен кристалл с 3мя ионами меди, который затем вымачивался в растворе CuCl, и из новоприсоединенных ионов меди был выбран тот, который вероятнее всего координирован не случайным образом, а в Т2 сайте (см рис 2). Лакказы относятся к классу оксидаз, они окисляют различные органические (полифенолы, естественный субстрат - клеточная стенка растений) и неорганические (ферроцианид, удобно изучать), используя в качестве акцептора электронов молукулу кислорода. |
Рисунок 1. Структура активного центра лакказы [1]. |
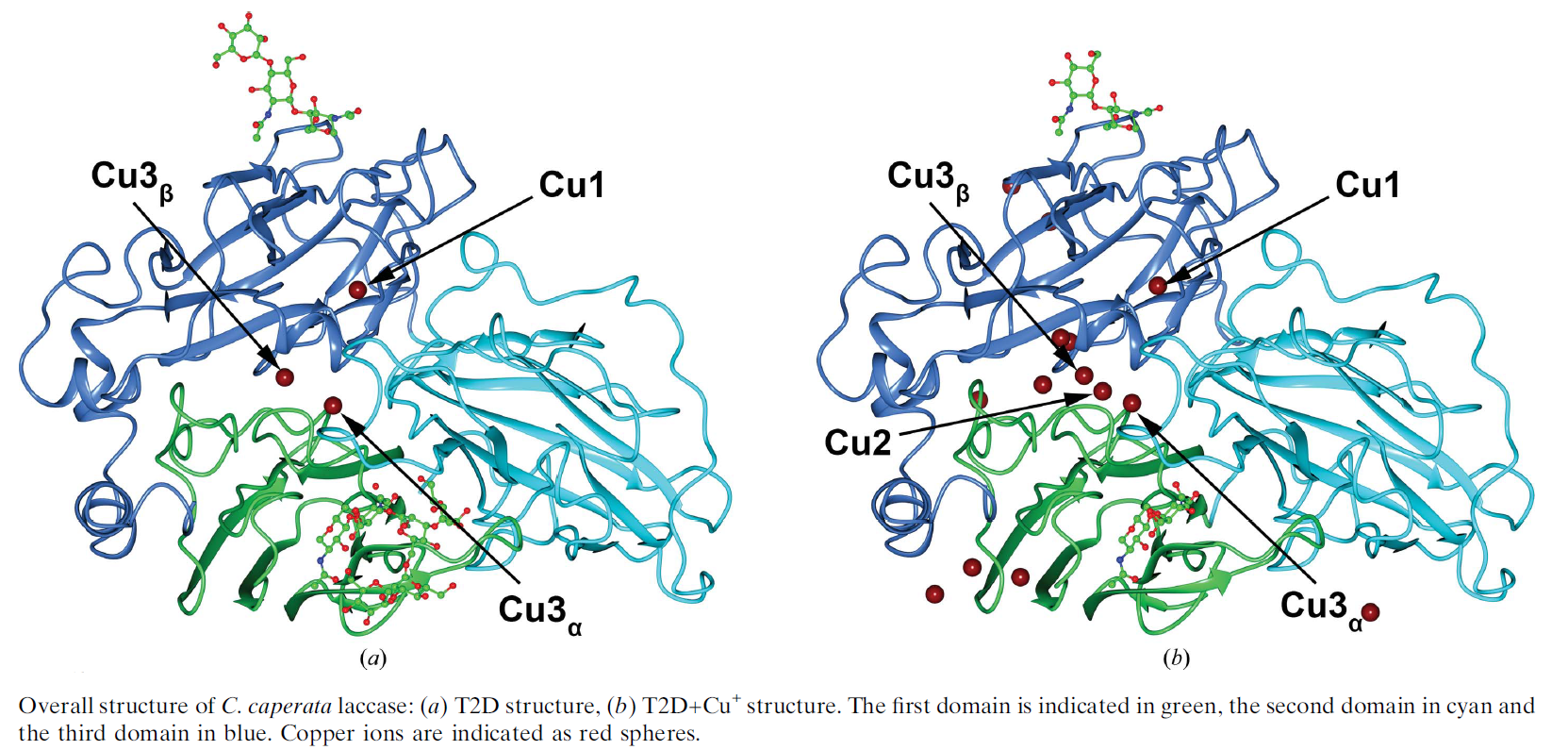
Рисунок 2. 3D структуры лакказы. Слева - без атома меди в Т2-сайте, справа - со множеством присоединившихся атомов меди, среди которых стрелкой указан тот, что предположительно находится в Т2-сайте [2].
Информация о выбранном объекте
- PDB код: 4jhu
- Название структуры: T2-depleted laccase from Coriolopsis caperata soaked with CuCl
- Разрешение 1.89 Å
- Публикация: http://dx.doi.org/10.1107/S1399004715001595
Электронная плотность
Для визуализации электронной плотности я загрузила PDB-структуру и данные об ЭП в PyMol, а затем построила ЭП вокруг остова полипептидной цепи всей молекулы белка (мономер) и вокруг трех отдельных аминокислот.
Z рассчитывается по формуле для ячейки кристалла М: Z = (R – M)/σ, где R — значение ЭП в данной точке пространства (x,y,z) в физических
единицах, σ - среднее квадратичное отклонение ЭП по ячейке кристалла, откуда получается, что Z — это значение ЭП в "сигмах" в той же точке.
Это значение я варьировала.
"Конспект" командной строки:
cd C:/my_directory load pdb4jhu.ent, pdb load 4jhu.ccp4, map hide all show sticks, backbone isomesh ED, map, 1.5, backbone, 1.5 ----- hide all show sticks, resi .. center resi .. isomesh ED1, map, 1.5, resi 257, 0.5 isomesh ED2, map, 1.5, resi 2 isomesh ED2, map, 1, resi 215, 1
Визуализация ЭП вокруг остова полипептидной цепи
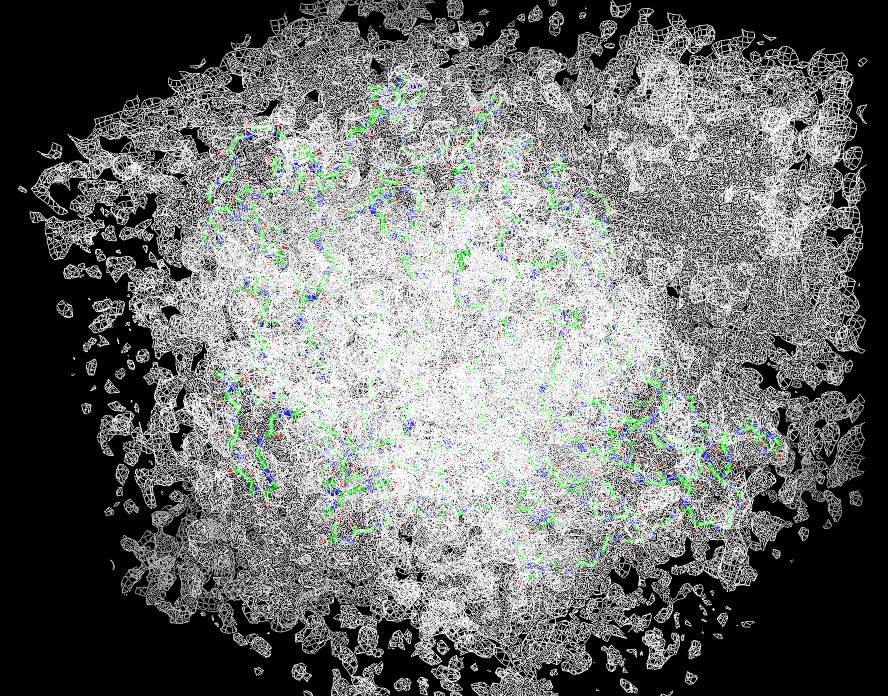
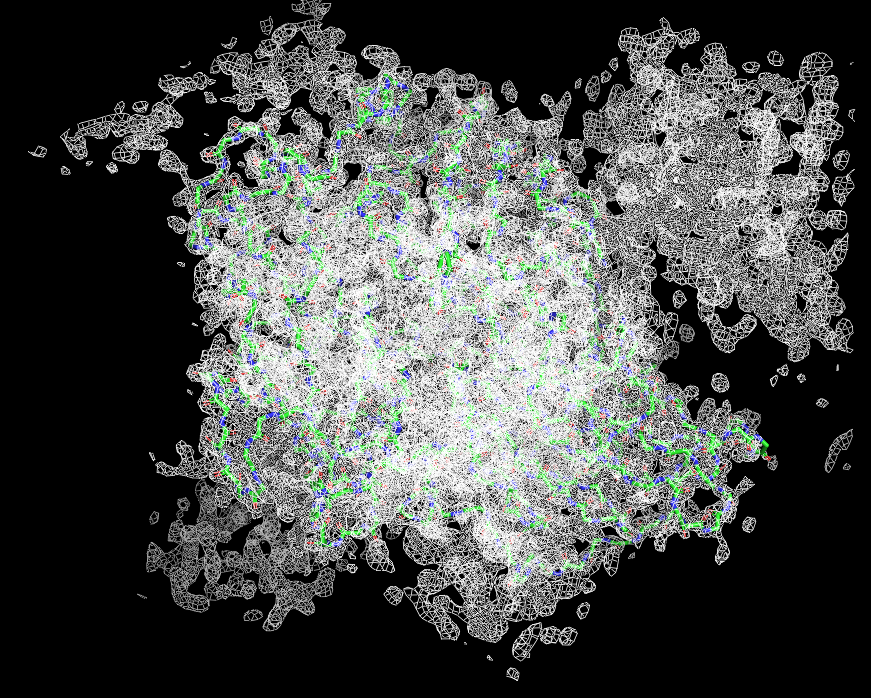
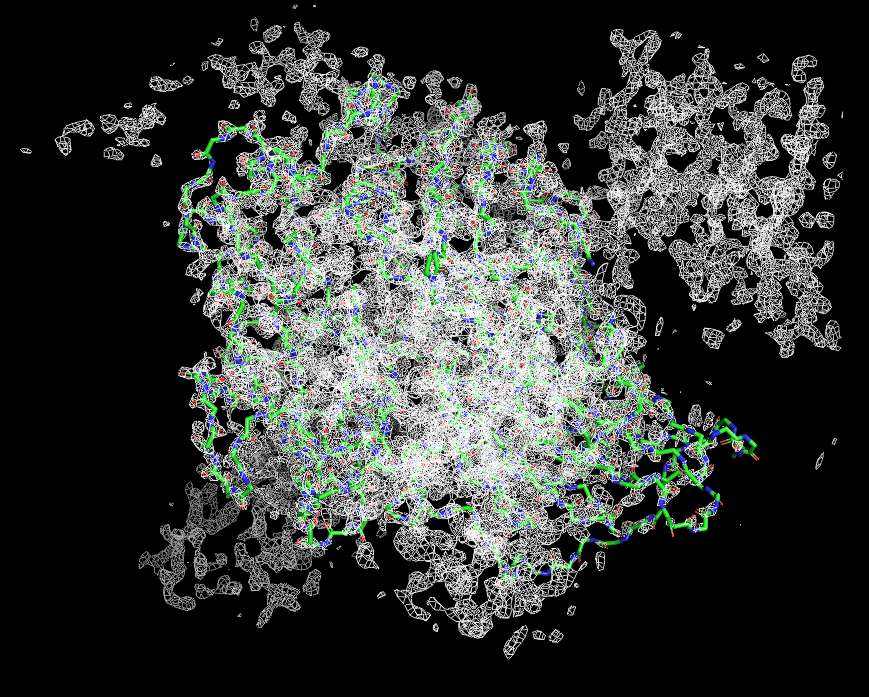
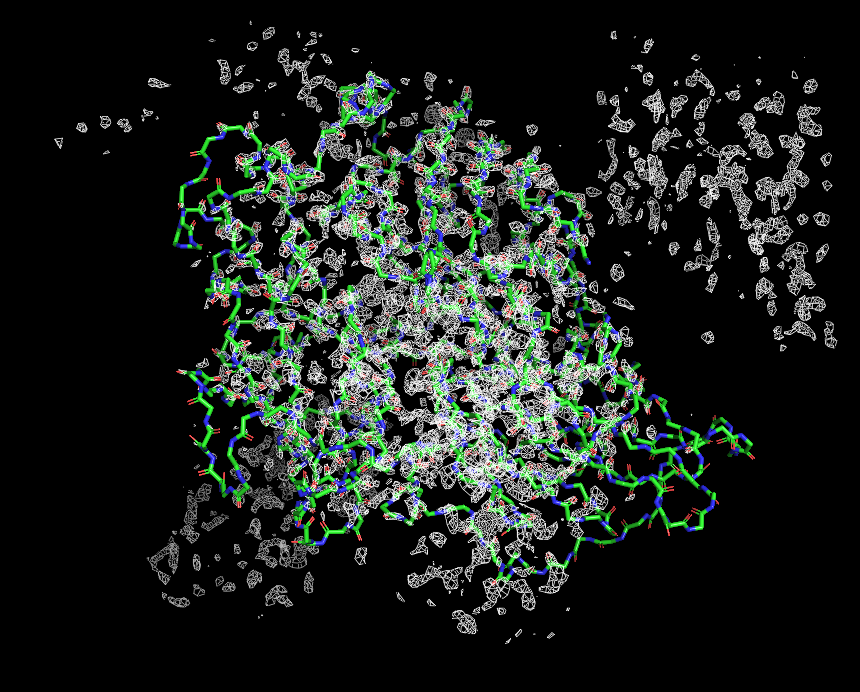
Я варьировала уровень подрезки от 0.5 до 3.5 с шагом 0.5 (см рис 3.1-3.8, в подписях указан уровень подрезки), оптимальным уровнем подрезки для визуализации ЭП всей молекулы мне показалось 1.3, тк при этом атомы попадают в центр ЭП, и ЭП для атомов в разных частях полипептидной цепи не перекрываются (см рис 4).
 |
 |
 |
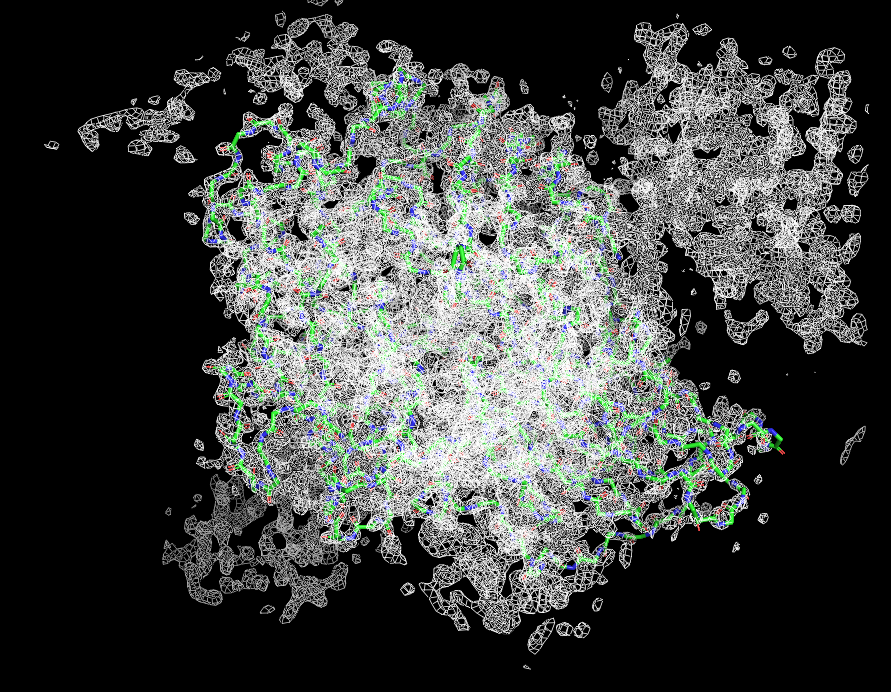 |
| Рисунок 3.1 0.5 σ. | Рисунок 3.2 1 σ. | Рисунок 3.3 1.5 σ. | Рисунок 3.4 2 σ. |
 |
 |
 |
 |
| Рисунок 3.5 2.5 σ. | Рисунок 3.6 3 σ. | Рисунок 3.7 3.5 σ. | Рисунок 3.8 100 σ. |
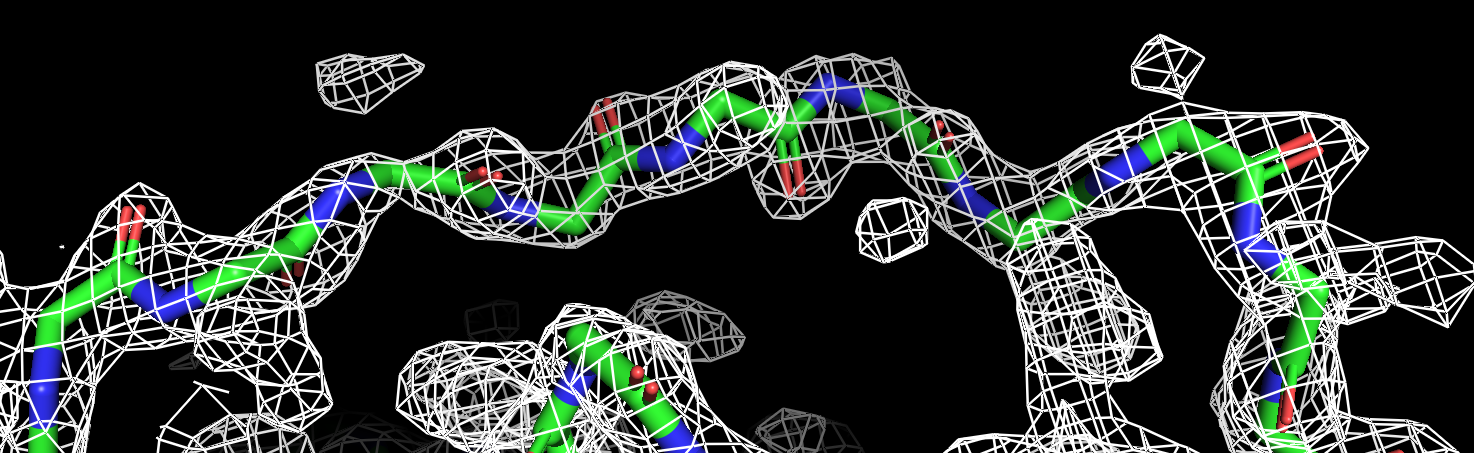
Рисунок 4. Изображение фрагмента остова полипептидной цепи, уровень подрезки 1.3 σ - оптимальный.
Визуализация ЭП вокруг различных остатков
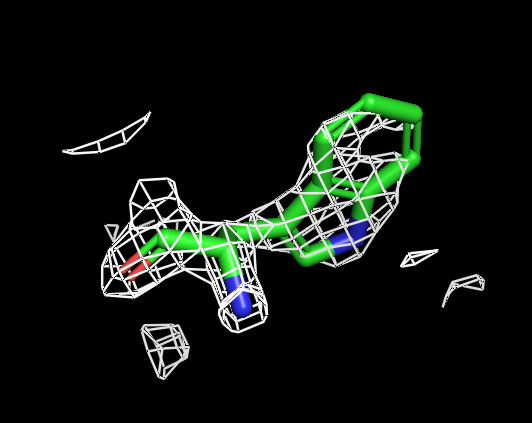
Я выбрала для работы остатки: TRP`257, ILE`2, HIS`215. Триптофан - годрофобная, ароматическая аминокислота; изолейцин - гидрофобная алифатическая; гистидин - гидрофильная, ароматическая, заряженная. Интресно, что для изолейцина (алиф.) оптимальный уровень подрезки самый высокие, и при уровне подрезки 2 практически не искажается структура, хотя это происходит для двух других ароматических аминокислот. Для меня это странно, тк мне кажется, что вокруг ароматических аминокислот как должна быть выше электронная плотность и "подрезка" этого облака до оптимальных размеров должна быть более существенной, чем для алифатических.
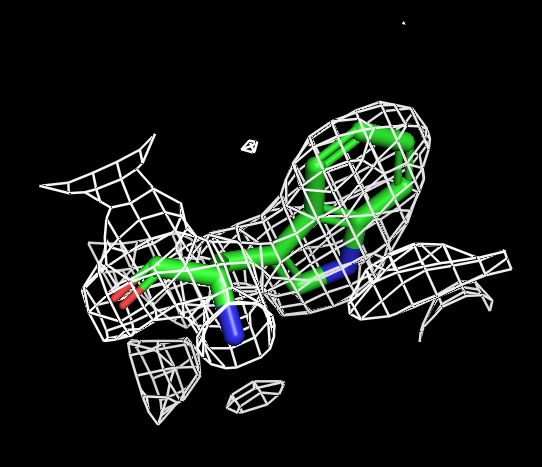
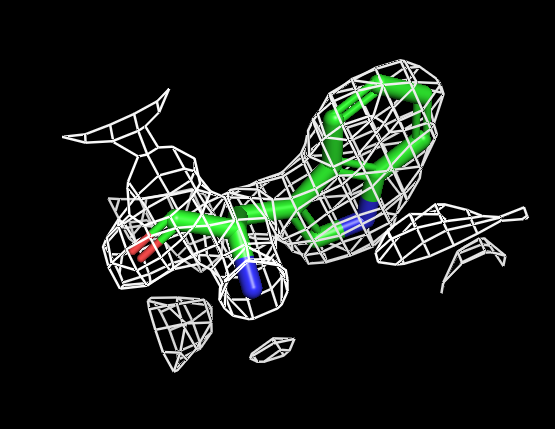
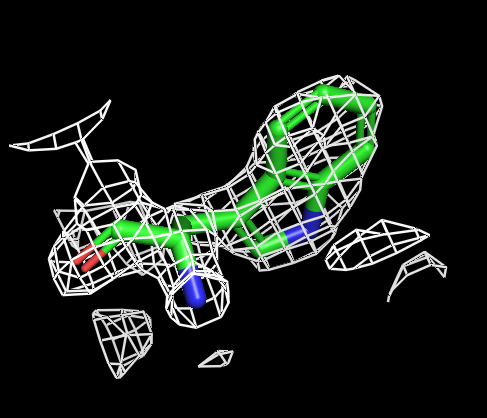
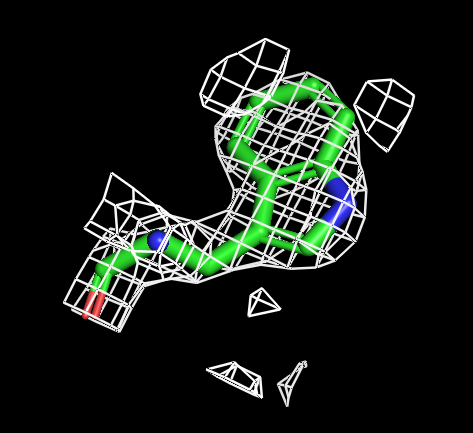
Для визуализации триптофана мне показался оптимальным буферный объем 0.5. Оптимальный уровень подрезки 1.3 или 1.5.
 |
 |
 |
 |
 |
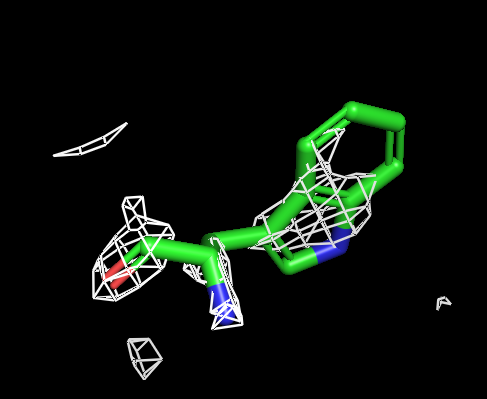 |
| Рисунок 5.1 TRP, 1 σ. | Рисунок 5.2 TRP, 1.3 σ. | Рисунок 5.3 TRP, 1.5 σ. | Рисунок 5.4 TRP, 1.6 σ. | Рисунок 5.3 TRP, 2 σ. | Рисунок 5.4 TRP, 2.5 σ. |
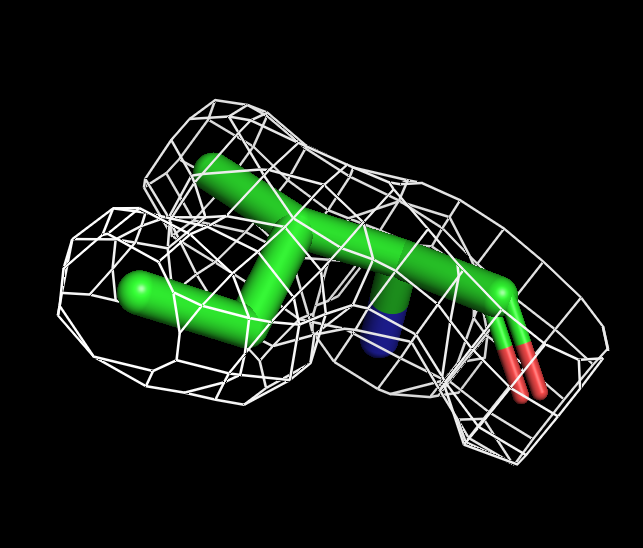
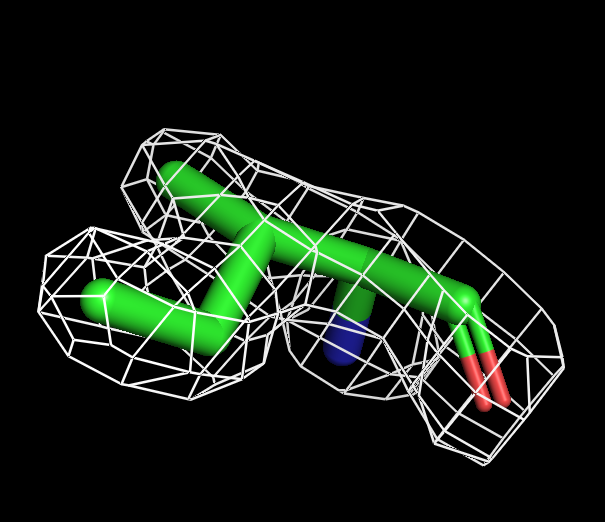
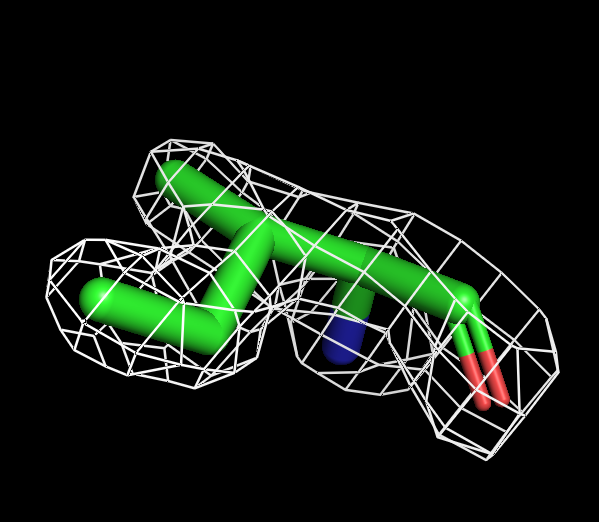
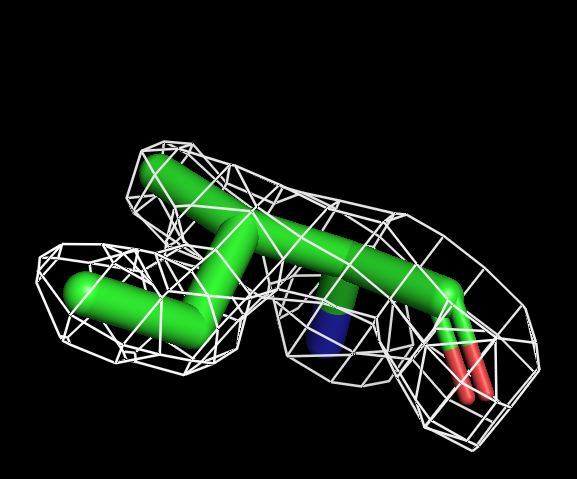
Для визуализации изолейцина буферный объем не нужен. Оптимальный уровень подрезки 1.8.
 |
 |
 |
 |
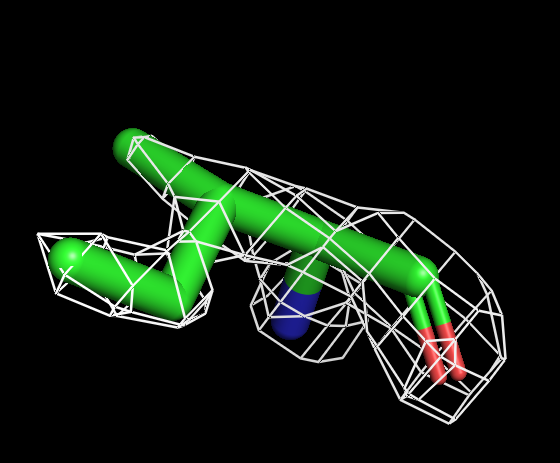 |
| Рисунок 6.1 ILE, 1 σ. | Рисунок 6.2 ILE, 1.5 σ. | Рисунок 6.3 ILE, 1.8 σ. | Рисунок 6.4 ILE, 2 σ. | Рисунок 6.3 ILE, 2.5 σ. |
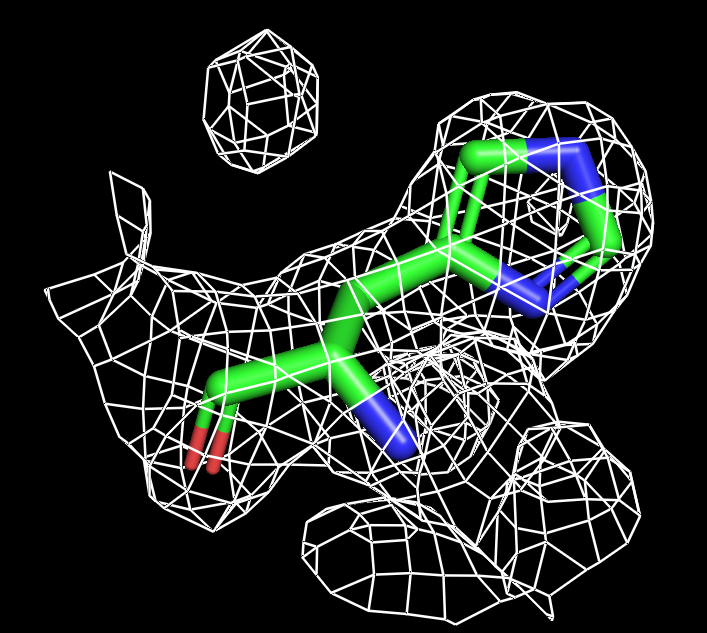
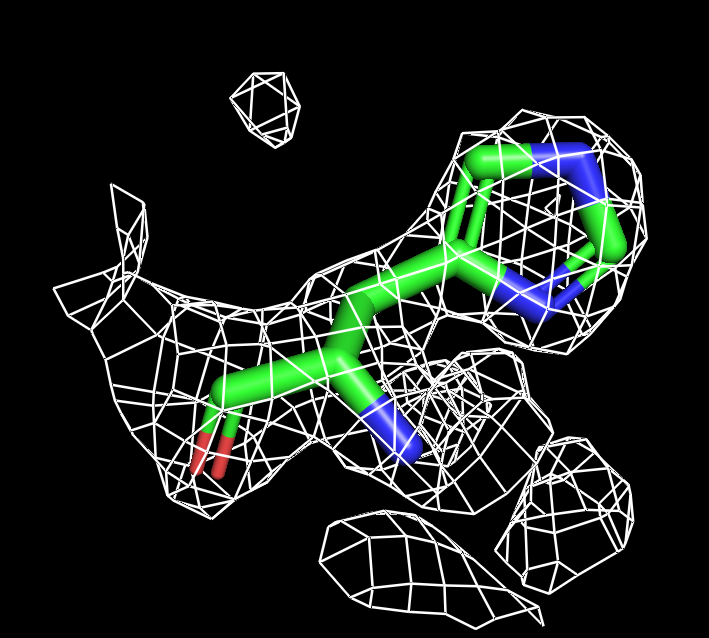
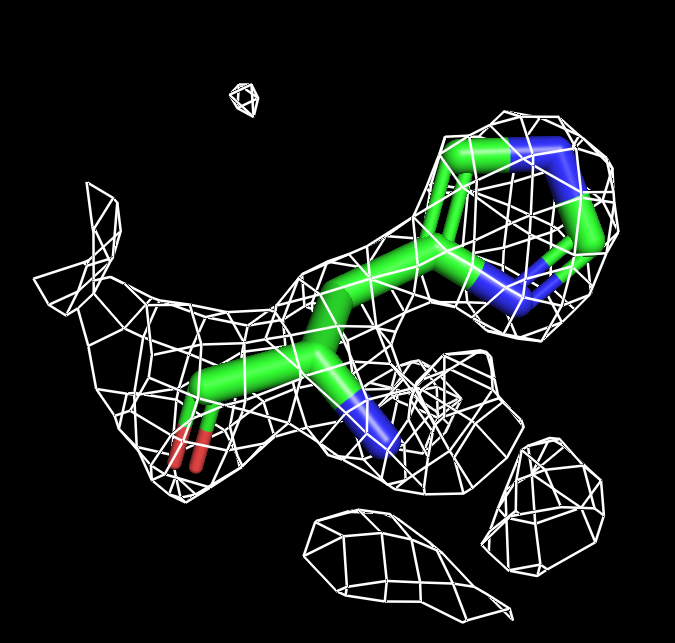
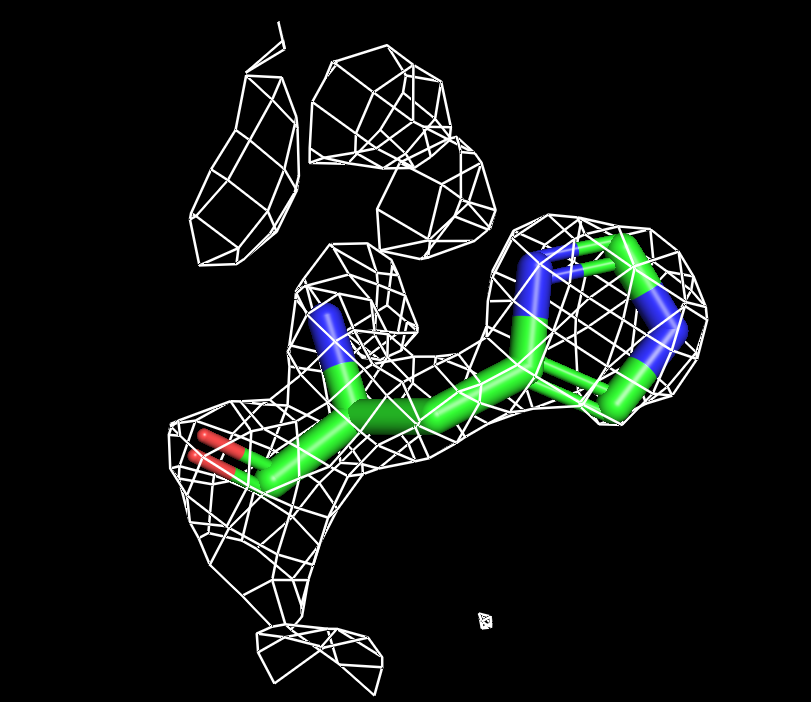
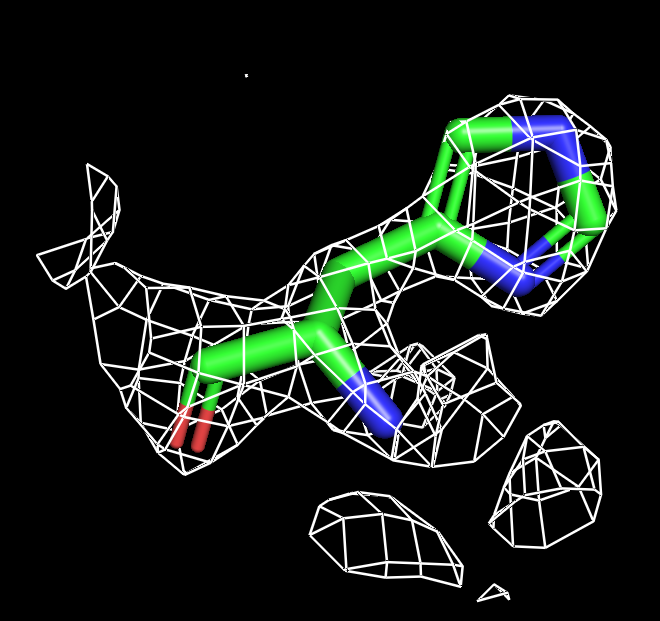
Для визуализации гистидина оптимальный буферный объем 1. Оптимальный уровень подрезки 1.5.
 |
 |
 |
 |
 |
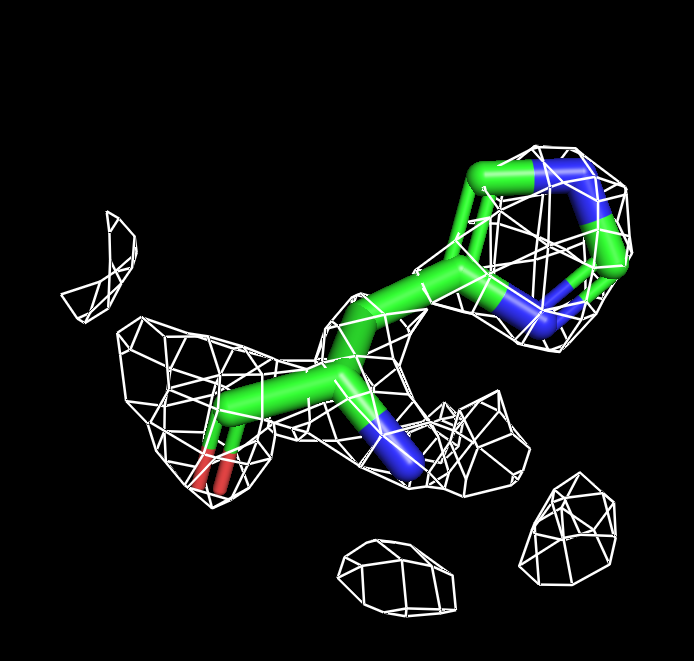 |
| Рисунок 7.1 HIS, 1 σ. | Рисунок 7.2 HIS, 1.5 σ. | Рисунок 7.3 HIS, 1.8 σ. | Рисунок 7.4 HIS, 1.9 σ. | Рисунок 7.3 HIS, 2 σ. | Рисунок 7.4 HIS, 2.3 σ. |
Список литературы
[1] L. P. Christopher, B. Yao, and Y. Ji, “Lignin biodegradaton with laccase-mediator systems,” Front. Energy Res., vol. 2, no. MAR, pp. 1–13, 2014.
[2] O. A. Glazunova, K. M. Polyakov, T. V. Fedorova, P. V. Dorovatovskii, and O. V. Koroleva, “Elucidaton of the crystal structure of Coriolopsis caperata laccase: Restoraton of the structure and actvity of the natve enzyme from the T2-depleted form by copper ions,” Acta Crystallogr. Sect. D Biol. Crystallogr., vol. 71, pp. 854–861, 2015.
| Семестр 7 |
| ← Предыдущий семестр | Практикум 2 → |
© Дарья Потанина, 2019