Главная
Практикум №11: "Предсказание генов прокариот"
Задание 1. Аннотирование последовательности и сравнение её с аннотацией генов в записи GenBank.
Для аннотации была выбрана единственная кольцевая хромосома бактерии Escherichia coli UMN026. Таксономия организма представлена на Рис.1.
GenBank: CU928163.2
Аминокислотная последовательность (GenBank) в fasta-формате.
 Рис. 1. Таксономия Escherichia coli UMN026.
Рис. 1. Таксономия Escherichia coli UMN026.
Для аннотирования последовательности данной хромосомы использовался сервер RAST. С его помощью была получена и обработана в Excel
таблица аннотаций RAST. Таблица аннотаций генов белков из NCBI, также обработанная в Excel: таблица аннотаций GenBank.
RAST аннотировал все гены, среди которых были не только нужные нам гены белков, но и гены РНК. Таких генов RAST аннотировал 109. Данные гены
не учитывались при дальнейшем выполении задания. Рассматривались только гены белков.
Всего генов белков RAST аннотировал 4997, а GenBank 4820.
Число совпадающих аннотаций (одинаково аннотированы и старт- и стоп-кодоны): 3773.
Число генов с совпадающими стоп-кодонами, но различными старт-кодонами: 4180.
Число генов, аннотированных в RAST и неаннотированных в GenBank: 402.
Число генов, аннотированных в GenBank и неаннотированных в RAST: 223.
Также в Excel была отредактирована таблица сравнения аннотаций генов в RAST и GenBank.
Проверка генов с несовпадающимим аннотациями RAST и GenBank с помощью BLAST.
Были выбраны белки - продукты трёх генов, у котрорых не совпадают старт-кодоны и совпадают стоп-кодоны:
Bifunctional riboflavin kinase/FMN adenylyltransferase.
ATP-dependent RNA helicase HrpA.
Putative zinc-dependent metallopeptidase.
Использовались аминокислотные последовательности данных белков (продуктов генов), которые загонялись в blastp. Банк: Swiss_prot;
Поиск сходных последовательностей был произведён только для штаммов E.coli: Escherichia coli (taxid:562).
Рассмотрим ген белка Bifunctional riboflavin kinase/FMN adenylyltransferase. На Рис. 2. видно, что старт-кодоны в аннотациях RAST и GenBank
не совпадают.
 Рис. 2. Координаты старт-кодонов выделены красным. Ген располагается на прямой цепи (
обозначение 1).
Рис. 2. Координаты старт-кодонов выделены красным. Ген располагается на прямой цепи (
обозначение 1).
Ген, аннотированный GenBank, длиннее. И именно аминокислотную последовательность с этого гена была задана на вход в blastp.
Была найдена лишь одна находка (Рис. 3).
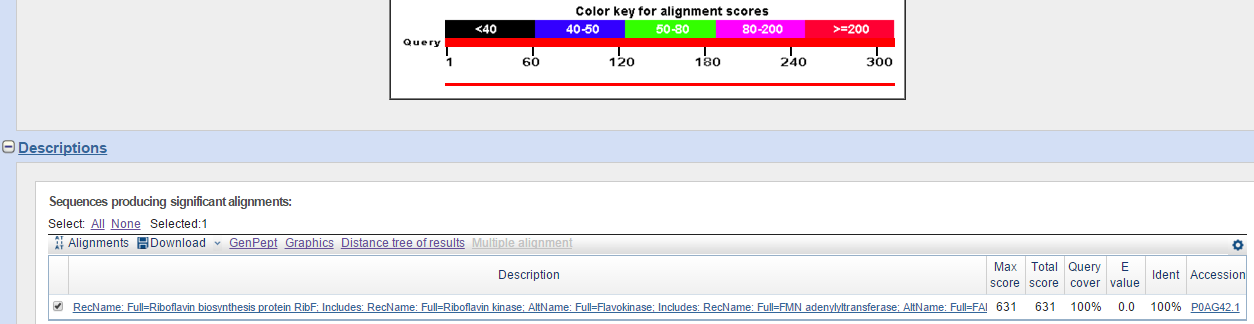 Рис. 3. Единственная находка для белка Bifunctional riboflavin kinase (аннотация из
GenBank).
Рис. 3. Единственная находка для белка Bifunctional riboflavin kinase (аннотация из
GenBank).
На Рис. 4. представлено выравнивание данных аминокислотных последовательностей, из которого видно, что последовательности полностью идентичны.
Это позволяет предположить, что аннотация GenBank данного гена более "правильная".
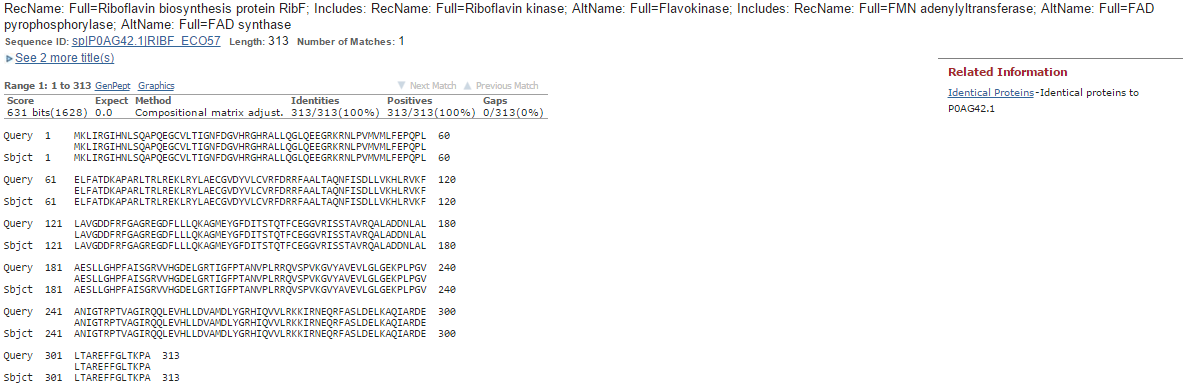 Рис. 4.
Если же проверить аналогично аннотацию RAST, то pblast
также находит одну эту же находку, выравнивание представлено на Рис.5. Но в данном выравнивании последовательность, гомологичная последовательности
нашего белка, входит не с первого остатка, как в предыдущем случае, а с 19.
Рис. 4.
Если же проверить аналогично аннотацию RAST, то pblast
также находит одну эту же находку, выравнивание представлено на Рис.5. Но в данном выравнивании последовательность, гомологичная последовательности
нашего белка, входит не с первого остатка, как в предыдущем случае, а с 19.
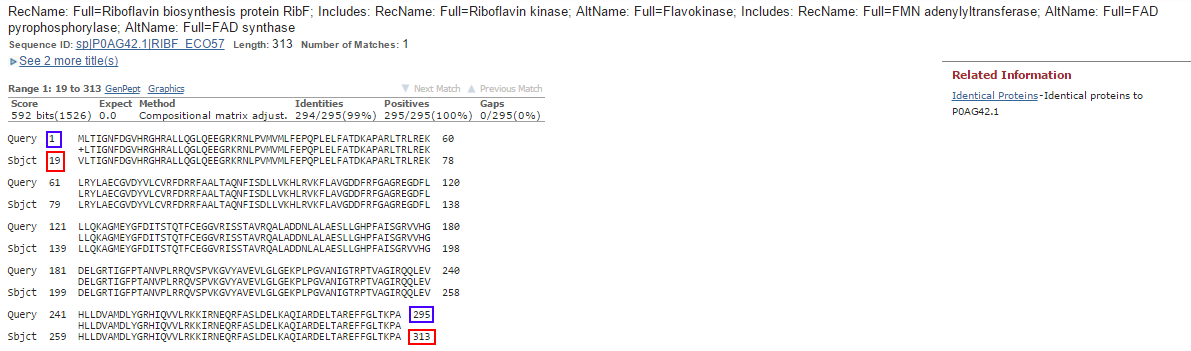 Рис. 5.
Рис. 5.
Всё это позволяет предположить, что аннотация GenBank данного гена более "правильная". Однако утверждать об этом с увереноостью нельзя, так как:
Во-первых, данная сходная последовательность получена по гомологии (экспериментально не подтверждена).
Во-вторых, она принадлежит Escherichia coli O157:H7, то есть другому штамму E.coli. То есть мы можем лишь говорить, что такой белок с идентичной последоватеьностью
может существовать в близкородственном штамме.
Рассмотрим ген белка ATP-dependent RNA helicase HrpA. На Рис. 6. видно, что старт-кодоны в аннотациях RAST и GenBank
не совпадают.
 Рис. 6. Координаты старт-кодонов выделены красным. Ген располагается на прямой цепи (
обозначение 1).
Рис. 6. Координаты старт-кодонов выделены красным. Ген располагается на прямой цепи (
обозначение 1).
В данном случае, наоборот, ген, аннотированный RAST, длиннее. Аминокислотная последовательность белка с данного гена была подана на вход в blastp.
Было найдено 13 находок, из которых только одна хорошая (Рис. 7).
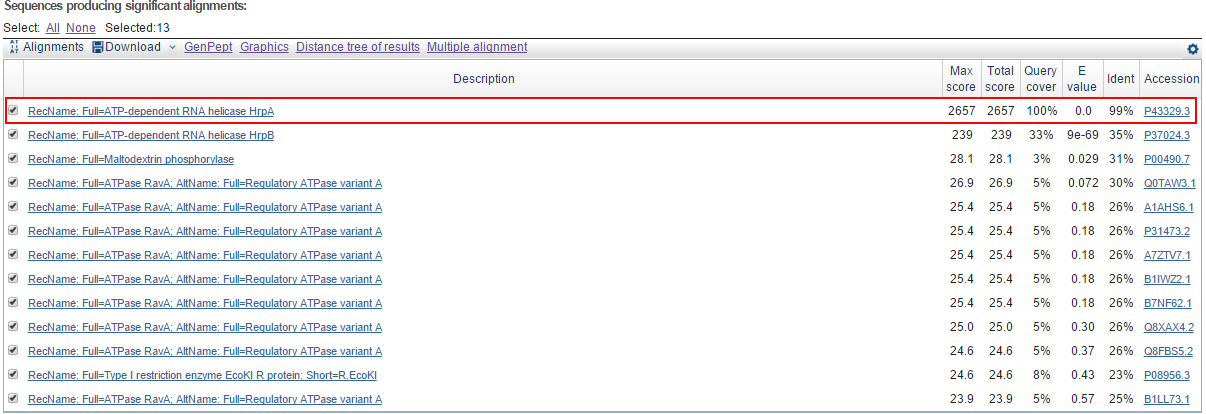 Рис. 7. Хорошая находка выделены красным цветом.
Рис. 7. Хорошая находка выделены красным цветом.
На Рис. 8a и Рис. 8b представлено выравнивание, соответствующее данной находке. Видно, что и входная последовательность нашего белка, и найденная
сходная последовательность входят в выравнивание полностью с 1 по 1300 остатки. E-value данного выравнивания 0.0, 99% идентичных остатков - всё это указывает
на то, что данные последовательности очень похожи.
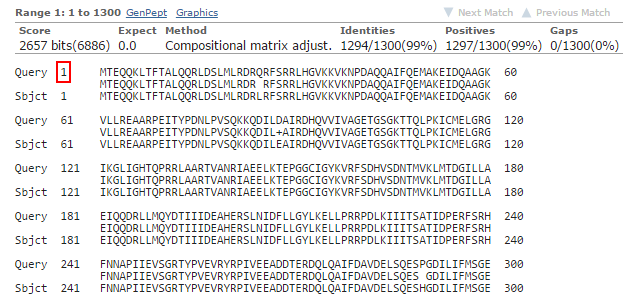 Рис. 8a. Фрагмент начала выравнивания.
Рис. 8a. Фрагмент начала выравнивания.
 Рис. 8b. Фрагмент конца выравнивания.
Рис. 8b. Фрагмент конца выравнивания.
Таким образом, в данном случае можно предположить, что аннотация RAST скорее всего верна.
Наконец, существуют гены по-разному аннотированные в RAST и GenBank, проверка продуктов которых через blastp так же ничего не проясняет.
В качестве примера рассмотрим ген белка Putative zinc-dependent metallopeptidase. На Рис. 9. видно, что старт-кодоны в аннотациях RAST и GenBank
для данного гена тоже не совпадают.
 Рис. 9. Координаты старт-кодонов выделены красным. Ген располагается на прямой цепи (
обозначение 1).
Рис. 9. Координаты старт-кодонов выделены красным. Ген располагается на прямой цепи (
обозначение 1).
По последовательности белка, соответствующему гену аннотированному в RAST, plastp не нашёл ни одной значительной находки среди белков E.coli (Рис. 10a).
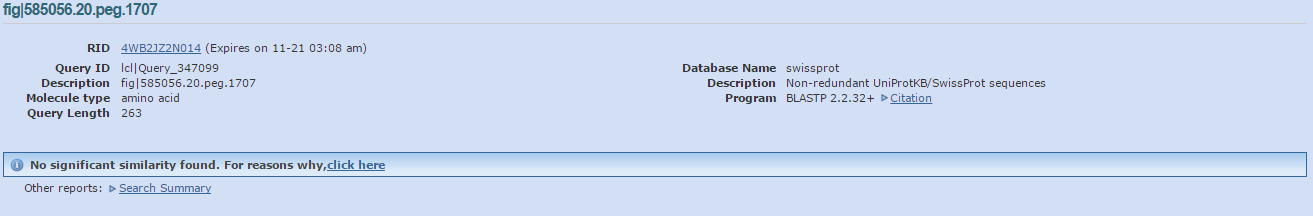 Рис. 10a.
Рис. 10a.
По последовательности белка, соответствующему гену аннотированному в GenBank, plastp нашёл во всех штаммах E.coli 6 последовательностей, однако все они очень плохие (Рис.10b).
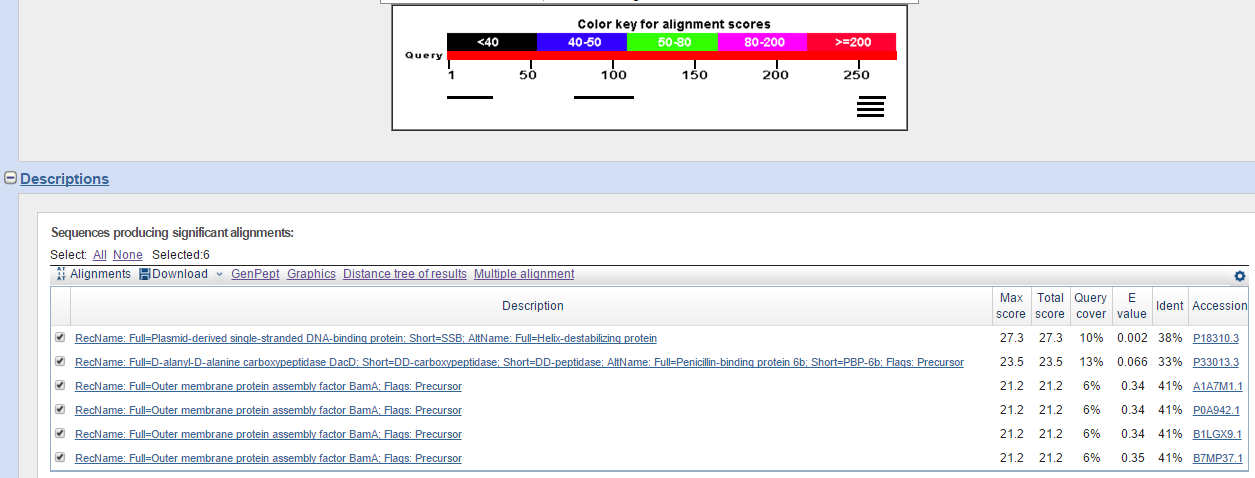 Рис. 10b. Все находки довольно случайные: query cover не более 13%,
Рис. 10b. Все находки довольно случайные: query cover не более 13%,
Большие значения E-value, % идентичности не достигает даже 50%.
Следовательно, выявить, какая аннотация для гена белка Putative zinc-dependent metallopeptidase предпочтительнее с помощью blastp затруднительно.
© Павел Волик
Факультет биоинженерии и биоинформатики, МГУ











