Главная
Практикум №10: "Поиск сигналов"
Задание 1. Сравнить состав систем рестрикции-модификации, закодированных в двух штаммах одного вида
В данном задании использовалась бактерия Alisipes shahii. Геномы были взяты из БД NCBI: FP902903.1,
и из данных о секвенировании метагенома кишечника человека. Геном, лежащий в NCBI, принадлежит немецкой коллекции микроорганизмов
и клеточных культур ("German Collection of Microorganisms and Cell Cultures").
Ссылки: полный геном, набор контигов.
Далее с помощью веб-сервиса, использующего метод Карлина, для генома было получено 25 сайтов с контрастом менее 0.78. Ссылка на файл с результатами:
все результаты, отобранные сайты.
При аналогичный анализ метагенома получили 20 сайтов с контрастом менее 0.78:
все результаты, отобранные сайты.
Полученные результаты для генома и метагенома были сравнены между собой: сравнение.
Число избегаемых сайтов, встретившихся только в полном геноме (в файле Excel выделены красным цветом), равняется 10, а сайтов, встретившихся лишь в
метагеноме, 5. Сайтов, которые есть и там, и там, 15.
Мы получили, что в метагеноме меньше сайтов, чем в полном геноме. Однако
судить об этом мы можем только косвенно, по недопредставленности некоторых сайтов и вполне можем ошибаться.
Задание 2. Поиск последовательности Шайна – Дальгарно
Последовательность Шайна — Дальгарно (англ. Shine-Dalgarno sequence, Shine-Dalgarno box) — сайт связывания рибосом на молекуле мРНК прокариот,
обычно на расстоянии около 10 нуклеотидов до стартового кодона AUG.[1]
Описана австралийскими учёными Джоном Шайном и Линн Дальгарно.[ 2]
Консенсусом является последовательность из шести нуклеотидов AGGAGG; в случае E. coli последовательность Шайна — Дальгарно — AGGAGGU.
Комплементарная последовательность CCUCCU, называемая последовательностью анти-Шайна — Дальгарно, располагается на 3'-конце молекулы 16S рибосомной РНК. Комплементарное взаимодействие межд
у последовательностями Шайна — Дальгарно и анти-Шайна — Дальгарно служит для помещения старт-кодона мРНК в P-сайт рибосомы для начала биосинтеза
белка.[3]
В данном задании необходимо было найти последовательности Шайна - Дельгарно в геноме бактерии из первого семестра - Caldilinea aerophila,
геном которой был скачан из БД NCBI.
Далее были выбраны "хорошие гены" - достаточно длинные (выбирались длиной более 600 п.н.), с адекватной аннотацией
(с определённой точностью известен продукт белка). В результате было выбрано 679 таких генов. Ссылка на файл Excel
Затем для каждого гена был вырезан участок перед СDS от -17 до -1. В этих вырезанных фагментах искали мотивы c помощью программы MEME:
Параметры: длина мотива - от 6 до 8, 0 или 1 мотив на одну последовательность, мотив искали на той же цепи. Для начала нашли 3 лучших мотива (Рис.1).
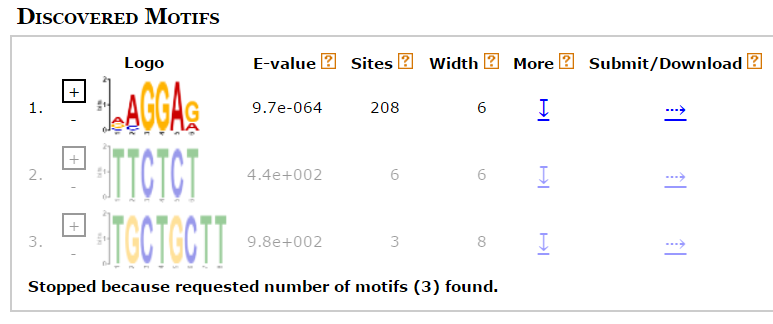 Рис. 1.
Рис. 1.
Видно, что лучший мотив (E-value: 9.7e-064) на много доставернее остальных и является SD (хотя и отличается от консенсуса AGGAGG).
Затем повторили поиск, но искали уже один лучший мотив. Получили позиционную матрицу весов PWM (Рис. 2).
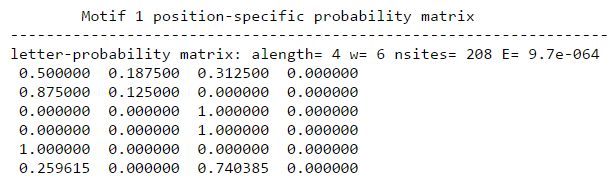 Рис. 2.
Рис. 2.
LOGO данного мотива представлено на Рис. 3.
 Рис. 3.
Рис. 3.
Далее использовалаь программа FIMO для поиска мотива для всех генов бактерии. Для этого вырезались участки от -26 до -1 до начала CDS.
Длина участков увеличена, чтобы снизить вероятность ошибок поиска для неправильно аннотированных генов.
В итоге для 4119 генов (всего столько) нашлось 13498 мотивов (p-value < 0.1), из которых 7859 имело p-value < 0.05, 5685 - p-value < 0.03,
2889 - p=value < 0.01, 789 - p-value < 0.001. Последовательность SD должна присутствовать в большинстве генов, поэтому для анализа была вязта граница
p-value < 0.01 (2889 мотивов найдено для 4119 генов). Результаты работы FIMO и
анализ полученных данных представлен в файле
FIMO.
Была построена гистограмма распределения начала SD от старта трансляции (Рис. 4).
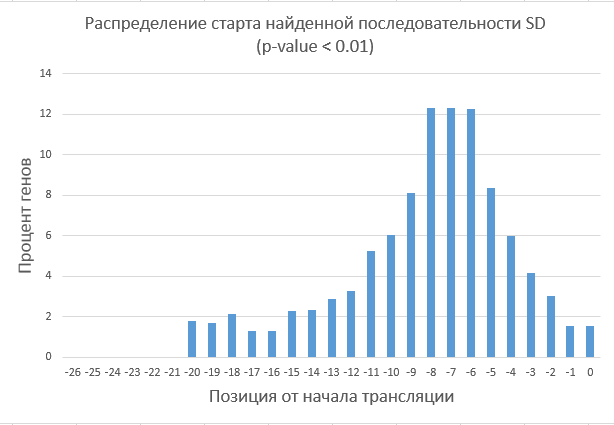 Рис. 4. Гистограмма распределения начала SD от старта трансляции
(p-value < 0.01).
Рис. 4. Гистограмма распределения начала SD от старта трансляции
(p-value < 0.01).
Видно, что у данных находок в основном старт SD приходится на позиции от -8 до -6 от начала трансляции, что
согласуется с литературными данными, по которым в основном SD должны начинаться в районе от -10 до -5 от начала трансляции
[4].
Далее для данных мотивов было построено logo (Рис. 5 для p < 0.01).
 Рис. 5.
Рис. 5.




