Проект JalView, содержащий все выравнивания из этого практикума: PR13-align-pfam.jar
Множественные выравнивания
С помощью BLAST были найдены гомологи белка CPAF в базе RefSeq, результаты были отфильтрованы по Identity = 40-80%, E-value < 0.00001. Из полученных белков были отобраны 8 из разных видов с Query cover > 90%.
1. Последовательности этих белков были использованы для множественного выравнивания программой MUSCLE. Выравнивание строилось на сервере kodomo командой
muscle -in sample.fasta -out align13.fasta
- Результат в формате fasta: muscle.fasta
2. Последовательности были выравнены веб-сервисом T-coffee. Эта программа использует метод расширенной библиотеки и работает примерно в N раз медленнее ClustalW (N - число последовательностей).
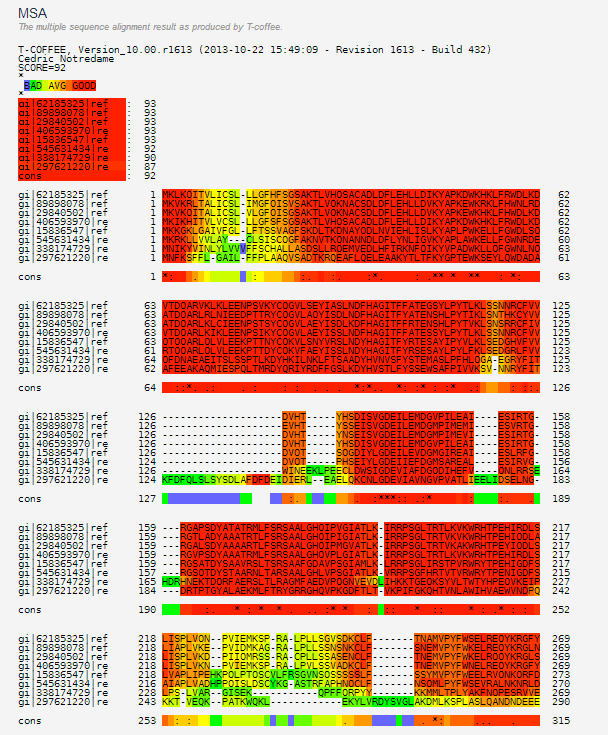
Рис.1. Пример выравнивания сервисом T-coffee. Красным показаны наиболее надежно выравненные позиции, зеленым и синим - менее надежные.
- Результат в формате fasta: tcoffee.fasta
3. Выравнивания, полученные Muscle и T-coffee, сравнивались посредством опции Muscle для выравнивания двух выравниваний. Области с примерами сходства и различия приведены на рисунках 2 и 3.
- Полученное выравнивание выравниваний можно скачать здесь: compare.fasta
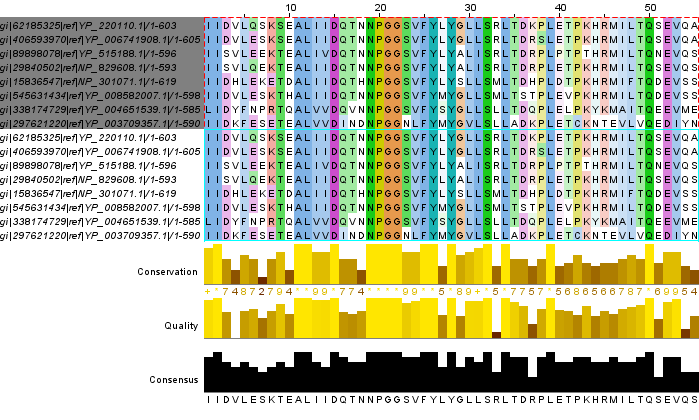
Рис.2. Область совпадения двух выравниваний. Верхняя группа - выравнивание Muscle, нижняя - T-coffee. Область занимает позиции 404-458 в общем выравнивании. Раскраска ClustalX с консервативностью 40%.
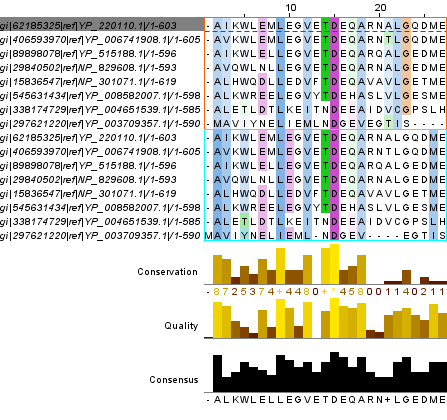
Рис.3. Область расхождения двух выравниваний. Область занимает позиции 459-486.
В целом можно отметить, что расхождения в выравниваниях происходят в основном за счет разной расстановки гэпов в двух нижних последовательностях, у которых были меньше Query cover и Identity. Это происходит лишь на коротких участках, за исключением окончаний белков, которые имеют разную длину и неконсервативны. В остальном выравнивания достаточно похожи, видимо, потому что белки в выборке высоко гомологичны (высокий процент Query cover) - синтезирующие их бактерии принадлежат одному роду.
Pfam
База Pfam (Protein families) позволяет осуществлять поиск по доменам белков. Я провела поиск по последовательности моего белка CPAF. Нашелся один домен - Peptidase_S41. Белки этого семейства периодически появлялись в результатах поиска в предыдущих заданиях. На сайте Pfam не приведено информации о белках этого семейства, но она есть на сайте MEROPS. Семейство объединяет некоторые сериновые эндопептидазы. Оно делится на два подсемейства: первое содержит протеазы, узнающие С-концы белков; его активный сайт содержит серин и лизин. Второе содержит в активном сайте тетраду Ser745, His746, Ser965, Glu1023 и предположительно расщепляет пептиды, оставшиеся после работы протеасомы.
Pfam известны 9366 последовательностей, содержащих домен Peptidase_S41. Я скачала seed-alignment - выравнивание небольшой выборки белков семейства.
- Seed-выравнивание последовательностей из семейства Peptidase_S41: Peptidase_S41_seed.fa