1. Поиск гомологов белка Cpaf из бактерии Chlamydia trachomatis в банке UniProt/SwissProt
Был проведен поиск гомологов последовательности белка Chlamydial protease-like activity factor бактерии Chlamydia trachomatis. Поиск производился с помощью программы protein blast на сайте NCBI.
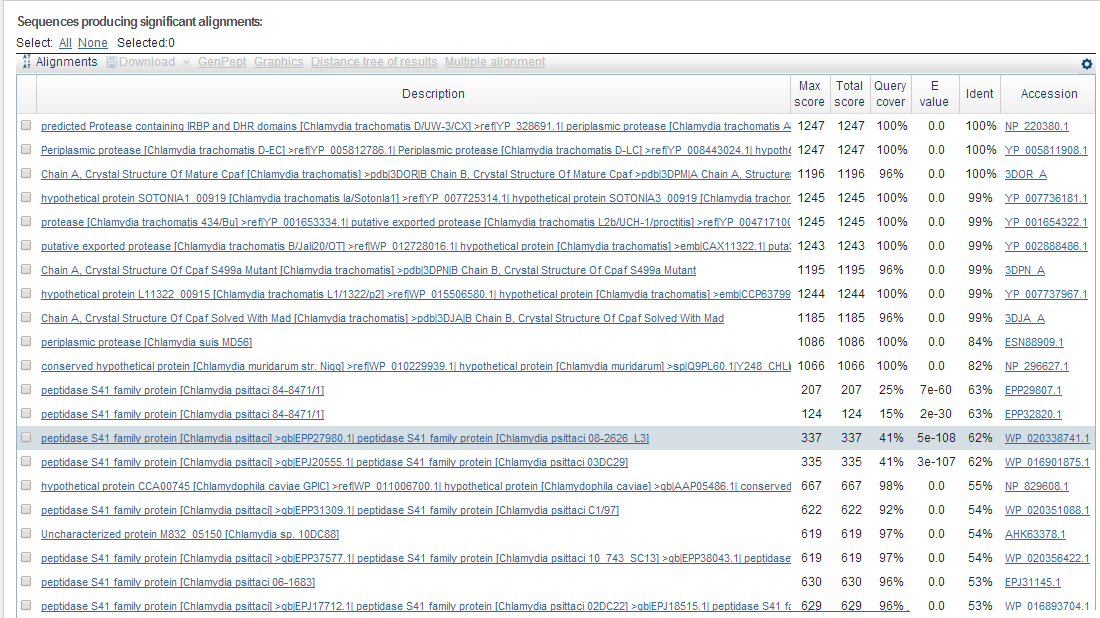
Рис.1. Таблица с найденными гомологами и их параметрами.
Из находок был выбран белок chlamydial protease-like activity factor из бактерии Candidatus Protochlamydia amoebophila. Параметры находки приведены в таблице 1.
| Description | Max score | Total score | Query cover | E value | Ident | Accession |
| chlamydial protease-like activity factor [Candidatus Protochlamydia amoebophila] | 275 | 275 | 93% | 2x10-80 | 32% | BAM93530.1 |
- Description - имя и описание белка
- Max score - максимальный вес выравнивания найденной последовательности относительно данной
- Total score - суммарный вес выравниваний гомологичных участков данной и найденной последовательностей
- Query cover - сколько процентов от найденной последовательности занято гомологичными данной участками
- E value - число различных выравниваний с весом выравнивания не меньше данного, которые найдутся в базе данных случайным образом.
- Ident - процент идентичных аминокислот.
- Accession - код доступа белка в базе данных.
На том же ресурсе было сделано выравнивание данной последовательности с выбранной.
Query 24 ESLVCKNALQDLSFLEHLLQVKYAPKTWKEQYLGWDLVQSSVSAQQKLRTQENPSTSFCQ 83
E + + ++DL ++H+ V YAP WK+ Y G DL + + + + + Q
Sbjct 28 EDIKKGSMIEDLKMIKHVFDVGYAPLEWKKNYTGLDLDEELNKSIDLILSNPTLTHKDFQ 87
Query 84 QVLADFIGGLNDFHAGVTFFAIESAYLPYTVQKSSDGRFY--FVDIMTF---SSEIRVGD 138
+++ +F+ D+H V FF+ E+A LP+ V K + R++ ++D + + I+VGD
Sbjct 88 RIVKNFLASTKDYHVDVIFFSTETATLPFDV-KGVNNRYFITWIDELKLPPSTYTIKVGD 146
Query 139 ELLEVDGAPVQDVLATL--YGSNHKGTAAEESAALRTLFSRMASLGHKVPSGRTTLKIRR 196
E++E DG P+ DV+ L GS + +++ A L +R+ LG +P G T+K+
Sbjct 147 EIIEFDGRPLGDVIEELKQQGSKNSNPLTDQALAEIKLTNRLGWLGDIIPQGPVTIKVH- 205
Query 197 PFGTTREV----RVKWRYVPEGVGDLATIAPSIRAPQLQKSMRSFFPKKDDAFHRSSSLF 252
++EV ++ W Y PE + +PS +++ SFFP K +
Sbjct 206 --SRSKEVPLSYQLIWDYRPELI-----FSPSF----FLQTVESFFPNKKISVR------ 248
Query 253 YSPMVPHFWAELRNHYATSGLKSGYNIGSTDGFLPVIGPVIW-------ESEGLFRAYIS 305
SP + L + S K + + F+P +GP +W E E + AYI
Sbjct 249 -SPCMTMM--NLTHRKMMSETKRDGTLCARKSFIPTLGPRVWMFDKIDKEKEISWYAYIY 305
Query 306 SVTDGDGKSHKVGFLRIPTYSWQDMEDFDPSGPPPWEEFAKIIQVFSSNTEALIIDQTNN 365
+++ K+G++RIP Y E +EF +++ T+AL+IDQ +N
Sbjct 306 KISE----EEKIGYIRIPHYIGYKEES---------KEFGELLNYLEQKTDALVIDQVHN 352
Query 366 PGGSVLYLYALLSMLTDRPLELPKHRMILTQDEVVDALDWLTLLENVDTNVESRLALGDN 425
GG + Y L SML PL+ PKH+M +TQ +V+ A L ++E + ++ D
Sbjct 353 GGGYASFQYELASMLALNPLKTPKHQMKITQKDVLTAYQILDVIEKIQQGID------DE 406
Query 426 MEGYTVDLQVAEYLKSFGRQVLNCWSKGDIELSTPIPLFGFEKIHPHPRVQYSKPICVLI 485
+E +D Q +LK+F + W G L+ P L G + I+PH +Y+KPI +LI
Sbjct 407 VEEGPIDYQHLLFLKAFYEFTIQEWD-GQRTLTDPTHLEGCDWINPHSDYRYTKPILMLI 465
Query 486 NEQDFSCADFFPVVLKDNDRALIVGTRTAGAGGFVFNVQFPNRTGIKTCSLTGSLAVREH 545
NE DFS DF P +++DN RA++ GTRT+GAGGFV FPN GI S TGS+A R
Sbjct 466 NELDFSGGDFMPAIMQDNQRAVLFGTRTSGAGGFVLQASFPNNNGIAAFSYTGSIAERPE 525
Query 546 GAF-IENIGVEPHIDLPFTANDIRYKGYSEYLDKVKKLVCQLI 587
IEN+GV P I T +D++ GY Y V + + LI
Sbjct 526 TLLKIENLGVTPDIVYSLTVDDLQ-NGYQGYKAAVNEAIQALI 567
Выровнялись участки с 24 по 587 аминокислотных остатка у данной последовательности и с 28 по 567 у выбранной. Идентичных данной аминокислот в выбранной последовательности 185/583 (32%), схожих аминокислот - 294/583 (50%). Всего с данной последовательностью выровнялось 97% найденной (563 остатка).
2. Карта локального сходства двух последовательностей
С помощью BLAST (опция Align two sequences) была построена карта локального сходства данной и выбранной последовательностей. Она приведена на рисунке 2.
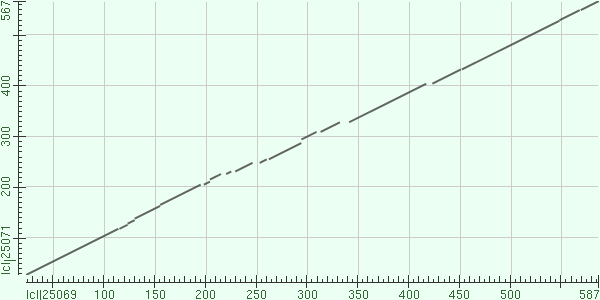
Рис.2. Карта локального сходства последовательностей белка CPAF Chlamydia trachomatis (по горизонтали) и Protochlamydia amoebophila (по вертикали). Сплошной линией отмечены соотнесенные участки. Разрывы - гэпы в первой или во второй последовательности.
Судя по карте, на всем выровненном участке последовательности прослеживается гомология, за исключением коротких гэпов. Это правдоподобно, поскольку белки выполняют одну и ту же функцию, а бактерии находятся в одном классе.
Гомологи в других таксонах
Результаты были отфильтрованы по таксону с помощью опции Organism из вкладки Formatting options на странице результатов. Сначала я посмотрела наличие гомологов в эукариотах, но их не было. Я выбрала тип Proteobacteria. Среди них оказалось 23 последовательности, из них все протеазы/пептидазы или их функция неизвестна.

Рис.3. Гомологи белка CPAF среди представителей типа Proteobacteria.
Хотя Ident лежит в пределах 31-39%, что схоже с белком из Protochlamydia amoebophila, параметр Query cover гораздо ниже - видимо, виды достаточно удалены от хламидии, и гомология прослеживается только в определенных консервативных областях белка.
Из находок было выбрано 9 последовательностей и произведено множественное выравнивание (опция Multiple alignment в разделе Descriptions). Его можно скачать здесь.
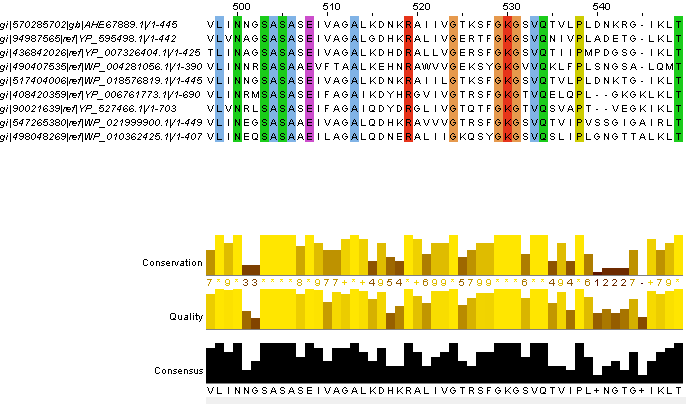
Рис.4. Участок множественного выравнивания гомологов CPAF среди Proteobacteria в JalView. Раскраска ClustalX с консервативностью 100%.
В выравнивании заметны участки (ближе к концу последовательностей) с большим числом совпадающих аминокислот. Видимо, в эти участки входит активный центр белков, или они играют важную роль в структуре.