На этой неделе мы работали с различными формами нуклеиновых кислот, используя пакет программ 3DNA и Jmol.
Задание 1.
С помощью программы fiber пакета 3DNA, я построила 3 формы ДНК - A, B и Z. В A и B формах использовалась 5 раз повторенная последовательность "GCAT", для Z-формы - только GT (10 повторов). Файлы с результатами: gcat-a.pdb gcat-b.pdb gcat-z.pdb
 Рис.1. A-форма ДНК. Вид с торца |
 Рис.2. B-форма ДНК |
 Рис.3. Z-форма ДНК |
Задание 2.
Упр.1
С помощью Jmol были получены изображения А-формы ДНК:
-
Сахарофосфатный остов выделен фиолетовым (фосфаты) и зеленым (сахара) цветом, основания покрашены каждый в свой цвет. Скрипт для такого изображения:
cartoons off select all wireframe cpk 40 define phs (*.O1P or *.O2P or *.P or *.O5' or *.O3') select phs color purple define sugar (*.C?' or *.O4') color limegreen select [T] and not (sugar or phs) color gold select [A] and not (sugar or phs) color darkred select [G] and not (sugar or phs) color cyan select [C] and not (sugar or phs) color redorange
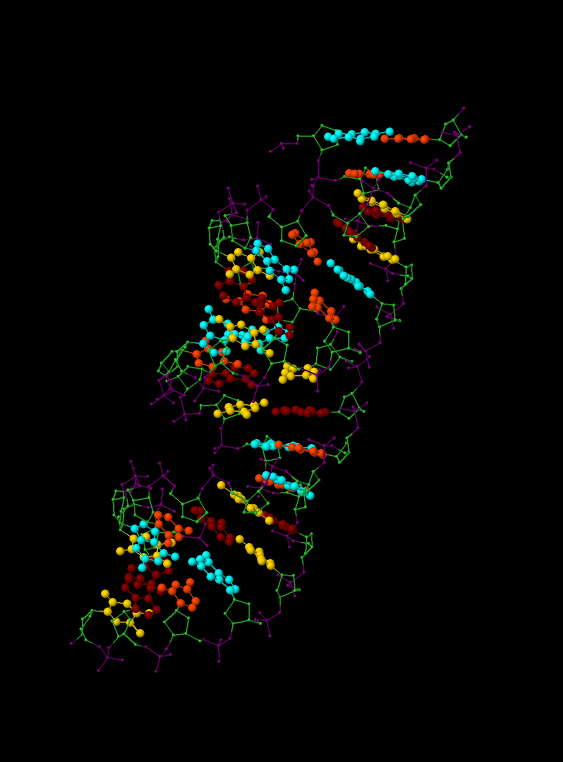
Рис.4. А-ДНК. Фосфаты выделены фиолетовым, сахара зеленым, тимин желтым, аденин красным, цитозин оранжевым, и гуанин голубым.
- Все нуклеотиды выделены - это значит, что выделена вся ДНК, т.к. она и состоит из нуклеотидов.
- Выделены все аденины

Рис.5. А-ДНК. Все аденины выделены оранжевым, остальные основания - белым.
- Выделен атом N7 во всех гуанинах
- Выделен атом N7 в первом гуанине последовательности.
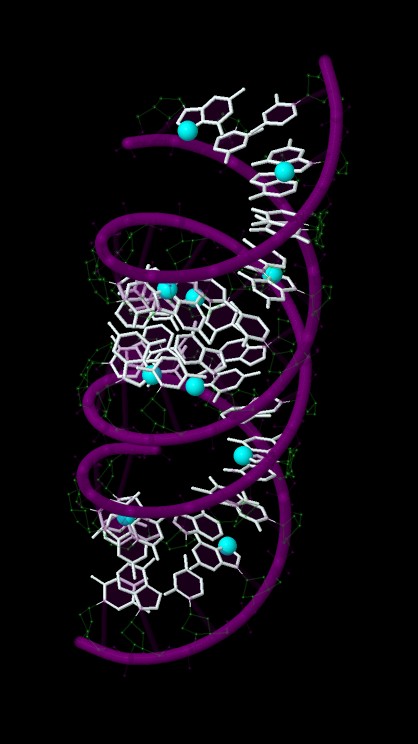
Рис.6. А-ДНК. N7 атомы в гуанинах показаны в cpk и выделены голубым.
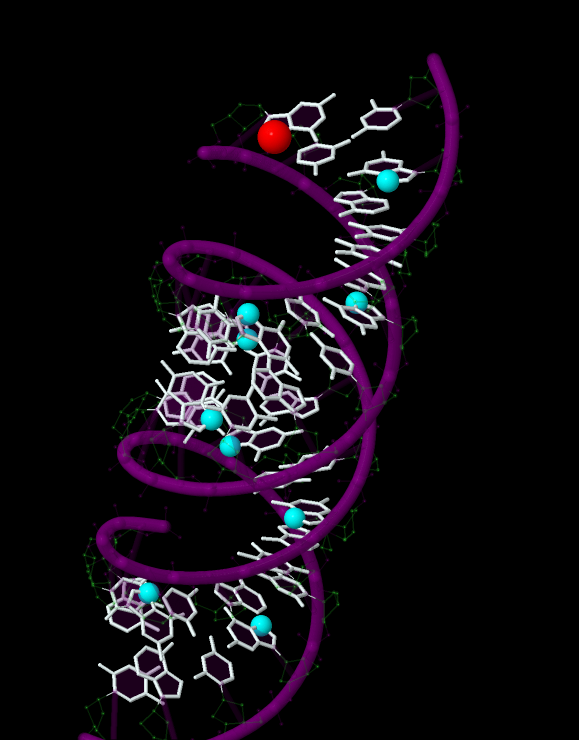
Рис.7. А-ДНК. N7 атомы в первом гуанине показан в cpk и выделены красным, остальные N7 в гуанинах - голубым.
Упр.2
Каждый получал свои структуры ДНК-белкового комплекса и тРНК и скачивал их pdb-файлы с сайта PDB. PDB-коды моих структур - 1h3e для тРНК и 1pp7 для ДНК.
Упр.3
Надо было проверить обе структуры на разрывы цепей. У меня было 2 разрыва в тРНК. Файлы, содержащие только тРНК и ДНК: тРНК, ДНК
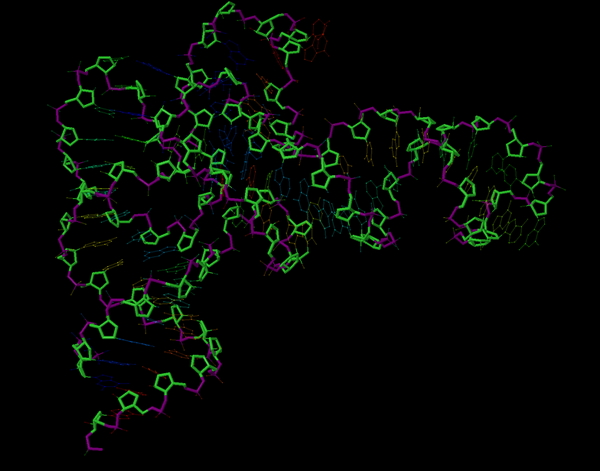
Рис.8. тРНК 1H3E
 Рис.9. Первый разрыв в 1H3E |
 Рис.10. Второй разрыв в 1H3E |
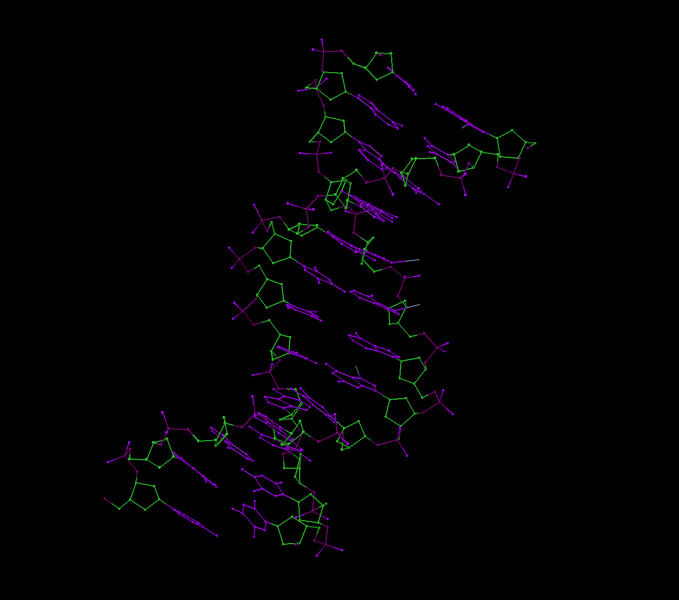
Рис.11. ДНК из ДНК-белкового комплекса 1PP7
Задание 3
Упр.1
Для структуры B-ДНК нужно было выбрать один из тиминов и посмотреть, какие из его атомов направлены в сторону большой бороздки ДНК, а какие - в сторону малой. Я выбрала тимин 34. Я решила , что в сторону большой бороздки явно обращены атомы С4, О4, С5 и СМ5, а сторону малой - N1, C2 и O2.
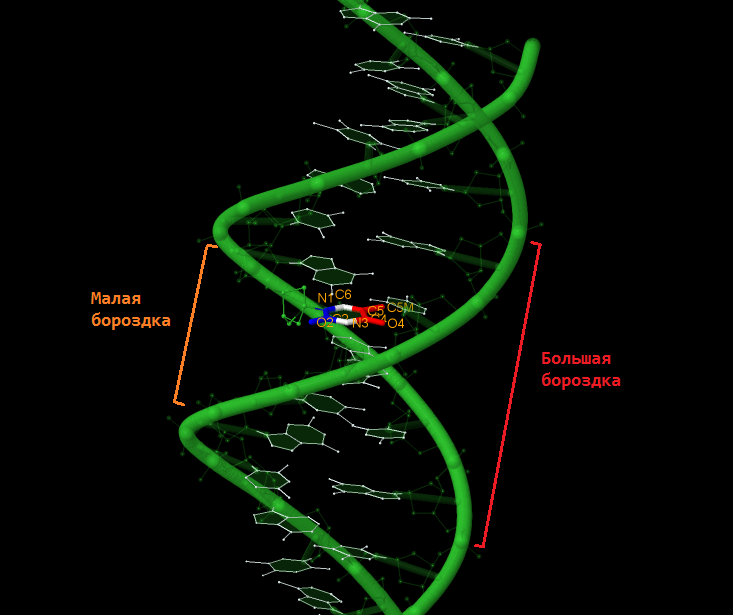
Рис.12. Тимин в B-ДНК. Красным выделены атомы, смотрящие в сторону большой бороздки, синим - в сторону малой, белым - остальные.
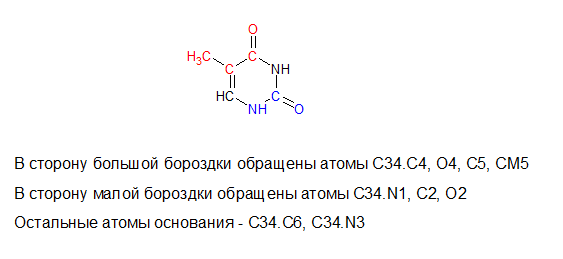
Рис.13. Тот же тимин, риснок выполнен с помощью ChemSketch.
Упр.2
Для А, В и Z-форм определялись такие параметры, как шаг спирали, ширина бороздок и др.
| А-форма | В-форма | Z-форма | |
| Тип спирали | правая | правая | левая |
| Шаг спирали (ангстрем) | 28 | 34 | 44 |
| Число оснований на виток | 11 | 10 | 12 |
| Ширина большой бороздки | 18.0 (от фосфата 6 цитозина) | 17.2 (от фосфата 14 цитозина) | 28.0 (от фосфата 9 гуанина) |
| Ширина малой бороздки | 8.0 (от фосфата 30 тимина) | 11.7 (от фосфата 30 тимина) | 7.2 (от фосфата 9 гуанина) |
Упр.3
Торсионные углы были измерены в Jmol для А и В форм ДНК. Если сравнить с приведенными на лекции значениями углов, у меня получились альфа-углы не того знака, и слишком большое значение дзета.
| Форма | alpha | beta | gamma | delta | epsilon | zeta | chi |
| А-ДНК | -51.7 | 174.8 | 41.7 | 79.1 | -147.8 | -75.1 | -157.2 |
| В-ДНК | -29.9 | 136.3 | 31.2 | 143.3 | -140.8 | -160.5 | -98.0 |
Задание 4
Упр.1
С помощью программ find_pair и analyze были найдены торсионные углы для каждого основания в ДНК из 1PP7 и тРНК из 1H3E. Средние значения этих углов приведены в таблице. Сильнее всего колеблется угол гамма - от -179 до 177 градусов.
| alpha | beta | gamma | delta | epsilon | zeta | chi | |
| DNA | -58.3 | -7.7 | 64.3 | 135.7 | -150.6 | -90.2 | -111.0 |
| Основание с отклоняющимися углами (№5, цитозин) | -97.5 | 71.0 | 178.3 | 125.3 | -149.8 | -68.0 | -171.3 |
| tRNA | -30.1 | 10.0 | 63.2 | 90.5 | -111.1 | -51.7 | -135.1 |
| Основание с отклоняющимися углами (№11, гуанин) | 147.0 | -164.9 | 177.8 | 84.4 | -139.9 | -70.6 | -174.3 |
Упр.2
В файле tRNA.out записаны водородные связи между основаниями. Ниже выделены цветом пары, образующие стебли тРНК:
Strand I Strand II Helix
1 (0.004) B:...1_:[..G]G-----C[..C]:..72_:B (0.003) |
2 (0.007) B:...2_:[..G]G-----C[..C]:..71_:B (0.005) |
3 (0.004) B:...3_:[..G]G-----C[..C]:..70_:B (0.004) |
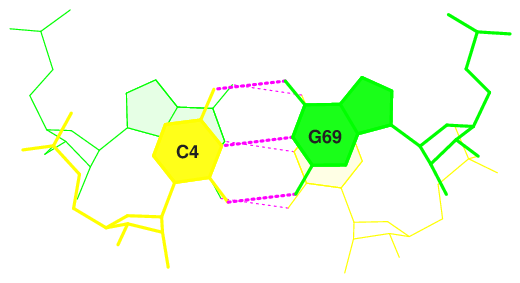
4 (0.003) B:...4_:[..C]C-----G[..G]:..69_:B (0.012) |
5 (0.004) B:...5_:[..A]A-----U[..U]:..68_:B (0.005) |
6 (0.005) B:...6_:[..G]G-----C[..C]:..67_:B (0.006) |
7 (0.007) B:...7_:[..G]Gx----C[..C]:..66_:B (0.003) |
8 (0.003) B:..49_:[..G]G-----C[..C]:..65_:B (0.006) |
9 (0.009) B:..50_:[..C]C-----G[..G]:..64_:B (0.006) |
10 (0.003) B:..51_:[..U]U-----A[..A]:..63_:B (0.007) |
11 (0.003) B:..52_:[..G]G-----C[..C]:..62_:B (0.004) |
12 (0.010) B:..53_:[..G]G----xC[..C]:..61_:B (0.005) |
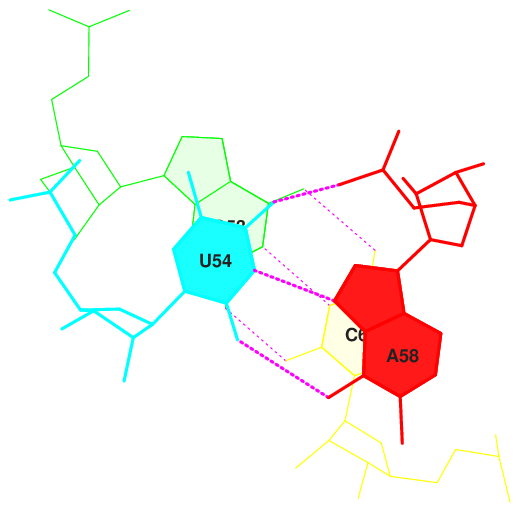
13 (0.011) B:..54_:[5MU]u-**-xA[MAD]:..58_:B (0.006) |
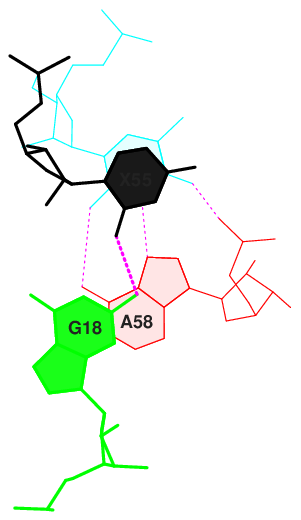
14 (0.045) B:..55_:[PSU]Px**+xG[..G]:..18_:B (0.004) x
15 (0.005) B:..47E:[..G]G-----C[..C]:..47_:B (0.006) |
16 (0.004) B:..47F:[..C]C-----G[..G]:..46_:B (0.006) |
17 (0.002) B:..47G:[..C]C----xG[..G]:..45_:B (0.010) |
18 (0.009) B:..47H:[..U]U-*---A[..A]:..20B:B (0.015) |
19 (0.008) B:..47I:[..U]U-*--xA[..A]:..21_:B (0.008) |
20 (0.006) B:..48_:[..C]Cx**+xG[..G]:..15_:B (0.008) x
21 (0.005) B:..19_:[..G]Gx---xC[..C]:..56_:B (0.005) +
22 (0.004) B:...8_:[..U]U-**--A[..A]:..14_:B (0.006) |
23 (0.017) B:...9_:[..U]Ux**--G[..G]:..13_:B (0.009) |
24 (0.005) B:..23_:[..G]G-----C[..C]:..12_:B (0.010) |
25 (0.005) B:..24_:[..G]G-----C[..C]:..11_:B (0.006) |
26 (0.006) B:..25_:[..G]G----xC[..C]:..10_:B (0.007) |
27 (0.007) B:..26_:[..G]G-*---U[..U]:..44_:B (0.011) |
28 (0.008) B:..27_:[..A]A-----U[..U]:..43_:B (0.007) |
29 (0.007) B:..28_:[..C]C-----G[..G]:..42_:B (0.008) |
30 (0.007) B:..29_:[..G]G-----C[..C]:..41_:B (0.006) |
31 (0.009) B:..30_:[..G]G-----C[..C]:..40_:B (0.003) |
32 (0.007) B:..31_:[..U]U----xA[..A]:..39_:B (0.006) |
33 (0.004) B:..32_:[..C]C-*---A[..A]:..37_:B (0.010) |
34 (0.007) B:..33_:[..U]Ux*--xA[..A]:..36_:B (0.009) x
35 (0.011) B:..47A:[..G]G-**--U[..U]:..47B:B (0.004) +
В структуре есть несколько неканонических пар - 5MU (5-метилуридин) и MAD (1-метиладенин), PSU (псевдоуридин) и гуанин, гуанин 26 и урацил 44, цитозин 32 и аденин 37, гуанин 13 и урацил 9.
Связи, стабилизирующие 3хмерную структуру тРНК, но не имеющие отношения к стеблям, должны связывать отдаленные участки тРНК - такие как G19 и C56, G15 и C48, U47H и A20B, U47I и A21, PSU55 и G18.
Упр.3
В файле tRNA.out можно найти величину перекрываний азотистых оснований при стекинг-взаимодействиях. Три пары с наибольшей площадью перекрывания я отметила оранжевым.
i2 3' 5' j2
/|\ |
| |
Strand I | | II
| |
| |
| \|/
i1 5' 3' j1
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GG/CC 1.63( 0.37) 0.00( 0.00) 0.25( 0.00) 0.24( 0.05) 2.11( 0.42)
2 GG/CC 2.14( 0.71) 0.00( 0.00) 0.31( 0.00) 0.54( 0.05) 2.99( 0.77)
3 GC/GC 6.59( 3.49) 0.00( 0.00) 0.00( 0.00) 5.81( 2.70) 12.40( 6.19)
4 CA/UG 0.41( 0.00) 0.00( 0.00) 1.73( 0.90) 0.00( 0.00) 2.14( 0.90)
5 AG/CU 2.86( 2.05) 0.00( 0.00) 0.80( 0.00) 0.00( 0.00) 3.66( 2.05)
6 GG/CC 3.34( 1.91) 0.00( 0.00) 0.47( 0.00) 0.00( 0.00) 3.81( 1.91)
7 GG/CC 2.30( 0.82) 0.00( 0.00) 0.99( 0.00) 0.00( 0.00) 3.29( 0.82)
8 GC/GC 3.50( 0.99) 0.00( 0.00) 0.00( 0.00) 6.81( 3.53) 10.31( 4.51)
9 CU/AG 1.64( 0.25) 0.00( 0.00) 0.03( 0.00) 1.64( 1.21) 3.31( 1.46)
10 UG/CA 0.52( 0.00) 0.00( 0.00) 3.18( 1.37) 0.00( 0.00) 3.71( 1.37)
11 GG/CC 4.52( 3.22) 0.00( 0.00) 0.16( 0.00) 0.00( 0.00) 4.68( 3.22)
12 Gu/AC 7.03( 2.02) 0.00( 0.00) 0.00( 0.00) 6.09( 2.73) 13.12( 4.75)
13 uP/GA 7.20( 3.39) 0.00( 0.00) 0.00( 0.00) 4.33( 0.17) 11.52( 3.56)
14 PG/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
15 GC/GC 4.75( 2.36) 0.00( 0.00) 0.00( 0.00) 5.83( 2.88) 10.58( 5.23)
16 CC/GG 0.24( 0.00) 0.00( 0.00) 0.04( 0.00) 3.52( 2.22) 3.80( 2.22)
17 CU/AG 0.00( 0.00) 0.00( 0.00) 0.65( 0.00) 3.36( 1.72) 4.01( 1.72)
18 UU/AA 0.00( 0.00) 0.00( 0.00) 1.80( 0.00) 0.00( 0.00) 1.80( 0.00)
19 UC/GA 0.00( 0.00) 0.00( 0.00) 2.45( 0.38) 0.00( 0.00) 2.45( 0.38)
20 CG/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
21 GU/AC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
22 UU/GA 0.00( 0.00) 5.43( 2.14) 0.00( 0.00) 0.00( 0.00) 5.43( 2.14)
23 UG/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 4.48( 2.20) 4.48( 2.20)
24 GG/CC 2.59( 1.12) 0.00( 0.00) 0.10( 0.00) 1.20( 0.36) 3.88( 1.48)
25 GG/CC 3.40( 1.93) 0.00( 0.00) 0.58( 0.00) 0.00( 0.00) 3.97( 1.93)
26 GG/UC 6.12( 4.17) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 6.12( 4.17)
27 GA/UU 6.04( 3.99) 0.00( 0.00) 0.00( 0.00) 4.21( 1.41) 10.25( 5.39)
28 AC/GU 4.31( 2.76) 0.00( 0.00) 0.00( 0.00) 5.30( 2.45) 9.61( 5.21)
29 CG/CG 0.00( 0.00) 0.00( 0.00) 5.13( 2.33) 0.00( 0.00) 5.13( 2.33)
30 GG/CC 3.78( 2.43) 0.00( 0.00) 0.36( 0.00) 0.00( 0.00) 4.15( 2.43)
31 GU/AC 4.58( 2.13) 0.00( 0.00) 0.00( 0.00) 4.88( 3.51) 9.47( 5.64)
32 UC/AA 4.17( 1.98) 0.00( 0.00) 0.00( 0.00) 0.36( 0.00) 4.52( 1.98)
33 CU/AA 2.43( 0.33) 0.00( 0.00) 0.00( 0.00) 4.80( 2.00) 7.24( 2.33)
34 UG/UA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Командой ex_str -8 stacking.pdb step8.pdb и stack2img -cdolt step8.pdb step8.ps было получено изображение стекинг-взаимодействий для выбранных пар.