Выбор объектов и получение выравнивания
Я выбрала домен RWD. Его функция неизвестна; название происходит от трех семейств белков, в которых содержится этот домен: RING-содержащие белки, белки, содержащие WD-повторы, и DEAD-подобные хеликазы. RWD относится к клану белков, подобных убиквитин-связывающим белкам. Структура формирует "сэндвич" из 4 антипараллельных бета-листов и 3 альфа-спиралей.
В базе Pfam содержится 1878 последовательностей, содержащих RWD, из 325 видов. Домен входит в 79 архитектур. Некоторые архитектуры действительно содержат DEAD и zf-RING, как и сказано в описании, а вот WD-повторов я не нашла. Я выбрала 2 архитектуры:

Архитектура RWD-DUF1115(RF06544), содержащая 157 последовательностей. 'DUF' означает 'Domain of unknown function', т.е. домен неизвестен.

Архитектура RWD-zf-RING_2(RF13639), содержащая 100 последовательностей. RING - это широко распространенный домен вида цинкового пальца, содержащий мотив Cys3HisCys4, который связывает 2 катиона цинка. RING-содержащие белки часто играют роль в убиквитинировании.
С помощью данных скриптов swisspfam-to-xls.py, uniprot_to_taxonomy.py, и скрипта Димы была получена таблица, содержащая ID белков семейства, их таксономию, и домены. Таблицу можно скачать здесь: [x].
В JalView были получено выравнивание последовательностей семейста RWD командой File-->Fetch Sequences. Выравнивание было раскрашено Blosum62 с консервативностью 20%. К последовательности RNF25_HUMAN прикреплена 3D-структура. Проект JalView: [x]
Для работы было выбрано 2 таксона - Arthropoda и Craniata. В среднем было выбрано около 15 последовательностей для каждого таксона среди обеих архитектур. С помощью скрипта filter-alignment.py из общего выравнивания были получены нужные последовательности. К описанию каждой были приписаны идентификаторы, показывающие принадлежность к таксону (Art для Arthropoda и Cra для Craniata) и домену. Выравнивание было открыто в JalView и из него были удалены 5 последовательностей с обрезанным началом. Выравнивание: [x]. Проект JalView: [x]
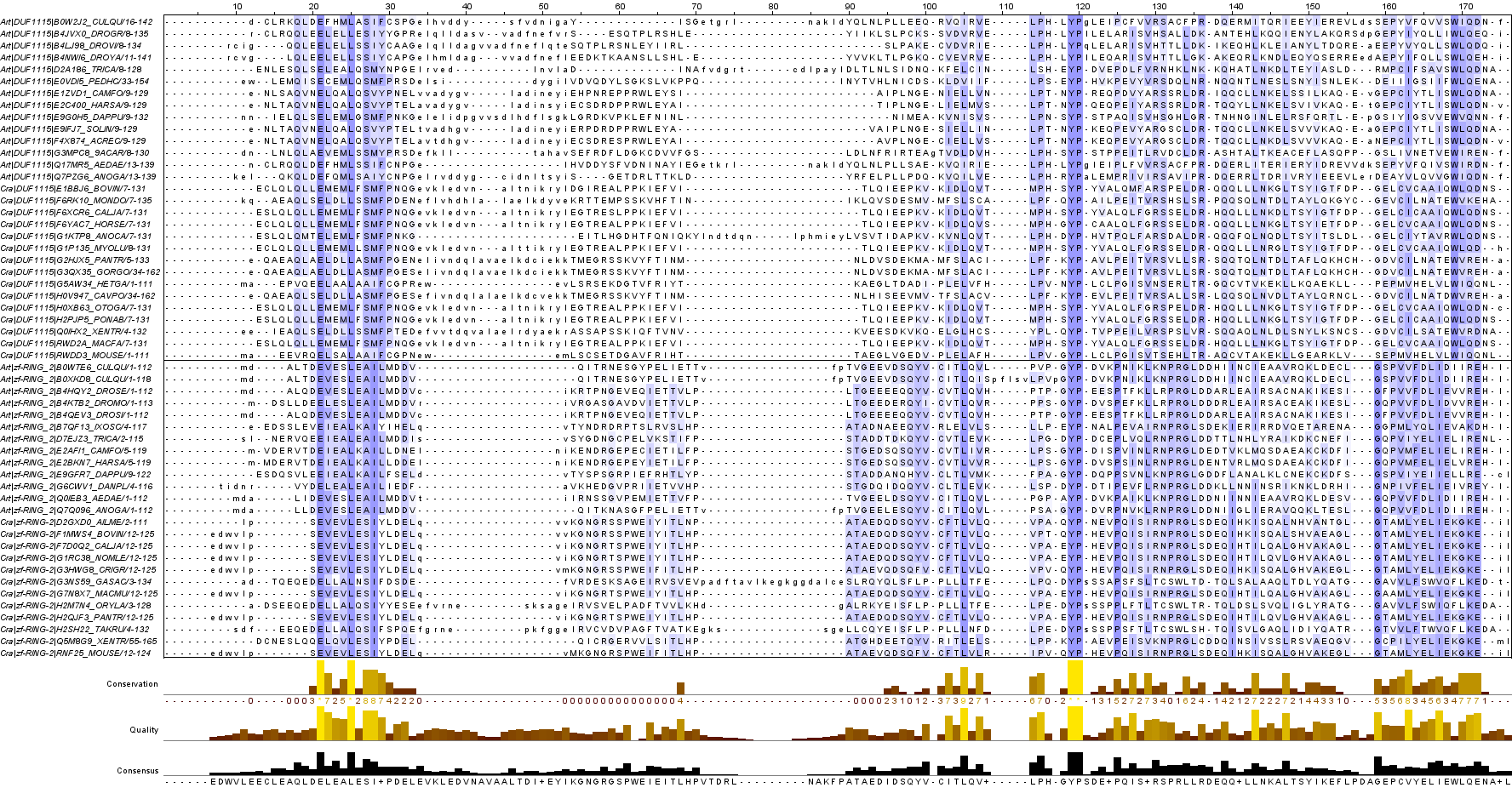
Рис.1. Выравнивание доменов RWD из архитектур RWD-DUF1115 и RWD-zf-RING_2. Раскрашивание по группам, Blosum62, с консервативностью 20%
Построение филогенетического дерева домена
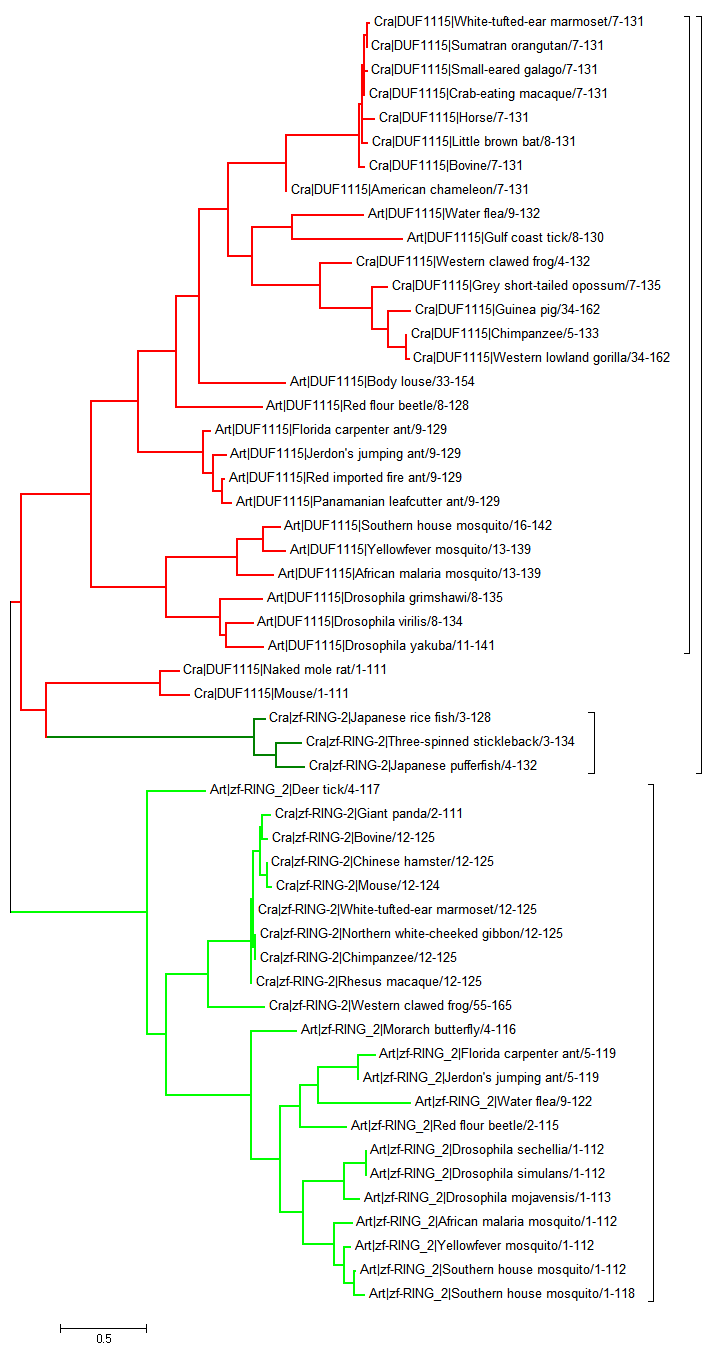
Выравнивание было открыто в программе MEGA, и по нему было реконструировано дерево методом Maximum likelihood. Результат представлен на рисунке 2:
Скобочная формула:
((((((((((((Cra|DUF1115|White-tufted-ear_marmoset/7-131:0.01351467,Cra|DUF1115|Sumatran_orangutan/7-131:0.00000000):0.01350212,(Cra|DUF1115|Small-eared_galago/7-131:0.01360409,Cra|DUF1115|Crab-eating_macaque/7-131:0.00000000):0.00000000):0.01352543,(Cra|DUF1115|Horse/7-131:0.06950729,Cra|DUF1115|Little_brown_bat/8-131:0.02710309):0.00000000):0.01523138,Cra|DUF1115|Bovine/7-131:0.02562599):0.42479297,Cra|DUF1115|American_chameleon/7-131:0.00000000):0.33802015,((Art|DUF1115|Water_flea/9-132:0.40982200,Art|DUF1115|Gulf_coast_tick/8-130:0.63966868):0.23308887,(Cra|DUF1115|Western_clawed_frog/4-132:0.17955236,(Cra|DUF1115|Grey_short-tailed_opossum/7-135:0.08844132,(Cra|DUF1115|Guinea_pig/34-162:0.12719166,(Cra|DUF1115|Chimpanzee/5-133:0.00000000,Cra|DUF1115|Western_lowland_gorilla/34-162:0.01286310):0.10476208):0.09564052):0.30080772):0.39642424):0.13916265):0.16935076,Art|DUF1115|Body_louse/33-154:0.50249941):0.12935119,Art|DUF1115|Red_flour_beetle/8-128:0.49534418):0.22375589,(Art|DUF1115|Florida_carpenter_ant/9-129:0.04093992,('Art|DUF1115|Jerdon''s jumping ant/9-129':0.07524855,(Art|DUF1115|Red_imported_fire_ant/9-129:0.01387682,Art|DUF1115|Panamanian_leafcutter_ant/9-129:0.05432418):0.05285230):0.05459955):0.37918501):0.27350120,(((Art|DUF1115|Southern_house_mosquito/16-142:0.09658266,Art|DUF1115|Yellowfever_mosquito/13-139:0.12812301):0.14933275,Art|DUF1115|African_malaria_mosquito/13-139:0.20839894):0.41434614,(Art|DUF1115|Drosophila_grimshawi/8-135:0.24487768,(Art|DUF1115|Drosophila_virilis/8-134:0.15670318,Art|DUF1115|Drosophila_yakuba/11-141:0.21753975):0.03481965):0.31299014):0.43578169):0.40409998,((Cra|DUF1115|Naked_mole_rat/1-111:0.10695660,Cra|DUF1115|Mouse/1-111:0.17008164):0.66145817,(Cra|zf-RING-2|Japanese_rice_fish/3-128:0.06079057,(Cra|zf-RING-2|Three-spinned_stickleback/3-134:0.14580748,Cra|zf-RING-2|Japanese_pufferfish/4-132:0.16060140):0.12750617):1.20617100):0.14585475):0.06159430,('Art|zf-RING_2|Deer tick/4-117':0.33746586,((((((Cra|zf-RING-2|Giant_panda/2-111:0.04894593,Cra|zf-RING-2|Bovine/12-125:0.02949084):0.01183511,(Cra|zf-RING-2|Chinese_hamster/12-125:0.00000000,Cra|zf-RING-2|Mouse/12-124:0.02590716):0.03911514):0.03980908,(Cra|zf-RING-2|White-tufted-ear_marmoset/12-125:0.00000000,(Cra|zf-RING-2|Northern_white-cheeked_gibbon/12-125:0.00000000,Cra|zf-RING-2|Chimpanzee/12-125:0.00000000):0.01291220):0.00000000):0.01292782,Cra|zf-RING-2|Rhesus_macaque/12-125:0.00000000):0.24740947,Cra|zf-RING-2|Western_clawed_frog/55-165:0.32287556):0.24660370,('Art|zf-RING_2|Morarch butterfly/4-116':0.25978951,(((('Art|zf-RING_2|Florida carpenter ant/5-119':0.09408165,'Art|zf-RING_2|Jerdon''s jumping ant/5-119':0.00000000):0.23204058,'Art|zf-RING_2|Water flea/9-122':0.53414136):0.10744353,'Art|zf-RING_2|Red flour beetle/2-115':0.26695868):0.11469437,((('Art|zf-RING_2|Drosophila sechellia/1-112':0.00000000,'Art|zf-RING_2|Drosophila simulans/1-112':0.00000000):0.12806110,'Art|zf-RING_2|Drosophila mojavensis/1-113':0.08933061):0.23583879,('Art|zf-RING_2|African malaria mosquito/1-112':0.10148582,('Art|zf-RING_2|Yellowfever mosquito/1-112':0.03980572,('Art|zf-RING_2|Southern house mosquito/1-112':0.01013159,'Art|zf-RING_2|Southern house mosquito/1-118':0.05915594):0.05815731):0.05491063):0.18204336):0.13258323):0.16748318):0.49701195):0.11016122):0.78994450);
Зеленым выделена ветвь архитектуры RWD-zf-RING_2, красным - архитектуры RWD-DUF1115. Темно-зеленым - затесавшиеся среди RWD-DUF1115 белки RWD-zf-RING_2, причем все 3 из рыб. Если не считать темно-зеленую ветвь, в дереве хорошо прослеживается деление на архитектуры. Деление на таксоны тоже довольно четкое.
В качестве подсемейства последовательностей я взяла последовательности с архитектурой RWD-zf-RING_2, т.к. они преимущественно в одной кладе. С помощью программ HMM был построен и откалиброван профиль по этим последовательностям, и по этому профилю проведен поиск среди всех последовательностей, содержащих RWD:
hmm2build hmm.out pr11_subfamily hmm2calibrate hmm.out hmm2search hmm.out pr11_all.fasta > pr11_subfamily
Из полученного файла была сделана таблица в excel: pr11_subfamily_tabl1.xlsx. В ней последовательности сортированы по E-value, к каждой приписано, есть ли в ней zf-RING домен, и по этим параметрам рассчитаны параметры True Positive, False Negative, False Positive и True Negative. Также рассчитаны чувствительность и селективность, и построена ROC-кривая.
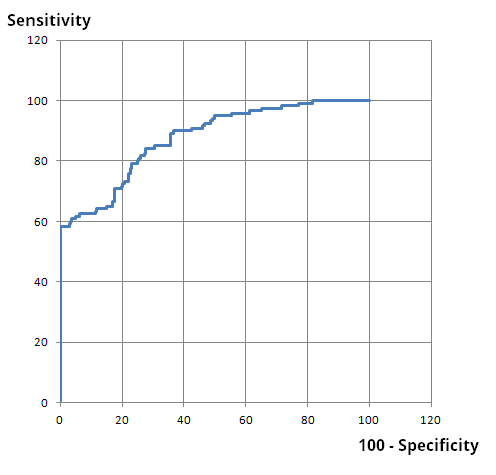
Поскольку ROC-кривая находится высоко над средней диагональю, то параметр неплохой (если ROC-кривая близка к диагонали, то выборка случайна). При пороге чувствительности 80% E-value равно 1,1E-6. При таком E-value:
| Приналежит подсемейству | Не приналежит подсемейству | Сумма | |
| Выше порога по профилю | 96 | 384 | 480 |
| Ниже порога по профилю | 24 | 1153 | 1177 |
| Сумма | 120 | 1537 | 1657 |