|
Работа с филогенетическим деревом нескольких бактерий
Отобранные бактерии
| Название | Мнемоника |
| Agrobacterium tumifaciens | AGRRK |
| Rhodobacter sphaeroides | RHOS4 |
| Ralstonia pickettii | RALPJ |
| Neisseria meningitidis | NEIMA |
| Escherichia coli | ECOLI |
| Yersinia pestis | YERPE |
| Yersinia pseudotuberculosis | YERPS |
| Proteus mirabilis | PROMH |
Скобочная формула дерева
(AGRRK, (RHOS4, (RALPJ, (NEIMA, (PROMH, (ECOLI, (YERPE,YERPS)))))));
Изображение дерева
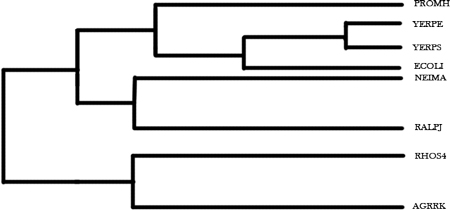
Ветви дерева
5 нетривиальных ветвей
(AGRRK, RHOS4) против (RALPJ, NEIMA, ECOLI, PROMH, YERPE, YERPS)
(RALPJ, NEIMA) против (AGRRK, RHOS4, ECOLI, PROMH, YERPE, YERPS)
(NEIMA, RALPJ, AGRRK, RHOS4) против (ECOLI, PROMH, YERPE, YERPS)
(PROMH, NEIMA, RALPJ, AGRRK, RHOS4) против (ECOLI, YERPE, YERPS)
(PROMH, ECOLI, NEIMA, RALPJ, AGRRK, RHOS4) против (YERPE, YERPS)
Таксономия отобранных бактерий
| Название | Мнемоника | Таксономия |
| Agrobacterium tumifaciens | AGRRK | Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Agrobacterium |
| Rhodobacter sphaeroides | RHOS4 | Bacteria; Proteobacteria; Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae; Rhodobacter |
| Ralstonia pickettii | RALPJ | Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Ralstonia |
| Neisseria meningitidis | NEIMA | Bacteria; Proteobacteria; Betaproteobacteria; Neisseriales; Neisseriaceae; Neisseria |
| Escherichia coli | ECOLI | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Escherichia |
| Yersinia pestis | YERPE | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Yersinia |
| Yersinia pseudotuberculosis | YERPS | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Yersinia |
| Proteus mirabilis | PROMH | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Proteus |

| 1 | Bacteria; Proteobacteria; Alphaproteobacteria |
| 2 | Bacteria; Proteobacteria; Betaproteobacteria |
| 3 | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae |
| 4 | Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales; Enterobacteriaceae; Yersinia |
Реконструкция филогенетического дерева
Для реконструкции филогенетического дерева был взят белок энолаза, мнемоника: ENO
(фермент, участвующий в предпоследнем этапе гликолиза. Энолаза катализирует
переход 2-фосфо-D-глицериновой кислоты в фосфоенолпируват. При этом от
2-фосфо-D-глицерата отщепляется одна молекула воды).
Последовательности белков энолазы отобранных мной бактерий из банка Swiss-Prot: prot.fasta
Далее, я получил множественное выравнивание программой muscle (mscl_prot.fasta), чтобы провести реконструкцию дерева при помощи программы fprotpars.
Результаты ее работы приведены ниже.
Скобочная формула:
((((RHOS4,AGRRK),(NEIMA,RALPJ)),((YERPS,YERPE),ECOLI)),PROMH);
Схематическое изображение:
+--RHOS4
+-----7
! +--AGRRK
+--------6
! ! +--NEIMA
! +-----5
+--4 +--RALPJ
! !
! ! +--YERPS
! ! +--3
1 +-----------2 +--YERPE
! !
! +-----ECOLI
!
+--------------------PROMH
Топология дерева совпадает с правильной.
Оценка эволюционных расстояний
При помощи программы frpotdist оценивалось эволюционное расстояние
между последовательностями. На выходе программа выдала файл: |*|
PROMH ECOLI YERPE YERPS RALPJ NEIMA AGRRK RHOS4
PROMH 0.000000 0.097775 0.108578 0.106114 0.457518 0.537510 0.562791 0.552563
ECOLI 0.097775 0.000000 0.056078 0.058451 0.434511 0.499523 0.545665 0.500399
YERPE 0.108578 0.056078 0.000000 0.002261 0.429656 0.497497 0.535921 0.520834
YERPS 0.106114 0.058451 0.002261 0.000000 0.426479 0.494206 0.532541 0.524325
RALPJ 0.457518 0.434511 0.429656 0.426479 0.000000 0.318335 0.433120 0.444920
NEIMA 0.537510 0.499523 0.497497 0.494206 0.318335 0.000000 0.483030 0.423113
AGRRK 0.562791 0.545665 0.535921 0.532541 0.433120 0.483030 0.000000 0.333097
RHOS4 0.552563 0.500399 0.520834 0.524325 0.444920 0.423113 0.333097 0.000000
Оценка отклонений расстояний от ультраметричности:
Рассмотрим RHOS4, NEIMA, YERPE.
1. d(RHOS4, NEIMA)=0.423113
2. d(NEIMA, YERPE)=0.497497
3. d(YERPE, RHOS4)=0.520834
Отклонение от ультраметричности равно примерно 4.48 %.
Рассмотрим ECOLI, YERPE, YERPS.
1. d(ECOLI, YERPE)=0.056078
2. d(YERPE, YERPS)=0.002261
3. d(YERPS, ECOLI)=0.058451
Отклонение от ультраметричности равно примерно 4.06 %.
Оценка отклонений расстояний от аддитивности:
Рассмотрим RHOS4, NEIMA, YERPE и YERPS.
1. d(RHOS4, NEIMA) + d(YERPE, YERPS)=0.423113 + 0.002261 = 0.425374
2. d(RHOS4, YERPE) + d(NEIMA, YERPS)=0.520834 + 0.494206 = 1.01504
3. d(RHOS4, YERPS) + d(NEIMA, YERPE)=0.524325 + 0.497497 = 1.021822
Наиболее близкие по зн. 2,3, также они больше 1, следовательно эта тройка почти удв. утв. аддитивности -
если есть 4 последовательности: A, B, C, D, - то из трех сумм
d(A,B) + d(C,D); d(A,C) + d(B,D); d(A,D) + d(B,C) две равны между собой и больше третьей.
Оценка эволюционных расстояний при помощи программы fneighbor
Было полученно две реконструкции дерева программой fneighbor, используя два алгоритма: UPGMA и Neighbor-Joining.
UPGMA
+--PROMH
+-----------3
! ! +-ECOLI
! +-2
! ! +YERPE
! +-1
--7 +YERPS
!
! +--------RALPJ
! +---4
! ! +--------NEIMA
+-6
! +---------AGRRK
+--5
+---------RHOS4
Neighbor-Joining
+-ECOLI
+-5
! ! +YERPE
! +-4
! +YERPS
!
! +-------RALPJ
! +-2
! ! +-----------NEIMA
6-------------3
! ! +----------AGRRK
! +----1
! +---------RHOS4
!
+---PROMH
Алгоритм UPGMA выдаёт укоренённое дерево с длинами ветвей, а Neighbor-Joining - неукоренённое.
Деревья, полученные при помощи разных алгоритмов не отличаются топологически, за исключением того,
что первое дерево имеет снова одну лишнюю ветвь почти, как и в результатах программы fprotpar,
также топология всех деревьев в общем то совпадает с правильным деревом.

|