
Семестры
Сайт ФББ МГУ
Kodomo Wiki
NCBI
RanHummer personal web-site
Поиск полиморфизмов у человека
Часть 1. Подготовка чтений
Очистка чтений с помощью Trimmomatic
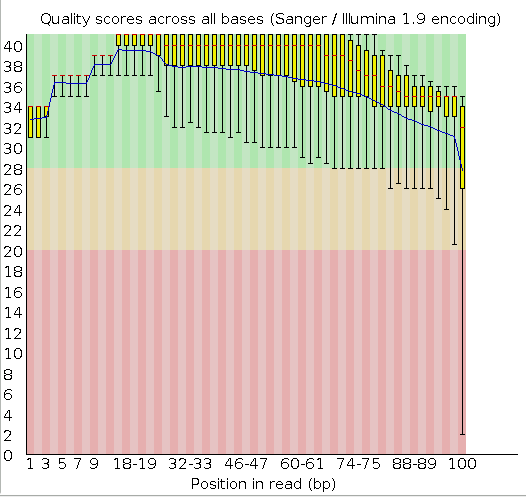
Рис. 1 Качество чтений из выдачи fastqc до очистки
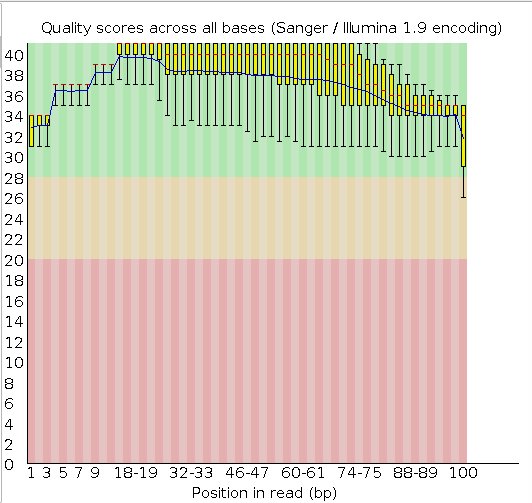
Рис. 2 Качество чтений из выдачи fastqc после очистки
fastqc chr12.fastq ## анализ качества чтений java -jar /usr/share/java/trimmomatic.jar SE -phred33 chr12.fastq chr12t.fastq TRAILING:20 MINLEN:50 ## удаление некачественных чтенийЧтения:
- До очистки: 7157
- После очистки: 7023
Часть 2. Картирование
bwa index chr12.fasta ## Индексация референсной последовательности bwa mem chr12.fasta chr12t.fastq > chr12_exom.sam ## Картирование чтения на референсную последовательность samtools view -bo chr12_exom.bam chr12_exom.sam ## Переводит данные о картированных чтениях в бинарник samtools sort chr12_exom.bam chr12_exom.sorted ## Сортировка чтений по координате начала чтения в референсе samtools index chr12_exom.sorted.bam ## Индексирование сортированных чтений samtools idxstats chr12_exom.sorted.bam ## Сколько чтений откартировались chr12 133851895 7024 0 ## Выход программы samtools idxstats. 1-ая колонка - имя референсной последовательности,
2-ая - её длина, 3-я - сколько чтений было откартировано, 4-ая - сколько чтений не было откартировано
Часть 3. Поиск SNP и инделей
samtools mpileup -uf chr12.fasta chr12_exom.sorted.bam > chr12_exom_SNPs.bcf ## создан файл с информацией об SNP bcftools call -cv chr12_exom_SNPs.bcf -o chr12_exom_SNPs.vcf ## формат bcf > формат vcf; отличие между референсом и чтениями
| Координата | Тип полиморфизма | Референс | Чтения | Глубина покрытия (в чтениях) | Качество |
| 9822387 | SNP | C | G | 31 | 190.009 |
| 9833398 | SNP | A | G | 30 | 141.008 |
| 66239573 | SNP | C | G | 1 | 6.20226 |
- Всего инделей: 3
- Всего SNP: 35
У большинства замен покрытие 1-2 чтения, однако есть замены с очень хорошим покрытием (максимальное - 133 чтения). Качество варьируется примерно от 6 до 220
Часть 4. Аннотация SNP
perl /nfs/srv/databases/annovar/convert2annovar.pl -format vcf4 chr12_exom_SNPs.vcf > chr12_exom_SNPs.avinput ## формат vcf > формат avinput perl /nfs/srv/databases/annovar/annotate_variation.pl --geneanno -out refGene -build hg19 -dbtype refGene chr12_exom_SNPs.avinput /nfs/srv/databases/annovar/humandb/ ## аннотация по б.д. refGene perl /nfs/srv/databases/annovar/annotate_variation.pl --filter -out dbsnp -dbtype snp138 -build hg19 chr12_exom_SNPs.avinput /nfs/srv/databases/annovar/humandb/ ## аннотация по б.д. dbsnp perl /nfs/srv/databases/annovar/annotate_variation.pl --filter -out 1000genomes -dbtype 1000g2014oct_all -build hg19 chr12_exom_SNPs.avinput /nfs/srv/databases/annovar/humandb/ ## аннотация по б.д. 1000genomes perl /nfs/srv/databases/annovar/annotate_variation.pl --filter -out gwas -dbtype gwasCatalog -build hg19 chr12_exom_SNPs.avinput /nfs/srv/databases/annovar/humandb/ ## аннотация по б.д. GWAS perl /nfs/srv/databases/annovar/annotate_variation.pl --filter -out clinvar -dbtype clinvar_20150629 -build hg19 chr12_exom_SNPs.avinput /nfs/srv/databases/annovar/humandb/ ## аннотация по б.д. Clinvar
- RefGene делит замены на: exonic, intronic, UTR3, ncRNA_exonic.
Из получившихся SNP:- 9 UTR3
- 3 exonic
- 26 intronic
- 1 ncRNA_intronic
- SNP попали в гены CLEC2D, RPSAP52, HMGA2 и TMEM263
- SNP привели к одной синонимичной замене и двум несинонимичным
- dbsnp: 28 SNP имеют идентификатор rs (б.д. RefSeq)
- 1000genomes: большинство замен распространённые (от 18% до 70%). Также есть несколько редких замен (несколько процентов) и одна очень редкая замена около 0.9%, но с плохим качеством и покрытием.
- Ни в базе данных GWAS, ни в базе данных Clinvar не была обнаружена ни одна из замен.