
Для определения функции и таксономии последовательности нуклеотидов, полученной в практикуме 6(консенсусная последовательность) я воспользовался алгоритмом blastn по базе данных Nucleotide collection (nr/nt) со следующими параметрами: длина слова 11 (по умолчанию), верхний порог E-value - 0.05 (выдача BLAST). Алгоритм blastn был выбран, т.к. о данной последовательности ничего не было известно, поэтому нужно найти сколько-нибудь похожие на неё последовательности разных видов.
Исследуемая нуклеотидная последовательность, вероятно, принадлежит морской полихете Brada inhabilis и является частью гена гистона H3 (процент идентичности - 99.14% (у следующей находки при том же E-value - 92.27%), E-value = 2e-172 (мало), AC: KJ530683).

Домен: Eukaryota
Класс: Polychaeta
Подотряд: Terebellida
Семейство: Flabelligeridae
Род: Brada
Вид: Brada inhabilis
В качестве исходной нуклеотидной последовательности был взят контиг сборки генома Xenopus laevis. Для поиска генов белков я воспользовался алгоритмом blastx (лучше транслировать последовательность нуклеотидов в последовательность аминокислот, она более консервативна, лучше найдутся гомологи) и в параметрах поиска указал БД swissprot (т.к. в записи только о подтвержденных белках), а также исключил из поиска организм Xenopus laevis (см. рис. 2), а также установил верхний порог E-value на уровне 0.01 для повышения достоверности находок.
С выдачей blastx можно ознакомиться по этой ссылке. 12 находок включают в себя "гомолог обратной транскриптазы", белок ORF2 ретротранспозона LINE-1 мыши домовой и человека (ORF2 также является обратной транскриптазой [3]), РНК-зависимая ДНК-полимераза (т.е. обратная транскриптаза) из мобильного элемента Drosophila melanogaster, полипротеин Pol из ретротранспозона R1 (который включает в себя обратную транскриптазу) и ген белка-эффектора RxLR effector protein 78 фитопатогена-оомицета Plasmopara viticola, включающий в себя регион, кодирующий обратную транскриптазу (см. рис. 3).

Исходя из вышеизложенного, предположу, что исследуемый контиг включает в себя ген ретротранспозона, кодирующий обратную транскриптазу.
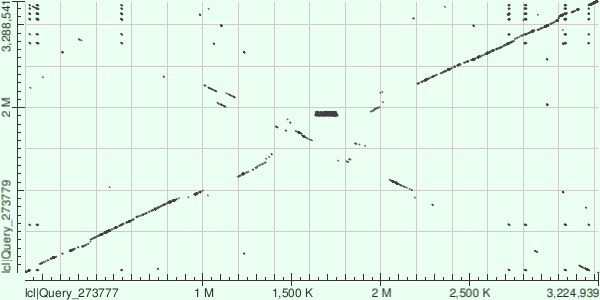
Для сравнения были выбраны хромосомы I бактерий рода Vibrio: Vibrio vulnificus и Vibrio parahaemolyticus. Для построения карты локального сходства я воспользовался megablast (Выдача BLAST). По карте видно, что геномы этих двух бактерий схожи, но есть значительные отличия - кроме шума, в районе 1M-1,200K видны инверсии и транслокации, в регионе 1,400K-2,200K видны многочисленные инверсии и делеции, инверсия есть в районе 3,100K-3,150K. Также можно отметить участок 1,650K-1,750K - это участок малой сложности (повторы).
1. Polychaeta (Arctic Ocean Diversity (ArcOD) project) Brada inhabilis