Для дальнейшей работы был выбран N-концевой домен убиквитин-лигазы RKP (AC - PF19322, средняя длина - 134 аминокислоты, среднее сходство - 52%, в seed - 30 последовательностей, в full - 184) и архитектуру из двух доменов: RKP_N (выбранный домен) и SPRY (такая архитектура - у 68 последовательностей из 184).
Файл со всеми последовательностями данного белка в формате .fasta. Список AC последовательностей с выбранной доменной архитектурой.
Все последовательности с выбранной доменной архитектурой я выгрузил в файл в формате .fasta. Выравнивание было построено в программе MEGA11 алгоритмом muscle.
Для построения HMM-профиля выбранной доменной архитектуры была использована команда
hmm2build profile chosen_seqs_aligned_short.fas
Далее профиль был откалиброван командой
hmm2calibrate profile Длина профиля - 1418 аминокислот
Затем я выполнил поиск по этому профилю в файле со всеми последовательностями белков с данным доменом - команда:
hmmsearch --cpu=2 profile PF19322_full.fasta > hmmsearch.txt
Лог поиска и составленная из него таблица по белкам
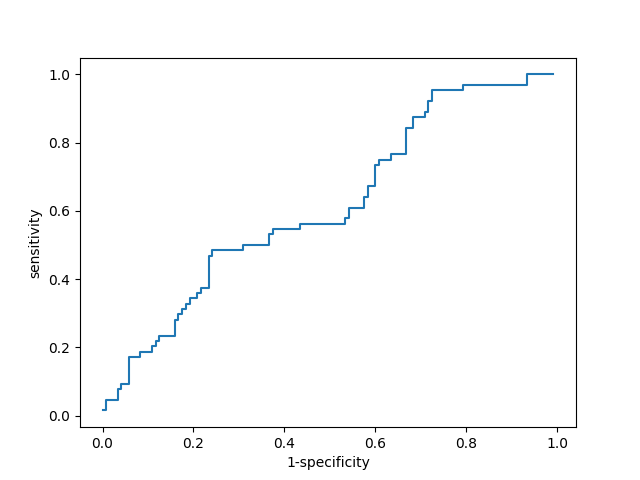
Для построения ROC-кривой я воспользовался скриптом своего однокурсника, Владислава Мурзина. Отмечу, что в hmmsearch попали все 184 последовательности из full, что свидетельствует о низком качестве HMM-профиля.
