Для работы был выбран домен Nucleoplasmin (PF03066). Пользуясь сайтом Uniprot
был осуществлен следующий поиск:
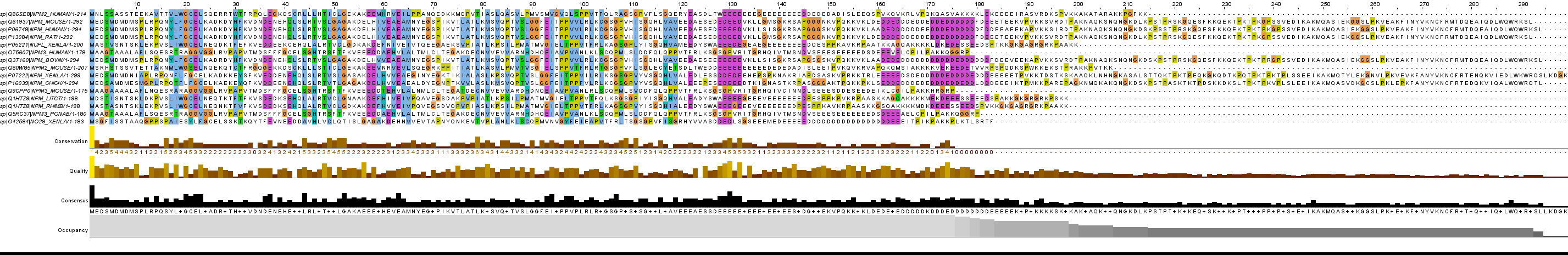
В результате было найдено 15 белков. Выравнивание этих белков представлено на рисунке 1. Проект выравнивания и fasta-файл можно скачать здесь:
Рисунок 1. Выравнивание найденных белков
Задание 2. Построение и калибровка профиля для целевого семейства
Далее был создан и откалиброван HMM-профиль по полученному выравниванию. Для этого были использованы
следующие команды:
| hmm2build -g hmm_nuc_profile3.out pr8_nuc_mfa.fa |
и
| hmm2calibrate hmm_nuc_profile3.out |
Полученный hmm-профиль вы можете увидеть здесь:
[ссылка]
Задание 3. Получение результатов поиска по профилю на множестве последовательностей SwissProt
Для поиска по профилю была применена следующая команда:
| hmm2search --domE 1000 --domT -50 hmm_nuc_profile3.out /srv/databases/emboss/data/uniprot/uniprot_sprot.fasta > pr8_nuc_search.out |
Рисунок 2. Полученная гистограмма

Задание 4. ROC-кривая
Для выбора порога необходимо максимизировать SE и SP (т.е. выбрать такую пару, для
которой максимально значение (SE+SP-1)). SE = TP/(TP+FN), a SP = TN/(TN+FP). Для выбранного профиля это
значение оказалось равным 0,5385, вес - 157,7. ( TP = 5, FN = 3, TN = 7, FP = 6).
[ссылка на таблицу с данными]
[ссылка на таблицу с построеной ROC-кривой]
Рисунок 3. ROC-кривая
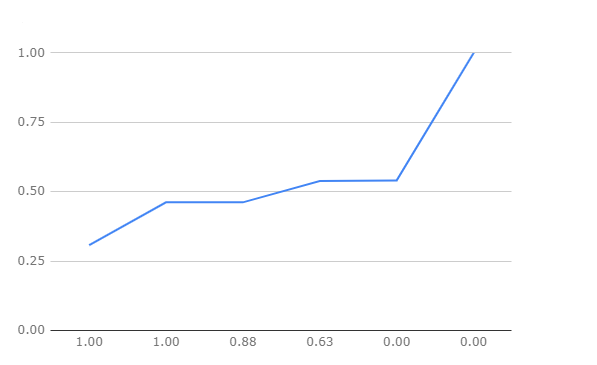
По рисунку 3 видно, что предложенный профиль позволяет отобрать представителей подсемейства, но качество его работы оставляет желать лучшего,
т.к. площадь под кривой весьма мала.