BLAST
Практикум 12 включает работу с NCBI BLAST — мощным инструментом для поиска последовательностей, сходных с конкретной заданной. Если уточнить, то работа велась с белками, соответственно, понадобившаяся разновидность BLAST называется BLASTp.
Поиск гомологов моего белка в Swiss-Prot
В первом задании нужно было найти последовательности белков, сходные с последовательностью моего белка, при помощи BLASTp по базе данных Swiss-Prot. После этого из результатов выбирались примеры с различными E-value, и для них было построено множественное выравнивание, свидетельствующее о гомологии.
В таблице 1 представлены использованные параметры BLAST.
| Параметр | Значение | Смысл |
|---|---|---|
| Accession number | ABC45135.1 | Код доступа исходной последовательности в NCBI. |
| Database | UniProtKB/Swiss-Prot(swissprot) | Где искать. |
| Max target sequences | 20000 | Верхнее ограничение размера выдачи. |
| Short queries | Yes | Автоподбор параметров в случае короткой последовательности. |
| Expect threshold | 50 | Верхний порог по E-value, на котором переставать искать. |
| Word size | 6 | Размер «затравки» локального выравнивания. |
| Max matches in a query range | 0 | Мы не ограничиваем число совпадений между исходной и найденной последовательностью. |
| Matrix | BLOSUM62 | По какой матрице считаем веса замен. |
| Gap Costs | Existence: 11 Extension: 1 | Штрафы за наличие и продолжение инделя. |
| Compositional adjustments | Conditional composition score matrix adjustment | Матрица будет подстроена под конкретный аминокислотный состав. |
| Filter low complexity regions | No | Не маскируем участки низкой сложности. |
| Mask for lookup table only | No | Если отметить это поле, то маска будет применяться только на этапе поиска «затравок», но уже не при построении выравнивания. |
| Mask lower case letters | No | Можно отметить, чтобы не учитывать строчные буквы (удобно для ручных выделений). |
По результатам поиска была создана полная таблица результатов (на второй её лист вынесены те последовательности, которые потом вошли во множественное выравнивание; процент покрытия вычислен формулой в Excel). Восемь позиций потом были выровнены в JalView, убедительно гомологичными из них оказались пять. Интересно, что пришлось исключить человеческий белок с UniProtAC Q8IX04, несмотря на то что у него было не худшее E-value: в нём всё-таки часто встречались различия с остальными. Рисунки 1 и 2 иллюстрируют блок, который был использован для доказательства гомологии. Итоговое выравнивание сохранено в проект.
Помимо консервативных позиций, есть и функционально консервативные.
Карта сходства двух белков
Для этого задания я выбрал два белка с UniProtAC W1Q7M1, U1LPR7. Так как они не принадлежат Swiss-Prot, их последовательности пришлось вручную копировать в окно BLAST (по AC они автоматически не находились). В BLAST я активировал опцию Align two or more sequences, после чего в выдаче стала доступна карта сходства заданных белков.
Карта сходства зависела от размера «затравки» BLAST и при значении 6 содержала 1 схожий участок, а при 3 — уже 2 (с E-value меньше 1). При этом у второго фрагмента E-value близко к единице (0,13), а в поле FT записей UniProt обоих белков был указан один птеринсвязывающий домен, соответствующий большому участку сходства, и больше ничего. Это наводит на мысль, что наиболее вероятна гомология только в одном участке. Карта сходства для затравки 3 приведена на рис. 3.
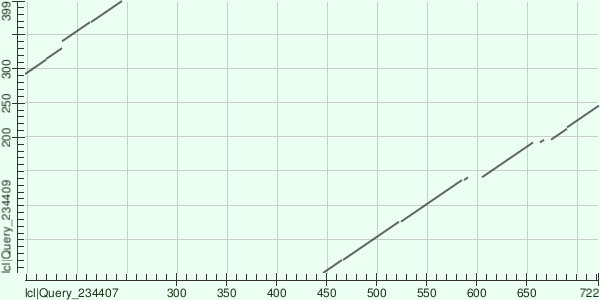
Если по карте пытаться описать второй белок относительно первого, не учитывая случайное сходство, то надо сказать, что произошла транслокация (с незначительными изменениями) участка с координатами 447..722 на места 2..246 нового белка. Остальные участки не гомологичны (можно сказать, претерпели делецию в первом белке и затем инсерцию во втором — появились de novo).
Играемся с параметрами
Поиск по случайной последовательности
Последовательность, которую я взял для поиска: MAFINNWERDPPYGGHLKSRET. Искал по Swiss-Prot. Ниже список параметров, отличных от значений по умолчанию:
- Max target sequences = 20000;
- Short queries = No;
- Expect threshold = 50;
- Word size = 2;
- Gap Costs = ‘Existence: 13 Extension: 1’.
Найдено 13 результатов. Их можно посмотреть в таблице. Интересно, что если для биологических последовательностей число результатов с низким E-value в выдаче превышает матожидание, то здесь ситуация противоположная и результатов даже меньше. Выходит, моя последовательность не слишком похожа на биологическую.
Поиск последовательности своего белка в Swiss-Prot
Здесь проводился поиск с разными параметрами. Каждый раз был изменён один параметр относительно запуска в первом задании. Таблицы здесь приводятся в текстовом формате и под теми же названиями, как сформированы в BLAST.
- Word size = 2 (таблица)
- Matrix = BLOSUM90 (таблица)
- Gap Costs = ‘Existence: 6 Extension: 2’ (таблица)
- Compositional adjustments = ‘No adjustment’(таблица)
Изменение размера «затравки» сильно повлияло на размер выдачи (увеличился с 594 до 772). Изменились и E-value: у второго в списке белка Q3B2R4 стало не 6e-125, а 8e-115.
Изменение матрицы весов тоже повысило число находок (до 642), при этом E-value белка Q3B2R4 изменилось не так сильно и составило 2e-122.
Новый штраф за индели мало изменил число находок (стало 590), но сильно повысил E-value: у Q3B2R4 стало 7e-101, и даже у самой исходной последовательности (сходство 100%) стало 3e-177. (С меньшим штрафом за открытие инделя стало проще набрать большие веса выравнивания).
Отмена подстраивания матрицы под аминокислотный состав тоже слабо повлияла на размер выдачи (стало 591), но сильно изменила веса. У Q3B2R4 стало 2e-111, а что более интересно, со второго места белок съехал аж на 7-е.