Отчёт по практикуму 4
Не всегда при построении дерева можно утверждать, что каждому белку из набора исходных данных соответствует один вид и что точка расхождения предковых форм белков совпадала с дивергенцией соответствующих видов. Практикум 4 как раз про этот случай.
Составление списка гомологичных белков, включающего паралоги
Здесь мы получаем последовательности достоверных гомологов белка CLPX_ECOLI у бактерий видов, отобранных в практикуме 1, или их близких родственников.
Для составления списка последовательность белка, гомологи которого нужно найти, была скачана из UniProt и использована в BLAST:
- blastp -query P0A6H1.fasta -db proteomes -out hits.blastp -evalue 0.001
Список находок в протеомах с kodomo можно скачать.
Реконструкция и визуализация
Дерево белков было реконструировано при помощи программы MEGA7 методом Maximum Likelihood после построения выравнивания программой muscle на kodomo. Его можнло скачать в формате Newick.
Приведём мнемоники трёх пар ортологов и трёх пар паралогов. Паралоги:
- Q898D1_CLOTE и Q899H3_CLOTE;
- HSLU_STAA8 и CLPX_STAA8;
- FTSH_STRPN и A0A0H2USJ7_STRPN.
Ортологи:
- CLPX_GEOKA и CLPX_BACAN;
- Q5SL436_GEOKA и A0A0H2USJ7_STRPN;
- Q5KUR3_GEOKA и Q895L6_CLOTE.
Рисунки 1 и 2 представляют построенное дерево. На рис. 1 ортологичные группы (от 2 белков) выделены утолщенными ветвями, а те, где белков более 3, окрашены. Окрашенные группы подписаны на обоих рисунках, на втором они ещё и схлопнуты.
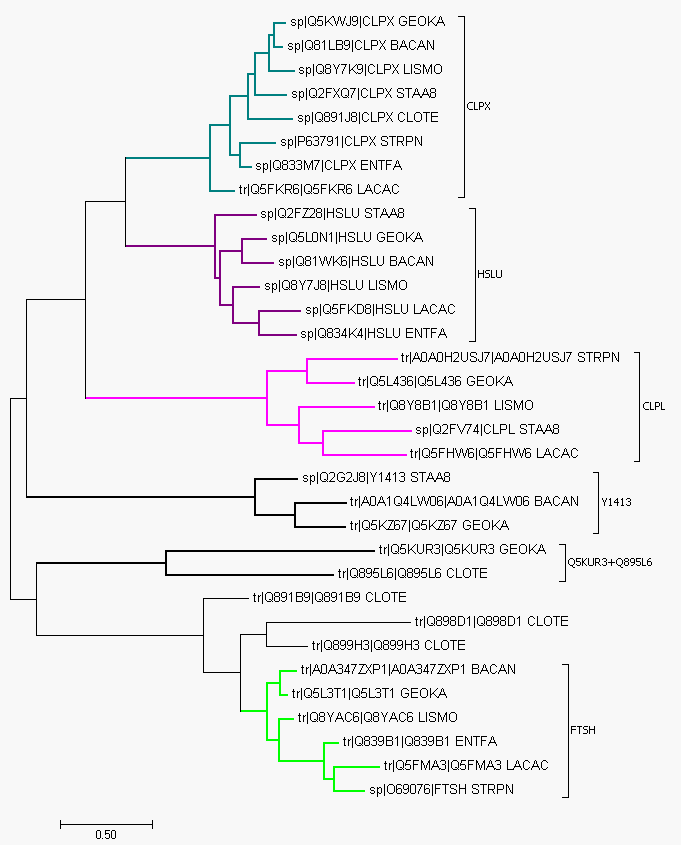
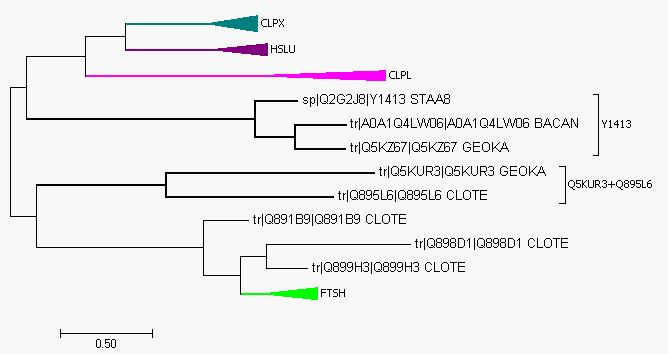
В группе CLPX белки из всех восьми бактерий — АТФ-связывающие субъединицы ClpX протеазы Clp, один из белков из TrEMBL. Оно не совпадает с филогенетическим деревом бактерий (ветви, отделяющей STRPN и ENTFA от остальных, быть не должно). HSLU — АТФазные субъединицы протеаз шести бактерий. Тут дерево тоже не совпало с принятым для бактерий (зря отделяет от остальных ENTFA, LACAC и LISMO). CLPL — субъединица того же белка, что и CLPX, с той же функцией. Поддерево опять не совпало, так как объединило STRPN и GEOKA против остальных. Наконец, FTSH — цинковая металлопротеаза. Дерево последовательности этого белка совпало с деревом для вошедших в него 6 бактерий.