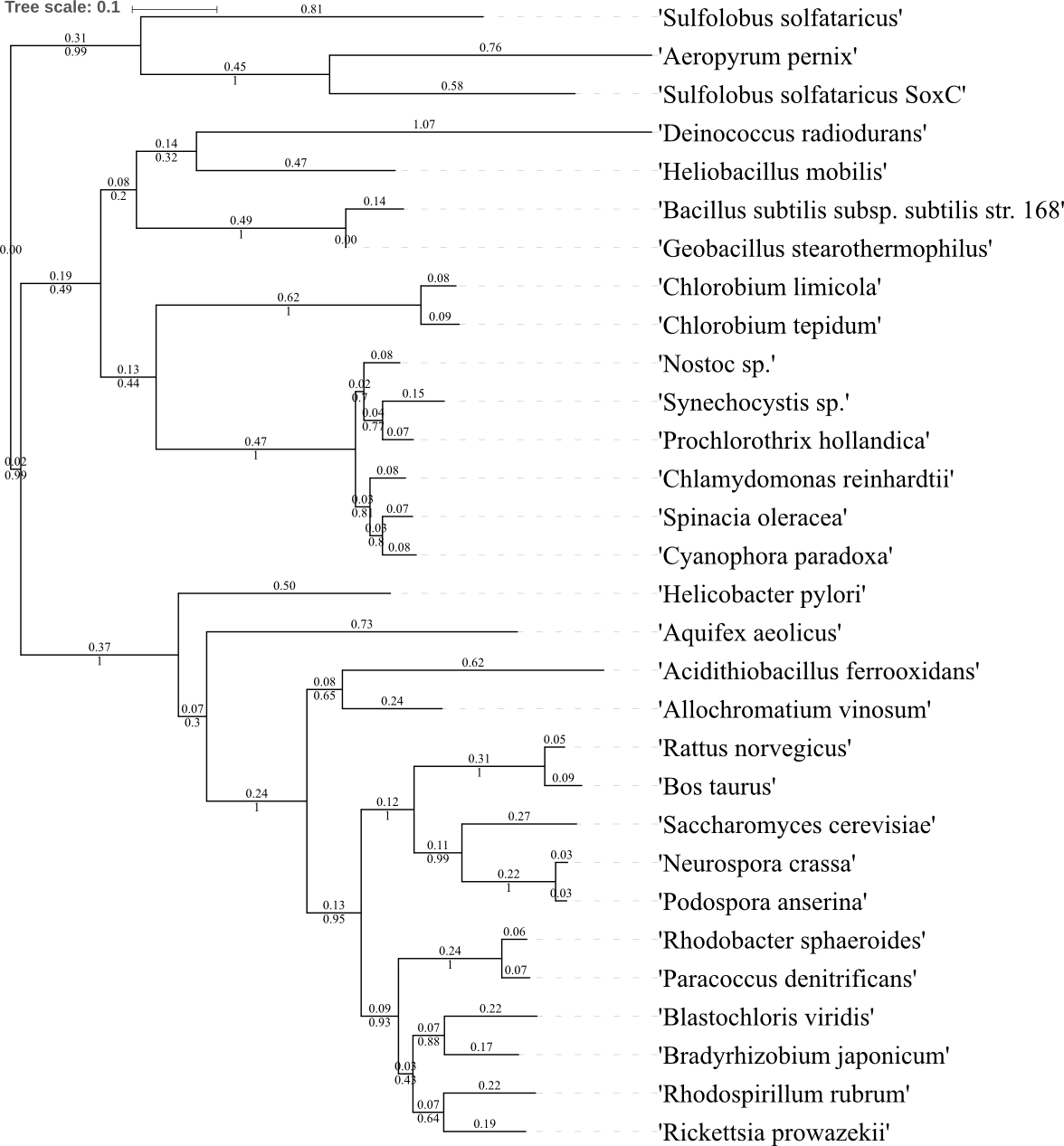I
При помощи программы retree из пакета PHYLIP было произведено новое укоренение уже имевшегося дерева в среднюю точку:
| Дерево, построенное JalView, с изначальным укоренением | Новое укоренение построенного при помощи JalView дерева |
|---|---|
| Дерево, построенное методом Neighbor-joining, в MEGA | «Идеальное» дерево |
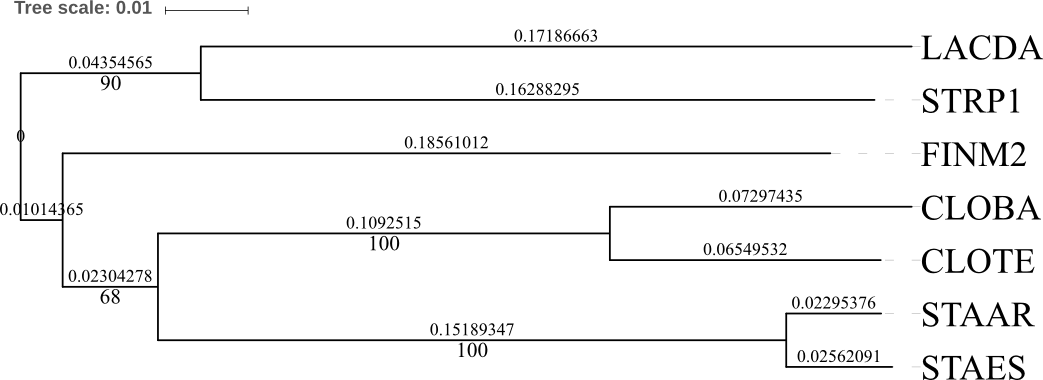
{STAES, STAAR, CLOTE, CLOBA} против {STRP1, LACDA, FINM2} (для сравнения: в «правильном» дереве укоренение находится в ветви {LACDA, STRP1, STAAR, STAES} против {CLOTE, CLOBA, FINM2},
а в построенном JalView дереве — в {STRP1} против остальных). Стоит отдельно прокомметировать случай с ветвью {STRP1, LACDA} против {FINM2, STAES, STAAR, CLOTE, CLOBA}: если бы на дереве не были нарисованы длины ветвей,
можно было бы подумать, что это небинарное (неразрешённое) дерево, однако это не так: просто длина этой нетривиальной ветви достаточно мала.
II
Далее было построено дерево с ипользованием метода «Maximum parsimony», причём для укоренения была использована внешняя группа в виде белка RS2 у кишечной палочки. Было получено выравнивание белков с белком внешнего организма:
На выравнивании чётко видно, что E. coli — это внешняя группа, общая консервативность позиций понизилась. Также получены деревья на основе этого выравнивания, укоренённые в ветку E. coli:
| Неукоренённое дерево с E. coli | Укоренённое дерево без E. coli |
|---|
III
Для начала снова построим в MEGA дерево, основываясь на выравнивании имеющихся белков, методом Neighbor-Joining, а также используя бутстрэп-тест. В итоге имеем cледующее дерево с бутстрэпами (изображение получено в другом сервисе, потому что скрипт, реализующий отрисовку деревьев, не поддерживает отображение бутстрэпов):
Оба дерева (Original tree и Bootstrap consensus tree) совпадают.
IV
Было произведено множественное выравнивание представленных цитохромов:
Прежде, чем начать строить деревья, уже следует отметить, что в предлагаемой статье филогения реконструировалась без учёта гэповых позиций; однако согласно некоторым исследованиям это не лучшая стратегия при построении филогенетического дерева. Воспроизводиться работа будет уже с учётом гэпов. Для построения дерева в статье был использован метод Neighbor-Joining; в нынешней работе будут использоваться этот и другие (Minimum evolution, Maximum likelihood) методы.
В результате работы были получены несколько деревьев:
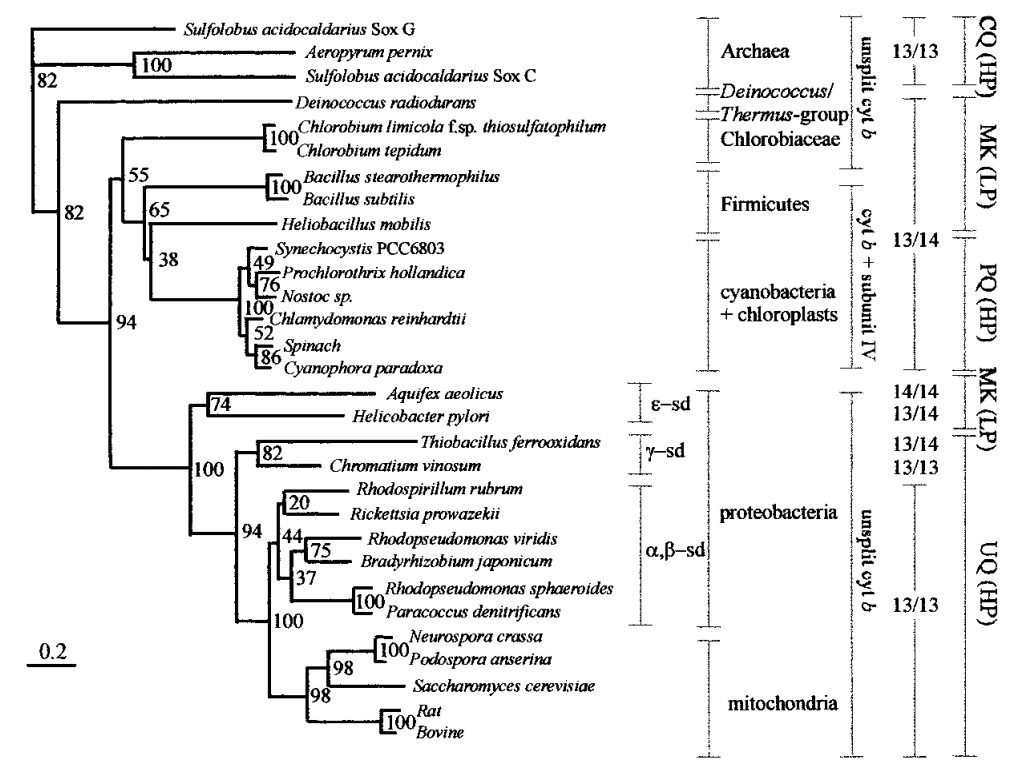 |
| Филогенетическое дерево из статьи (метод Neighbor-Joining) |
|---|
- Методы Neighbor-Joining и Minimum evolution дали абсолютно идентичные результаты;
- Все три дерева были автоматически укоренены в ветвь, отделяющую архей и бактерий (то, что это два разных домена, было доказано ещё в середине XX века; авторы статьи указывают, что используют белки архей именно как внешнюю группу);
- Хорошо видно, что на всех трёх деревьях длины ветвей, несущих группы Cianobacteria и Firmicutes, значительно меньше, чем остальных — возможно, это говорит о том, что эти группы действительно медленно эволюционируют;
- Значения бутстрэпов в целом похожи — естественно, они не совпадают полностью, однако это оправдывается тем, что у них бутстрэп-реплик было 5000, а в данной работе — 100 (например, для алгоритма Maximum likelihood 5000 бутстрэп реплик — это вычислительно очень много);
- Бактерия Deinococcus radioduran на всех трёх построенных деревьях находится топологически иначе, чем на дереве, представленном в статье;
- В целом все деревья хорошо отражают макроэволюционные связи, но не микро- — есть порой существенные отличия в топологии ветвей, отвечающих за протеобактерии или цианобактерии (однако принадлежность митохондрий к протеобактериям показана ясно).
Подводя итог, можно сказать, что в целом результаты работы принципиально не отличаются от результатов, представленных в статье 2000 года.