I
Были взяты следующие последовательности 16S рРНК:
- Clostridium botulinum (strain Alaska E43 / Type E3)
[CLOBA] - Staphylococcus aureus (strain MRSA252)
[STAAR] - Staphylococcus epidermidis (strain ATCC 12228)
[STAES] - Lactobacillus delbrueckii subsp. bulgaricus (strain ATCC 11842 / DSM 20081 / JCM 1002 / NBRC 13953 / NCIMB 11778)
[LACDA] - Streptococcus pyogenes serotype M1
[STRP1] - Finegoldia magna (strain ATCC 29328) (Peptostreptococcus magnus)
[FINM2] - Clostridium tetani (strain Massachusetts / E88)
[CLOTE]
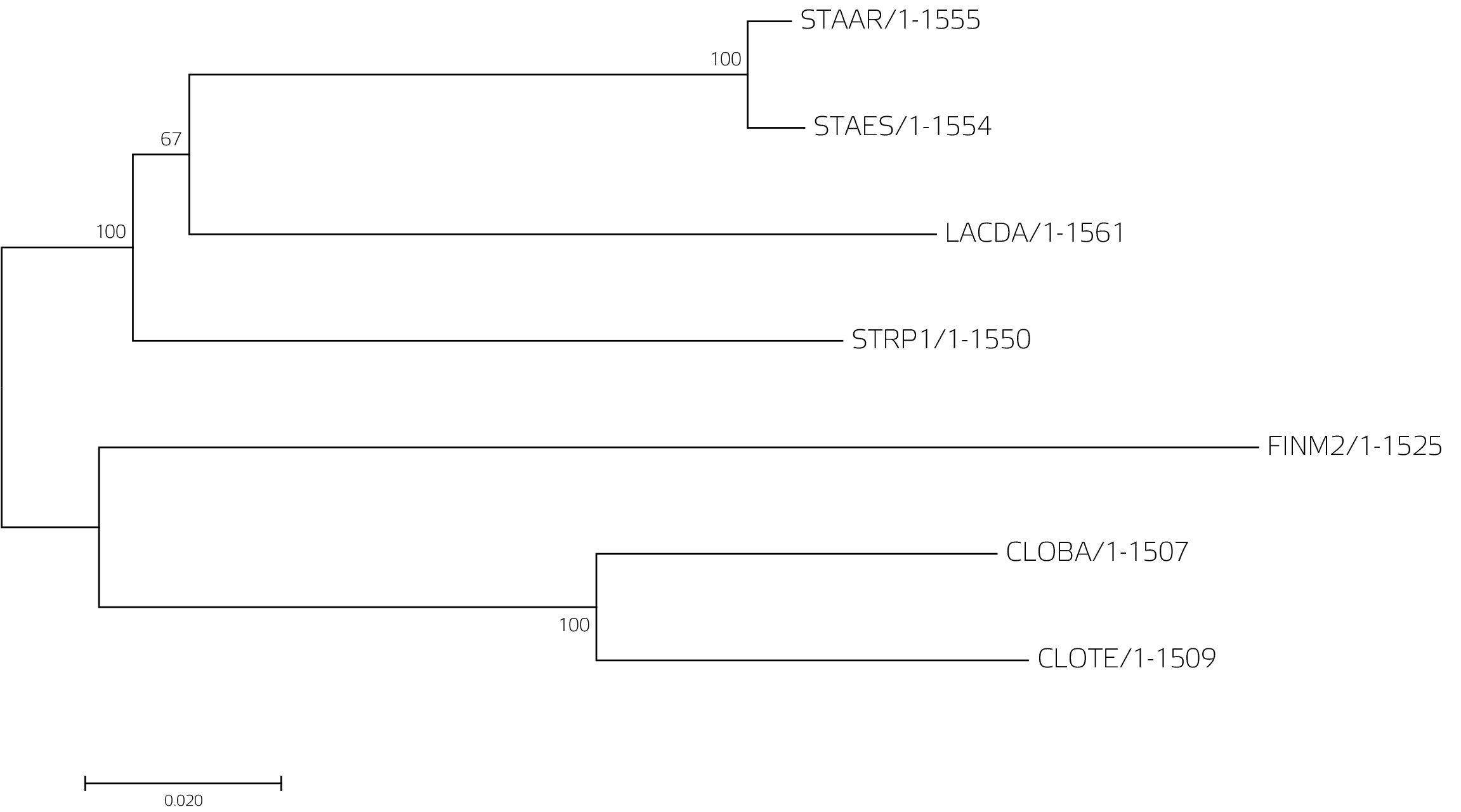
Полученное дерево немного отличается от «истинного»: так, STRP1 образует сестринскую группу по отношению к группе (LACDA, STAES, STAAR), хотя должен показать монофилию с LACDA и быть одной нетривиальной ветвью. Однако значение бутстрепа над веткой (LACDA, STAES, STAAR) показывает, что в принципе она не самая достоверная. В принципе, качество реконструкции сопоставимо с той, что была произведена по белкам.
II
По указанным организмам был произведён поиск гомологов предложенного белка при помощи BLAST (на локали был собран единый файл из всех протеомов, по нему вёлся поиск). Найденные последовательности были собраны в один файл, по которому было построено множественное выравнивание с последующей реконструкцией филогении. В результате получилось дерево следующего вида (см. ниже).
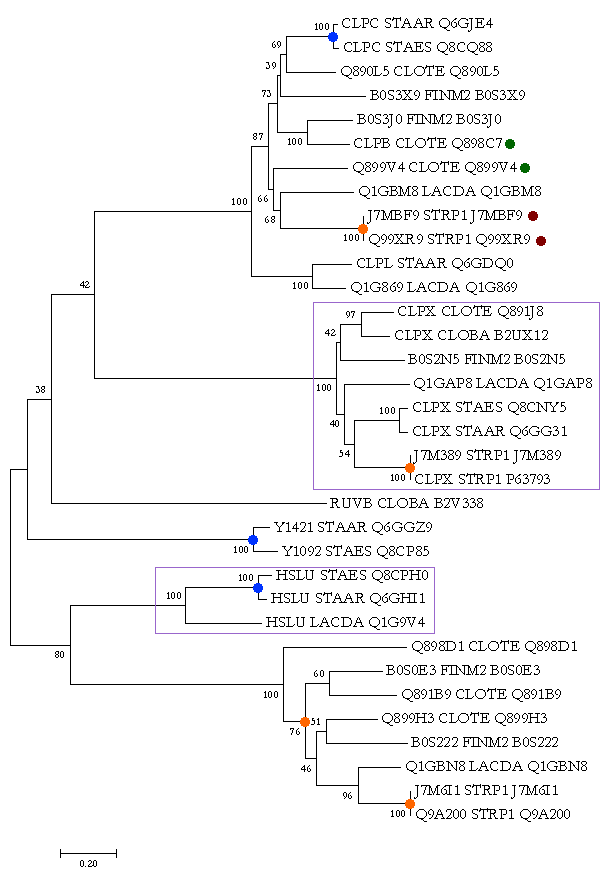
Оранжевыми точками на этом дереве обозначены моменты, когда происходила дупликация. Синими точками — моменты видообразования. Фиолетовое обведение — это ортологичные группы. Если же рядом с белком поставлена точка какого-либо цвета (зелёная или коричневая), то эти два белка — паралоги.