Поиск по сходству проводился на до боли знакомой структуре 4PNE. Из результатов поиска были взяты белки с порогом по RMSD (от 0.8 до 2.5) и длине выравнивания (как минимум 50% от длины нашей [4+2]-циклазы). В результате были выбраны 4 схожих белка (3BUS, 2O57, 1KPG и 1TPY), удовлетворяющих условиям. Всего с этими последовательностями выравнялось 207 остатков, а суммарный RMSD — 1.814. Краткое описание выбранных находок представлено в таблице ниже:
| Structure | Nres | NSSE | Consensus scores | |
| RMSD | Q-score | |||
| PDB 4pne:A | 271 | 18 | 1.5075 | 0.6099 |
| PDB 3bus:B | 243 | 16 | 1.1372 | 0.7448 |
| PDB 2o57:D | 265 | 18 | 1.1858 | 0.6756 |
| PDB 1kpg:D | 284 | 20 | 0.9275 | 0.6653 |
| PDB 1tpy:A | 286 | 20 | 0.8646 | 0.6683 |
А ниже представлено множественное выравнивание вторичных структур анализируемых белков:

Структурное выравнивание этих белков представлено ниже. Заметно, что в этом выравнивании консервативна даже позиция лиганда.
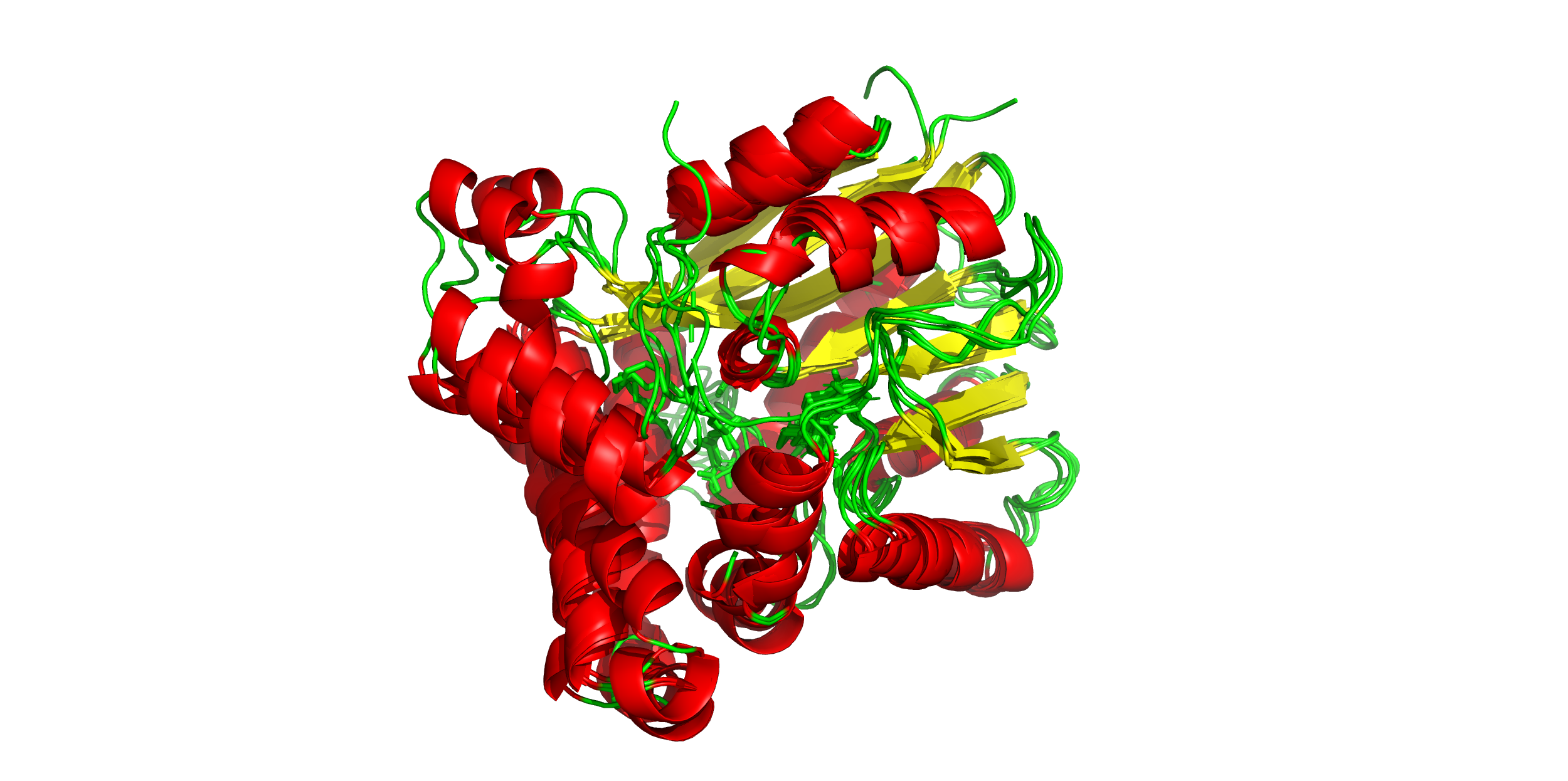
Выравнивание же последовательностей на самом деле оставляет желать лучшего. Много гэпов и мисмэтчей: такое выравнивание точно нельзя назвать хорошим (причем как алгоритмом Muscle, так и алгоритмами PDBeFold): Вот тут выравнивание, сделанное PDBeFold, а тут — при помощи Muscle. Плюс-минус консервативные блоки видны и схожи в обоих выравниваниях, отличия есть только в низковариабельных областях, что ожидаемо.