Для анализа был взят уже известный нам белок [4 + 2]-циклаза SpnF (4PNE). При помощи программы DSSP было произведено определение вторичной структуры. Для сравнительного анализа описания, данного в PDB, и предсказания программы, были выбраны две альфа-спирали и два участка одного большого бета-тяжа.
| Структура | Начало по DSSP | Конец по DSSP | Начало по PDB | Конец по PDB |
| β-тяж 1 | 167 | 180 | 167 | 180 |
| β-тяж 2 | 270 | 278 | 270 | 278 |
| α-спираль 1 | 16 | 34 | 15 | 35 |
| α-спираль 2 | 53 | 68 | 52 | 69 |
Визуализация разницы представлена ниже (красное — это совпавшие элементы альфа-спиралей, зелёное — это совпавшие элементы бета-листов, синее же — несовпавшие элементы альфа-спиралей):
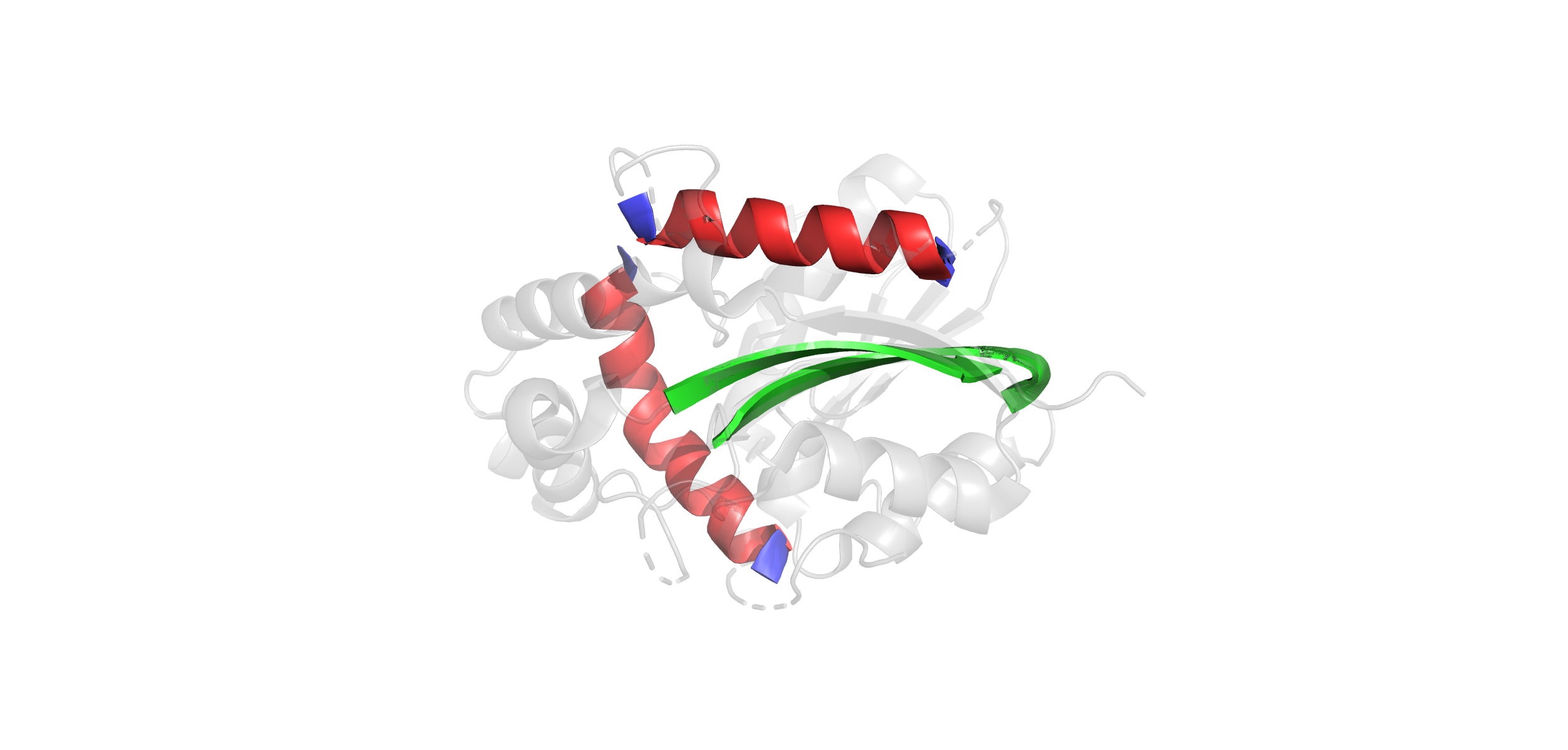
По всей видимости, алгоритм DSSP определяет лучше бета-листы, чем альфа-спиральные участки (по бета-листам совпадение полное, по альфа-спиралям есть орегулярные ошибки в одну аминокислоту).