Учебный сайт
Бредихина Данилы
- VII
- Электронная плотность. Рассеяние на электроне
Выбор структуры белка
Структура белка цитидиндезаминазы Bacillus subtilis (PDB ID 1UWZ, разрешение – 1.99 Å) была выбрана для дальнейшей работы. Было проверено, что структура удовлетворяет следующим требованиям:
- Сервис EDS (Electron Density Server) содержит информацию для выбранной структуры. Это означает, что в PDB, помимо модели структуры, помещены экспериментальные данные – файл структурных факторов (Structure factors).
- Имеются подходящие структурные гомологи белка. Для проверки этого был использован сервис PDBeFold, позволивший выполнить структурные выравнивания 1UWZ с моделями из PDB. Было получено 76 результатов поиска. Если ограничиться параметрами 0.8 Å ≤ RMSD ≤ 2.5 Å и 68 ≤ Nalgn ≤ 125 (от 50% до 92% числа остатков 1UWZ), то среди подходящих структур будут, например, 1R5T, 1MQ0, 2FR5, 3MPZ, 3R2N.
[RMSD (Root Mean Square Deviation) является оценкой среднего расстояния между Cα-атомами соответствующих остатков при суперпозиции структур. Nalgn – длина выравнивания, т. е. число сопоставленных остатков. При наложении структур их ориентация оптимизируется так, чтобы минимизировать RMSD и максимизировать Nalgn.]
Структурные факторы
Модель структуры, файл mmCIF (macromolecular Crystallographic Information File) 1UWZ и файл структурных факторов были загружены из PDB. Каждый структурный фактор расположен в отдельной строке, представляющей собой последовательность чисел, при этом прямого соответствия атомам модели нет. Число атомов в модели можно определить по файлам .pdb или .cif (mmCIF), число отражений (reflections) – по файлу структурных факторов:
grep -Eh ^ATOM\|^HETATM 1UWZ.pdb | wc -l # 2126 = number of atoms
grep -Eh ^ATOM\|^HETATM 1UWZ.cif | wc -l # 2126 = number of atoms
grep -Eh '^(\d ){3}' 1uwz-sf.cif | wc -l # 15661 = number of reflections
Поля, приведённые в файлах структурных факторов, могут отличаться от файла к файлу. В файле 1uwz-sf.cif присутствуют следующие данные для каждого отражения (в том числе h, k и l – индексы рефлекса):
Crystal_ID λ_ID Scale_Group_Code h k l Status F_mean F_sigma 1 1 1 -37 1 11 o 68.6 27.6 1 1 1 -37 1 12 o 79.6 28.9 1 1 1 -37 1 13 o 52.3 24.6
Изображение электронной плотности
Файл с картой электронной плотности для 1UWZ был загружен с сайта EDS. Для визуализации структуры и электронной плотности была использована программа для визуализации молекулярных данных PyMol.
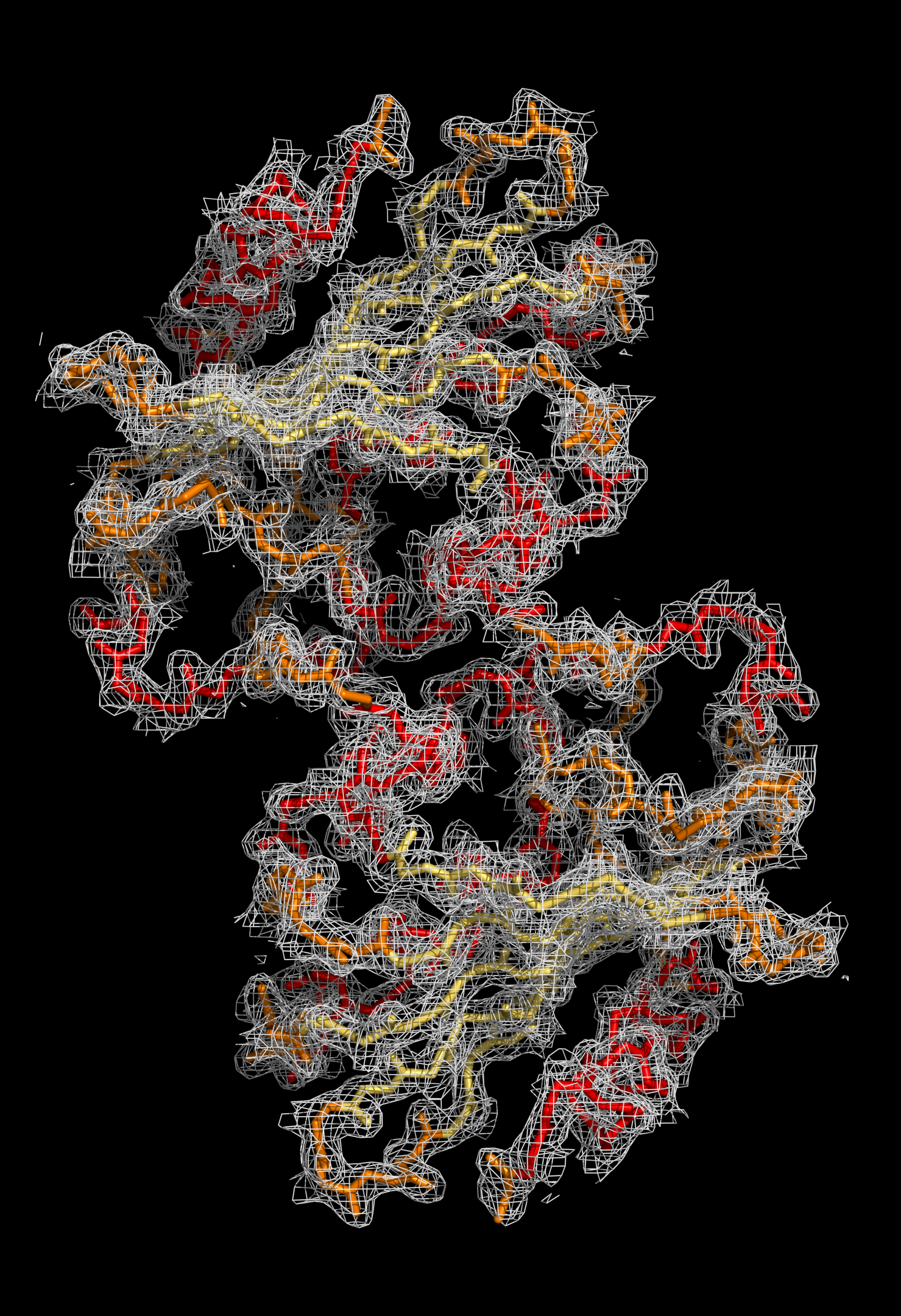
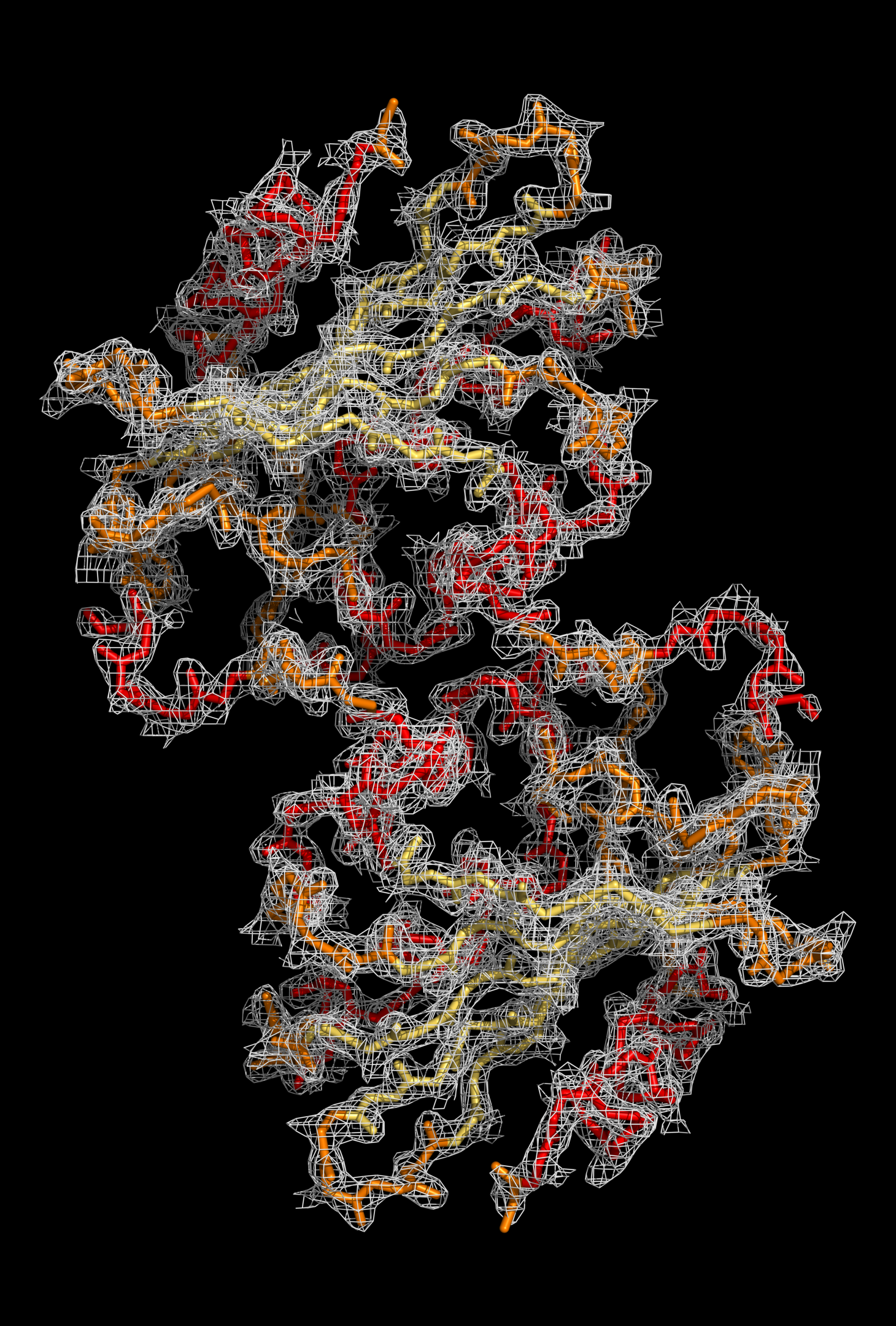
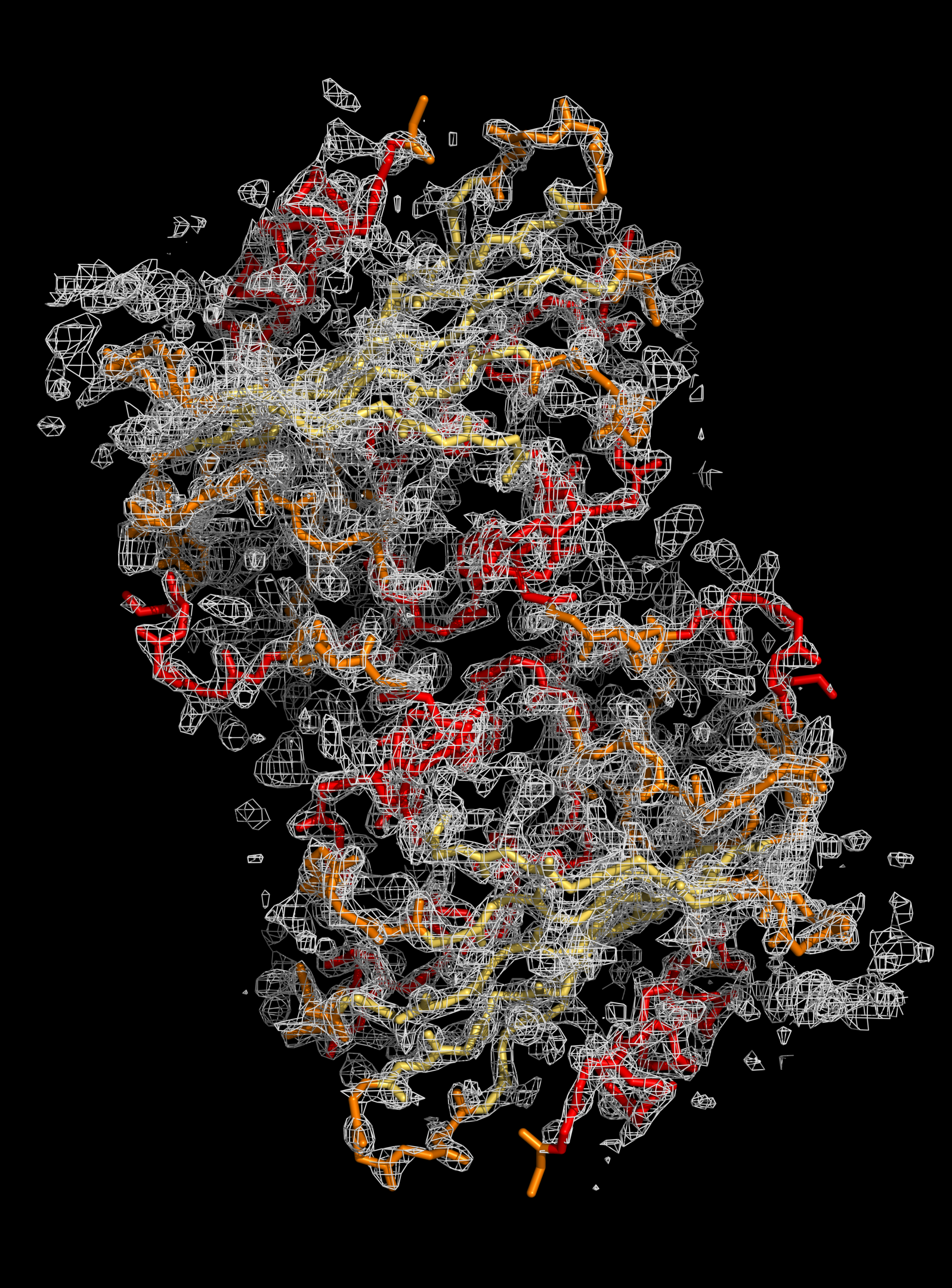
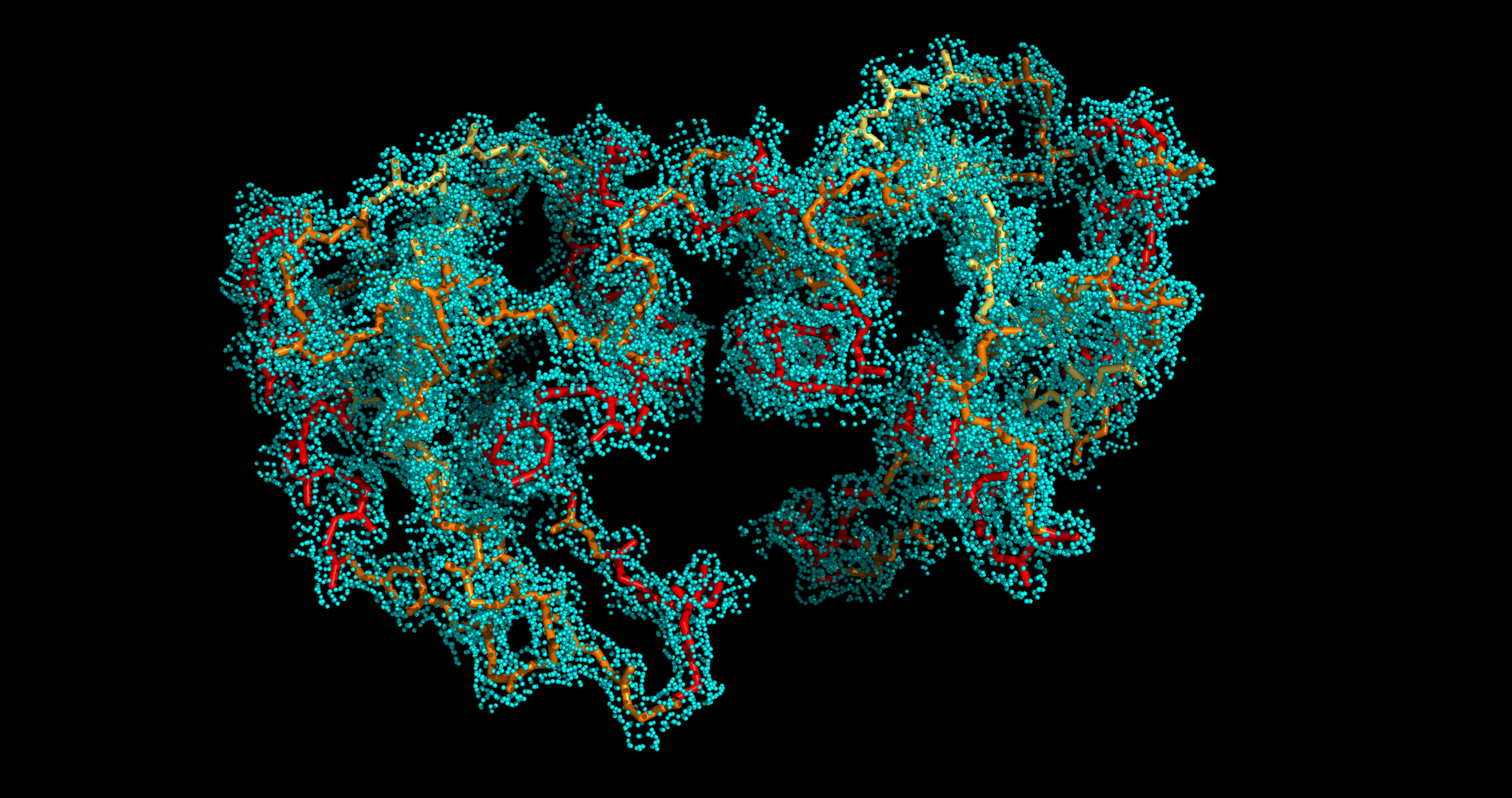
↑ Изображения электронной плотности вокруг полипептидной цепи (для уровней изолиний 1.0 σ, 1.5 σ и 2.0 σ на указанных расстояниях от выбранного множества – параметр carve) были получены, в частности, с помощью команд:
select backbone, name c+n+o+na isomesh backbone_map, 1uwz_map, 1, backbone, carve=2 isomesh backbone_map, 1uwz_map, 1.5, backbone, carve=2 isomesh backbone_map, 1uwz_map, 2, backbone, carve=5 isodot backbone_map, 1uwz_map, 1, backbone, carve=2 color cyan, backbone_map
Электронная плотность вокруг аминокислотных остатков пролина (Pro85); аланина (Ala124) и фенилаланина (Phe125); тирозина (Tyr21), серина (Ser22) и лизина (Lys23); цистеина (Cys97) и валина (Val92) показана на изображениях ниже (для уровня 1.0 σ). ↓
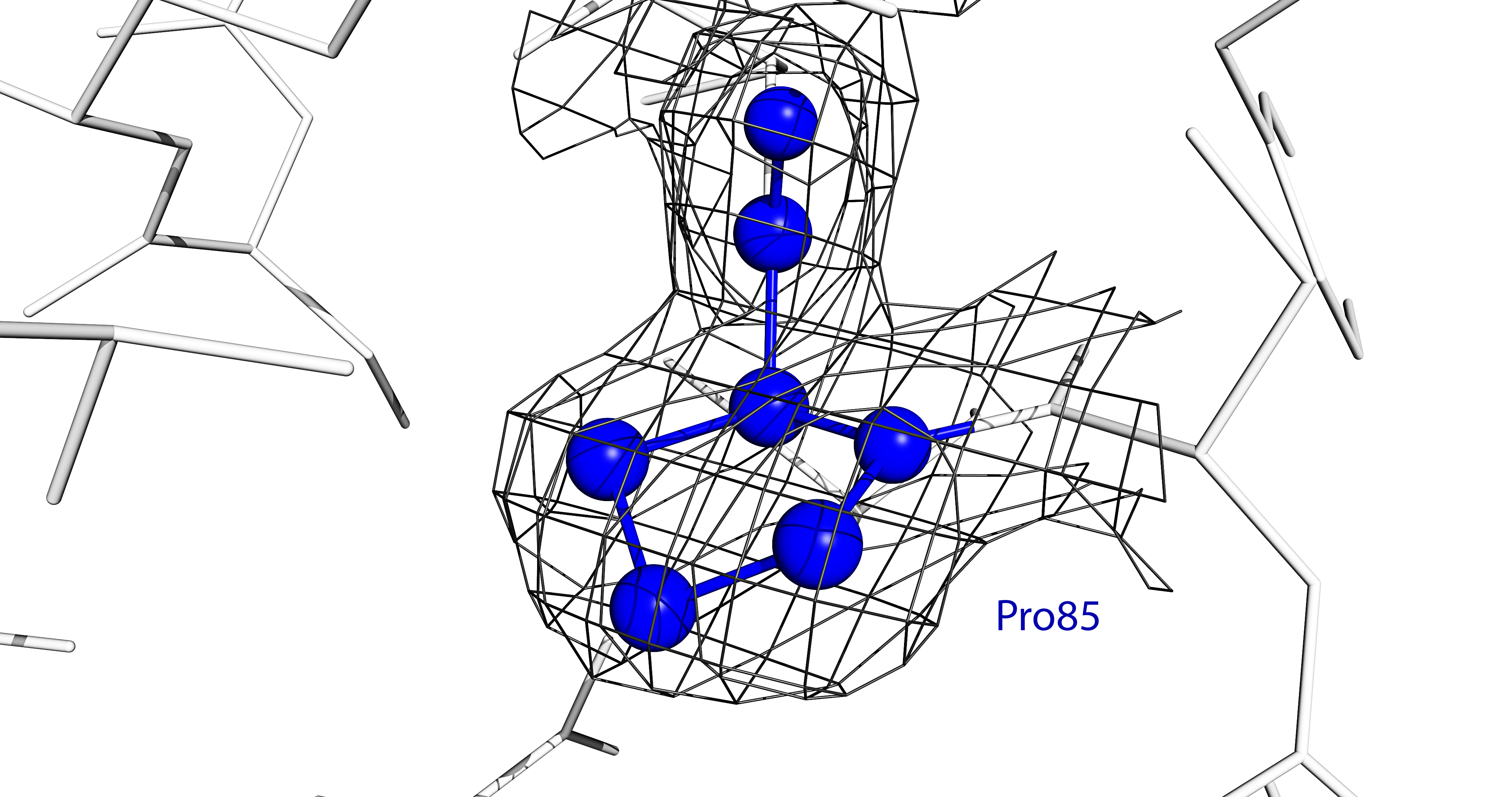
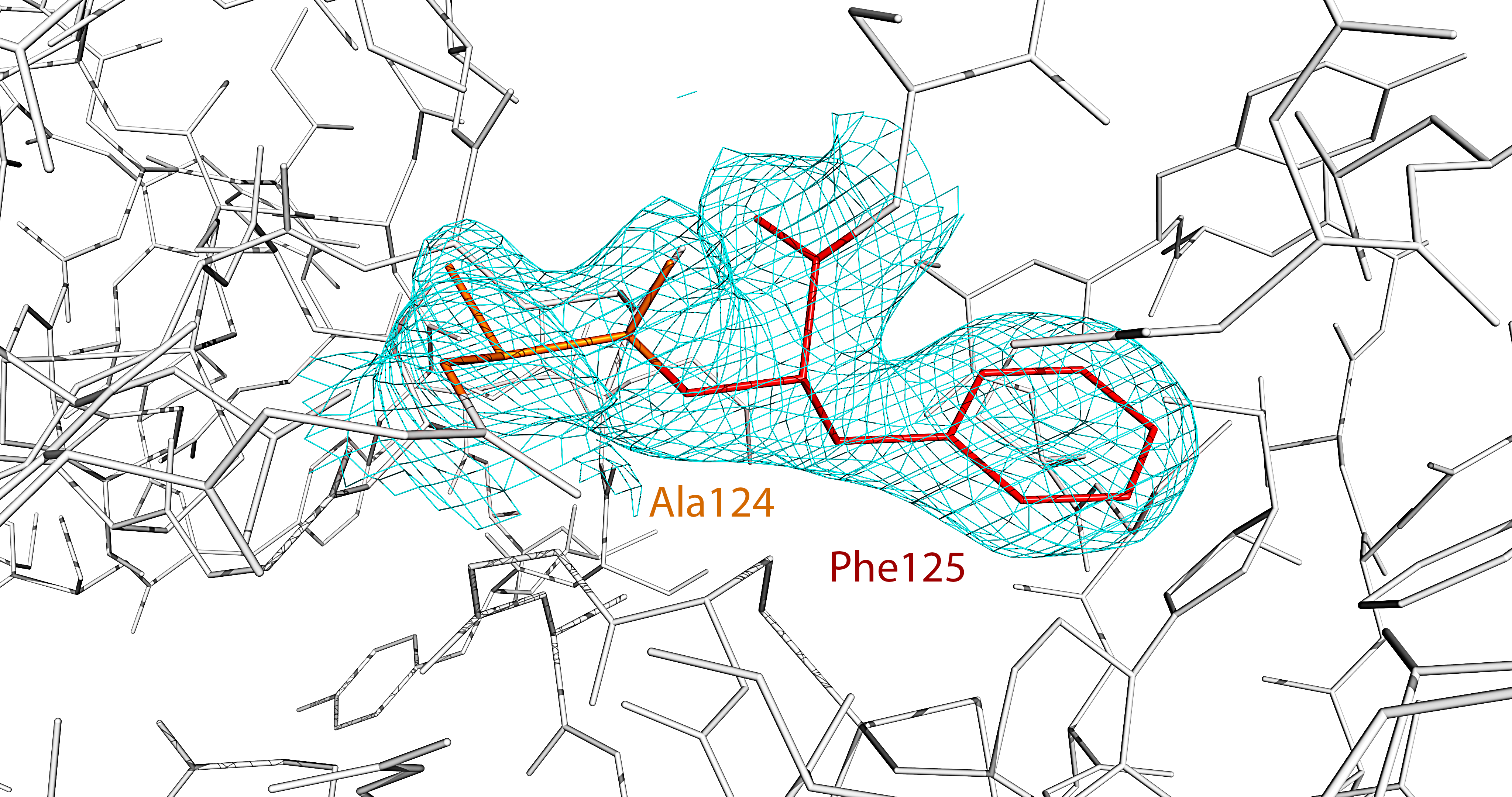
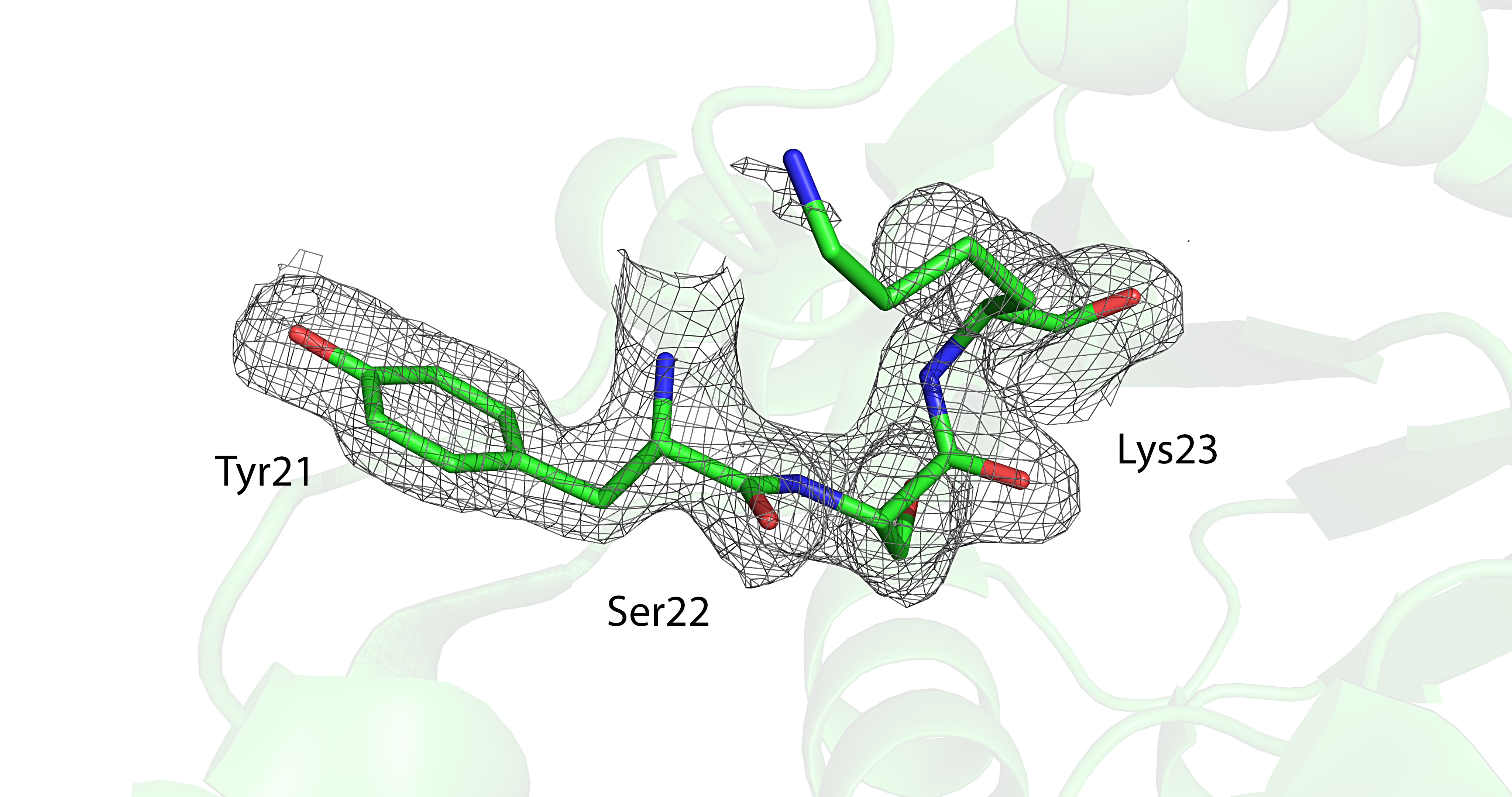
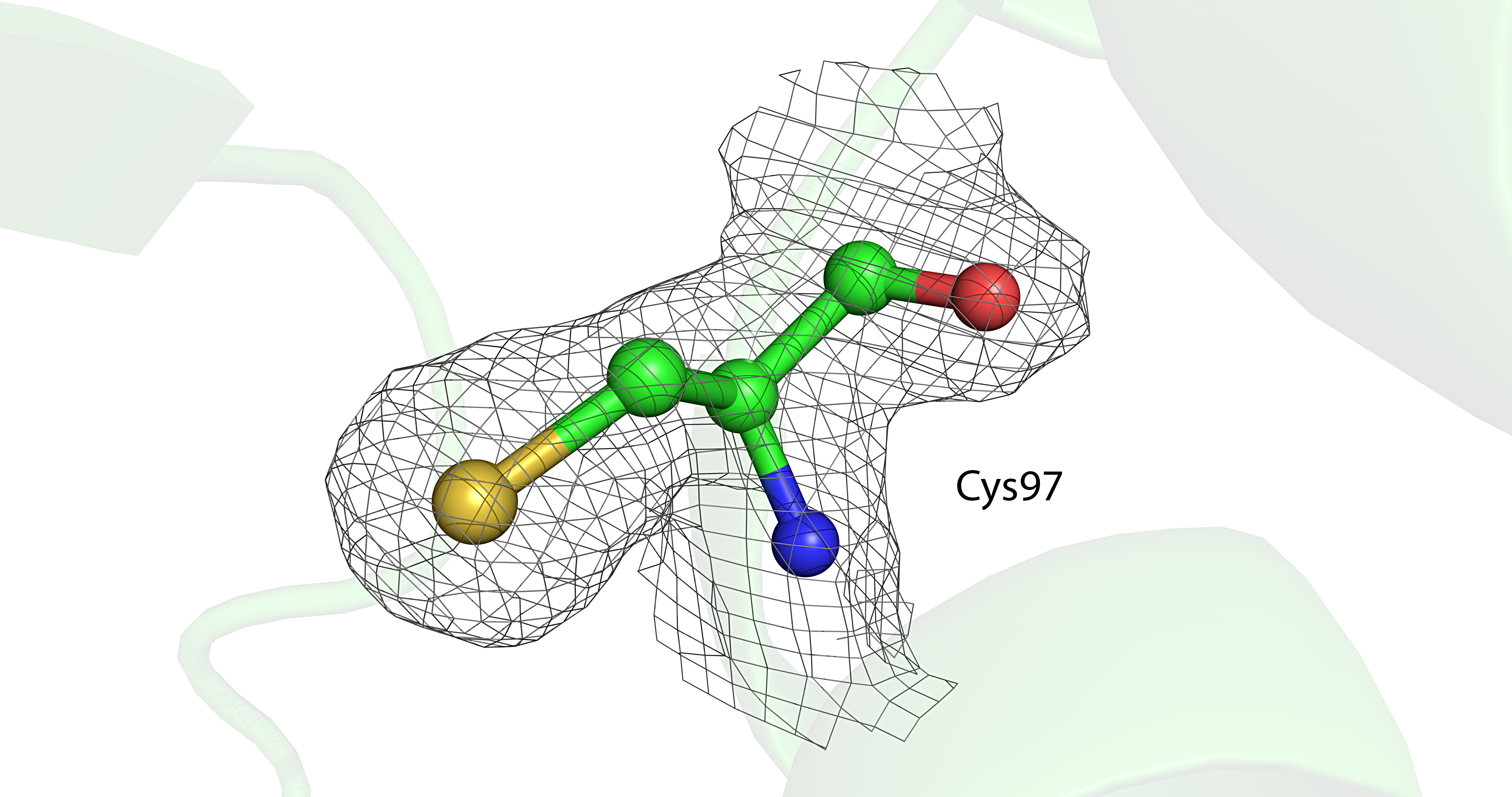
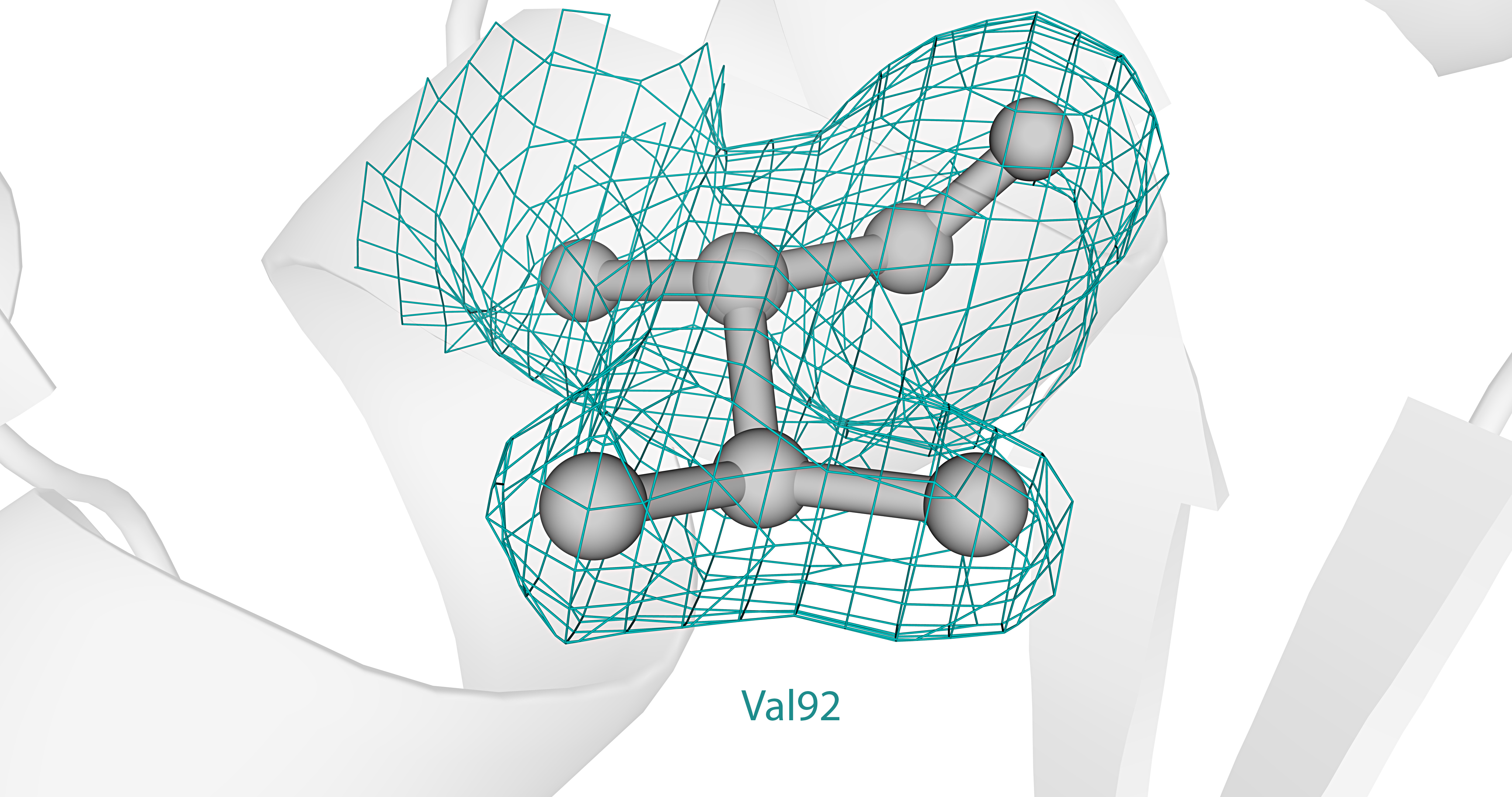
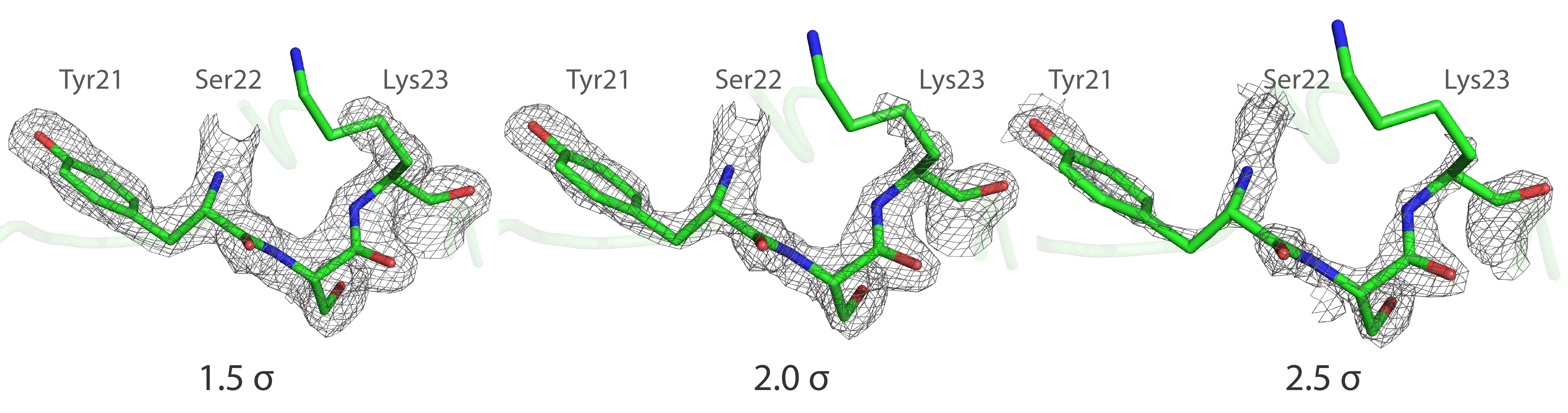
На приведённых изображениях по сгущениям электронной плотности видно положение боковых групп аминокислот, однако наблюдаются и случаи несоответствия. Так, по сгущению электронной плотности нельзя определить положение атомов радикала лизина Lys23 (в частности, атомов Cε и ε-N). Различные уровни электронной плотности (1.5 σ, 2.0 σ и 2.5 σ) для этого участка структуры показаны на следующем изображении. ↓
Фрагмент кода, с помощью которого были получены изображения электронной плотности вокруг аминокислотных остатков:
select proline, resi 85 and chain B show spheres, proline set sphere_scale, 0.2, proline isomesh map, 1uwz_map, 1.0, proline, carve=2.5 color gray30, map set mesh_width, 0.5
На основании визуализации электронной плотности вокруг полипептидной цепи и отдельных аминокислотных остатков структуры белка можно заключить следующее: разрешение структуры 1UWZ не является достаточным для того, чтобы наблюдать отдельные атомы на изображении карты электронной плотности, однако по этой карте виден ход полипептидной цепи и уже угадывается положение боковых групп аминокислот.