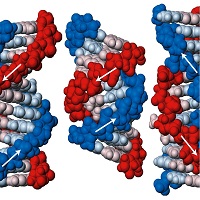
Сравнение структур 3-х форм ДНК с помощью средств JMol
Работа, описанная на этой странице, заключается в сравнительном анализе
канонической ДНК и стеблей тРНК. Для этого были построены модели структур
A-, B- и Z-формы ДНК с помощью инструментов пакета 3DNA. Пакет 3DNA —
один из популярных пакетов программ для анализа и простейшего моделирования
структур нуклеиновых кислот, который работает под операционной системой Linux.
С помощью программы fiber пакета 3DNA были построены A-, B- и Z-формы дуплекса
ДНК, последовательность одной из нитей которого представляет собой 5 раз
повторенную последовательность "gatc". Полученные структуры были проанализированы
с помощью средств JMol. На рис. 1 представлено изображение трех форм ДНК, полученных
в JMol.
Скачать полученные структуры в формате PDB:
А-ДНК,
B-ДНК,
Z-ДНК.
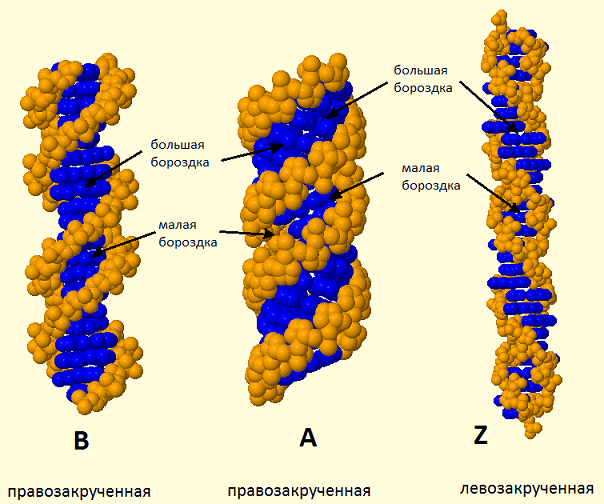 Рис. 1.Разные формы ДНК (JMol)
Большие и малые бороздки.
Рис. 1.Разные формы ДНК (JMol)
Большие и малые бороздки. Файлы со структурами разных форм ДНК были открыты
в JMol. Затем в каждой из структур был выбран цитозин. Сравнительная характеристика
атомов выбранных оснований и основные спиральные параметры разных форм ДНК приведены
в табл.1 и табл.2 соответственно:
Таблица 1. Сравнение цитозина 3-х форм ДНК
| |
A-форма |
B-форма |
Z-форма |
| В сторону большой бороздки обращены атомы: |
C24:B.C2, C24:B.O2, C24:B.N1 |
C24:B.C4, C24:B.N4, C24:B.C5, C24:B.C6 |
C24:A.C4, C24:A.C5, C24:A.C6, C24:A.N4 |
| В сторону малой бороздки обращены атомы: |
C24:B.C5, C24:B.C4, C24:B.N4 |
C24:B.C2, C24:B.O2, C24:B.N1 |
C24:A.C2, C24:A.O2 |
| Остальные атомы основания |
C24:B.C6, C24:B.N3 |
C24:B.N3 |
C24:A.N3 |
Таблица 2. Основные спиральные параметры разных форм ДНК
| |
A-форма |
B-форма |
Z-форма |
| Тип спирали |
правая |
правая |
левая |
| Шаг спирали (Å) |
28,03 |
33,75 |
43,5 |
| Число оснований на виток |
11 |
10 |
12 |
| Ширина большой бороздки (Å) |
16,81 |
17,21 |
16,08 |
| Ширина малой бороздки (Å) |
7,98 |
11,69 |
9,87 |
Измерение торсионных углов. Торсионный угол - угол между двумя связями,
расположенными в разных плоскостях (т.е. это двугранный угол). Для разных форм
ДНК были измерены торсионные углы Cys24. На рис.2 приведены атомы, которые обычно
используются для измерения торсионных углов, а на рис.3 показаны различные торсионные
углы. Полученные результаты измерений каждого из них с помощью JMol приведены в таблице 3.
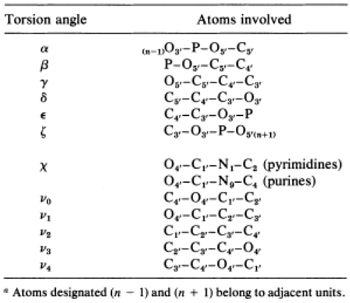 Рис. 2. Торсионные углы в нуклеотидах Рис. 2. Торсионные углы в нуклеотидах |
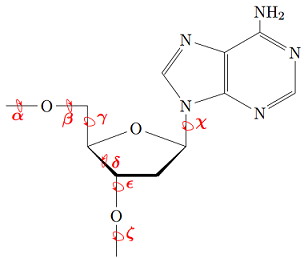 Рис. 3. Торсионные углы в нуклеотидах Рис. 3. Торсионные углы в нуклеотидах |
Таблица 3. Торсионные углы разных форм ДНК в градусах
(в скобках указаны теоретические значения)
| |
α |
β |
γ |
δ |
ε |
ζ |
χ |
| A-форма |
-51,7 (62) |
174,8 (173) |
41,7 (52) |
79,1 (88/3) |
-147,8 (178) |
-75,1 (-50) |
-157,2 (-160) |
| B-форма |
-29,9 (63) |
136,3 (173) |
31,2 (54) |
143,3 (123/131) |
-140,8 (155) |
-160,5 (-90) |
-98,0 (-117) |
Определение параметров структур нуклеиновых кислот с помощью программ пакета 3DNA.
Пакет 3DNA — один из популярных пакетов программ для анализа и простейшего
моделирования структур нуклеиновых кислот. Для анализа структур нуклеиновых кислот
были использованы программы find_pair и analyze. Программа find_pair определяет
спаренные основания и положения спиралей в структуре. Полученные данные необходимы
для работы analyze. В результате были получены файлы для каждой структуры, в которых
можно найти описание водородных связей, значения всех торсионных углов, ширину малой
и большой бороздки и т.д. Для измерений использовалась заданна тРНК (PDB ID:2dxi).
Полученные результаты приведены в таблице 3.
Скачать структуры ДНК и тРНК в формате PDB:
ДНК,
тРНК.
Скачать описания структур:
А-ДНК,
B-ДНК,
Z-ДНК
ДНК,
тРНК.
Таблица 4. Торсионные углы разных форм ДНК и тРНК в градусах
(в скобках указаны выбранные нуклеотиды)
| |
α |
β |
γ |
δ |
ε |
ζ |
χ |
| A-форма (C24:B) |
-51,7 |
174,8 |
41,7 |
79,1 |
-147,8 |
-75,1 |
-157,2 |
| B-форма (C24:B) |
-29,9 |
136,3 |
31,2 |
143,3 |
-140,8 |
-160,5 |
-98,0 |
| Z-форма (C24:A) |
-139,5 |
-136,8 |
50,9 |
137,6 |
-96,5 |
82,0 |
-154,3 |
| тРНК (C24:A) |
-61,0 |
-175,7 |
42,6 |
81,2 |
-154,4 |
-61,5 |
-156,0 |
Если попробовать сравнить полученные с помощью пакета 3DNA значения со значениями
углов, измеренных с помощью JMol, можно заметить, что величины всех углов
совпадают, что логично.При этом значительные отличия измерений для некоторых
углов (например, для угла α) от теоретических значений
можно объяснить тем, что углы, скорее всего, измерялись для разных атомов. Небольшие
же отличия, скорее всего, объясняются обычным варьированием величины углов от
нуклеотида к нуклеотиду (точно такое же можно встретить среди значений одного
угла для разных нуклеотидов, к примеру, ДНК).
Согласно полученным данным торсионные углы для выбранного нуклеотида тРНК больше
всего похожи на торсионные углы в А-ДНК (не считая угла β, который очень
сильно отличается). При этом хочется отметить, что для ДНК значения торсионных
углов практически не менялись вне зависимости от выбранного нуклеотида, в то время
как для тРНК значения варьировались как для разных типов нуклеотидов (А, Т, С, G),
так и для нуклеотидов одного типа, отличающихся расположением в цепи (например, A23
и A10). Для сравнения параметров в тРНК был выбран С24, хотя нужно учитывать, что опять же
значения торсионных углов для нуклеотидов тРНК отличались (и иногда довольно
сильно).
В Excel были проанализированы данные, полученные с помощью пакета 3DNA для
заданной ДНК (PDB ID:1by4) и тРНК. В обоих случаях рассматривались цепи В.
В таблице 5 приведены вычисленные средние значения
торсионных углов для ДНК и тРНК. Самыми "деформированными" нуклеотидами
(с наиболее отклоняющимися значениями нескольких углов) можно назвать
A13, G14, C27, C29, A41, G42, C55 для тРНК и C3, T8, C10, C11, A18,
C26, G28 для ДНК (все это можно увидеть, открыв файл Excel). Из-за
сильно выбивающихся значений углов для этих нуклеотидов средняя величина
значительно отличается от измеренной в предыдущем задании. Примечательно,
что в случае тРНК эти нуклеотиды явно не случайны, т.к. заметна закономерность,
что нельзя сказать о ДНК.
Файл Excel с расчетами:
скачать.
Таблица 5. Средние значения торсионных углов в нуклеотидах
ДНК и тРНК 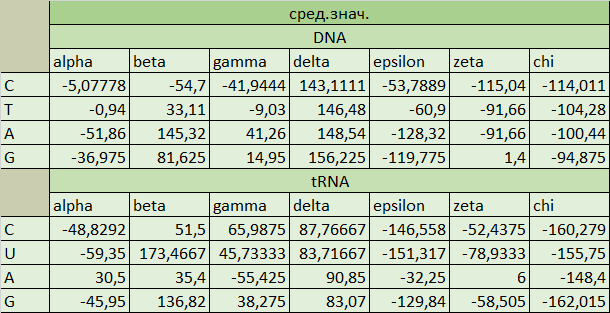
С помощью программы find_pair для тРНК (find_pair -t 2dxi.pdb stdout > tRNA_pairs)
были найдены нуклеотиды, образующие водородные связи. В таблице 6 приведены разные
полученные таким образом данные. Нуклеотиды стеблей (stems) выделены цветом,
так что цвет характеризует их положение на рис.4. Дополнительными водородными
связями, стабилизирующими третичную структуру тРНК, считались связи, не имеющие
отношения к стеблям).
Файл с расчетами:
открыть.
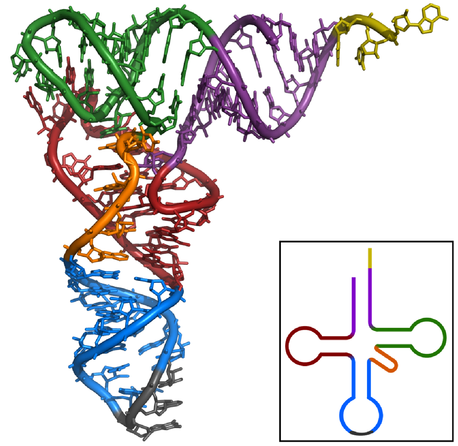 Рис. 4. тРНК
Рис. 4. тРНК
Таблица 6. Участие различных нуклеотидов тРНК в образовании
водородных связей (рассмотрена только цепь C, для цепи D все аналогично)
| |
Стебли тРНК |
Неканонические пары |
Дополнительные
водородные
связи |
Нуклеотиды, образующие
водородные связи |
C:501[G]---[C]572:C
C:502[G]---[U]571:C
C:503[C]---[G]570:C
C:504[C]---[G]569:C
C:505[C]---[G]568:C
C:506[C]---[G]567:C
C:507[A]---[U]566:C
________________
C:549[G]---[C]565:C
C:550[G]---[C]564:C
C:551[G]---[C]563:C
C:552[G]---[C]562:C
C:553[G]---[C]561:C
________________
C:538[A]---[C]532:C
C:539[G]---[C]531:C
C:540[G]---[C]530:C
C:541[C]---[G]529:C
C:542[C]---[G]528:C
C:543[G]---[C]527:C
C:544[A]---[G]526:C
________________
C:510[G]---[C]525:C
C:511[U]---[A]524:C
C:512[C]---[G]523:C
C:513[U]---[G]522:C
|
C:502[G]---[U]571:C
C:538[A]---[C]532:C
C:544[A]---[G]526:C
C:513[U]---[G]522:C
C:555[U]---[G]518:C
|
C:514[A]---[U]508:C
C:515[G]---[C]548:C
C:519[G]---[C]556:C
C:554[U]---[A]558:C
C:555[U]---[G]518:C
|
Нахождение возможных стекинг-взаимодействий. После запуска программы analyze
в полученном файле
staking.pdb
записаны преобразованные координаты всех динуклеотидных пар, которые можно
использовать для получения общепринятого изображения стекинг-взаимодействий.
В файле staking.pdb были найдены данные о величине площади "перекрывания"
2-х последовательных пар азотистых оснований. Для пар с наибольшими (GC/GU)
и наименьшими (AG/CG) значениями были получены стандартные изображения
стекинг-взаимодействий. Для этого для них сначала были найдены номера структур (Section#000n - это структура №n),
описывающих координаты искомых нуклеотидных остатков. Затем эти структуры
были вырезаны каждая в отдельный файл с помощью команды "ex_str -n stacking.pdb stepn.pdb".
Потом для каждой из них было создано изображение с помощью команды
"stack2img -cdolt stepn.pdb stepn.ps". Получившиеся ps-файлы были открыты ассоциированной
программой Ghost и конвертированы в формат JPEG. На рис. 5 представлены полученные
изображения.
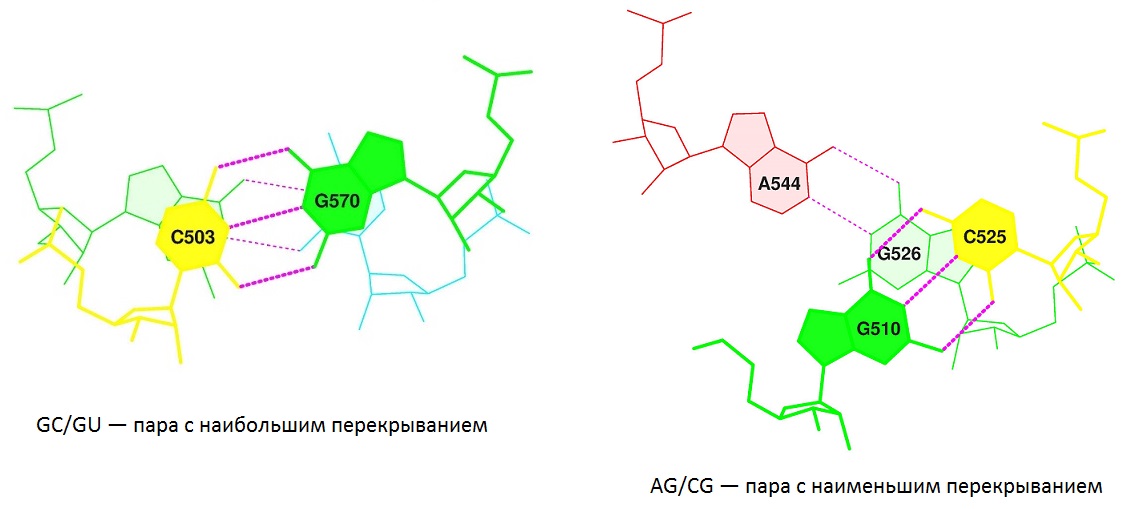 Рис. 5. Стекинг взаимодействия между нуклеотидами
Рис. 5. Стекинг взаимодействия между нуклеотидами