EMBOSS: пакет программ для анализа последовательностей
1. С помощью команды
seqret необходимо было несколько файлов в
формате
fasta собрать в единый файл. Для этого был создан список
mylist.txt, содержащий адреса исходных последовательностей в формате usa.
Далее с помощью команды:
seqret @mylist.txt mysequences.fasta
был получен файл
mysequences.fasta, который
содержит уже сами последовательности.
2. С помощью команды
seqretsplit нужно было один файл в формате
fasta с несколькими последовательностями разделить на отдельные fasta файлы. В
качестве исходного файла использовался файл
mysequences.fasta, полученный в прошлом пункте. С помощью команды:
seqretsplit mysequences.fasta -auto
были получены отдельные файлы с последовательностями:
1a1l1_takru.fasta,
1a34_human.fasta,
060l_iiv6.fasta,
081r_frg3g.fasta и
1433b_xentr.fasta.
3. С помощью команды
seqret необходимо было из файла с хромосомой
в формате .gb три кодирующих последовательности по указанным координатам "от",
"до", "ориентация" и сохранить в одном fasta файле. Использовался файл
sequence.gb, содержащий хромосому I
Encephalitozoon
romaleae. Далее был создан список
list.txt, в
котором содержит координаты кодирующих последовательностей. С помощью команды:
seqret @list.txt seqs.fasta
были вырезаны соответствующие кодирующие последовательности по указанным
координатам, каждая записана в файл:
seqs.fasta.
4. С помощью команды
transeq нужно было транслировать кодирующие
последовательности, лежащие в одном fasta-файле, в аминокислотные, используя
указанную таблицу генетического кода, при этом результат — в одном
fasta-файле. На вход команде подавался файл
seqs.fasta,
полученный в предыдущем пункте:
transeq seqs.fasta tran_seqs.fasta
Таким образом был получен файл
tran_seqs.fasta,
содержащий трансляции исходных последовательностей.
5. С помощью той же команды нужно было транслировать данную нуклеотидную
последовательность в шести рамках. На вход команде подавалась последовательность
gene.fasta:
transeq gene.fasta tran_gene.fasta -frame 6
В итоге был получен файл
tran_gene.fasta,
содержащий шесть трансляций данной нуклеотидной последовательности.
Ссылка на остальные задания (для подготовки к коллоквиуму):
tasks.html.
Построение карты локального сходства двух геномов
В этом задании необходимо было построить карту локального
сходства двух бактерий и описать крупные эволюционные события на пути от общего
предка.
Рис. 1. Электронная фотография
Desulfurococcus kamchatkensis 1221n1 |
Для выполнения задания были выбраны две археи из одного рода:
Desulfurococcus
kamchatkensis 1221n и
Desulfurococcus fermentans DSM 16532.
Систематика:
>Archea
>Crenarchaeota
>Thermoprotei
>Desulfurococcales
>Desulfurococcaceae
>
Desulfurococcus
Геномы:
1)
Desulfurococcus fermentans
DSM 16532, размер генома: 1384116 п.н.
2)
Desulfurococcus kamchatkensis 1221n, размер генома: 1365223 п.н.
Desulfurococcus — род термофильных анаэробных архей, найденных в
зонах с высокой температурой среды, таких как глубоководные гидротермальные
и подземные горячие источники. Оптимальная температура роста — 85ºC.
Клетки сферические, окружены необычной белковой оболочкой, напоминающей решетку.
Есть один длинный жгутик
2.
Для построения карты локального сходства использовался
blast2seq (
алгоритм blastn) на сайте NCBI.
Характеристики полученного выравнивания (
alignment.txt):
e-value = 0.0
identity = 97%
query cover = 91%
total score = 2.069e+06
На рис. 2 представлена полученная карта.
По оси абсцисс в ней располагается геном Desulfurococcus kamchatkensis
(для удобства будем обозначать как "геном А"),
а по оси ординат — геном Desulfurococcus fermentans (как
"геном В").
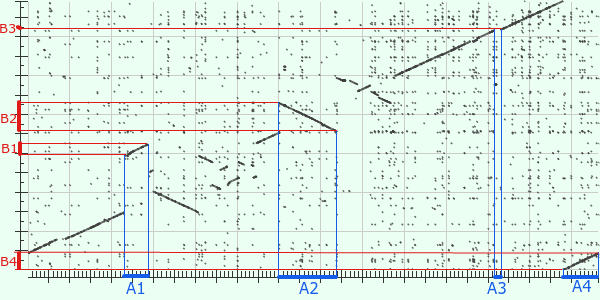
Рис. 2. Карта локального сходства исследуемых
последовательностей. |
Среди всех событий было выбрано четыре примера; синтеничные участки обозначены
буквами с одинаковыми цифрами. Разберем эти примеры по пунктам:
- Участок А1 в геноме В распологается дальше, чем в геноме А, т.е.
имеет смысл говорить о транслокации участка в одном из геномов.
- Видно, что участок В2 — это перевернутый на 180°, т.е.
в одном из геномов произошла инверсия этого участка.
- Участок А3 отсутствует в геноме В (на графике спроецирован в "точку"
В3; понятно, что это условное обозначение). Это либо результат вставки
в геноме А, либо делеции в геноме В.
- И, наконец, участки А4 и В4. Учитывая тот факт, что у рассматриваемых
архей ДНК кольцевые, можно предположить, что это вовсе не результат
делеции/вставки, а просто по-разному установленные координаты начала
последовательностей.
Все остальные случаи можно объяснить, как один из приведенных примеров.
Таким образом, гомологичные участки занимают большую часть геномов.
Построение нуклеотидного пангенома для 3 геномов близкородственных
бактерий.
Для выполнения задания было выбрано 3 штамма бактерии Streptococcus pneumoniae
(AP200, ATCC 700669, CGSP14).
Рис. 3. S. pneumoniae3 |
Систематика:
>Bacteria
>Firmicutes
>Bacilli
>Lactobacillales
>Streptococcaceae
>Streptococcus
>S. pneumoniae
Пневмококк — грамположительная шаровидная бактерия, факультативный анаэроб.
Пневмококки являются одним из основных возбудителей менингита, среднего отита, синусита,
внебольничной пневмонии у детей и взрослых. В последние годы проблема резистентности
пневмококков к антимикробным препаратам обсуждается во многих странах4.
Далее идет общая информация о программе и пангеномах, приведенная здесь для того,
чтобы мне было куда обратиться в будущем. Если вам это не нужно, переходите к п.1.
Пангеном может быть построен на основе ортологичных генов или ортологичных участков
генома (нуклеотидный пангеном). Для построения пангенома NPGE использует данные о
гомологичных участках, а не генах.
Один блок может включать ортологи и паралоги.
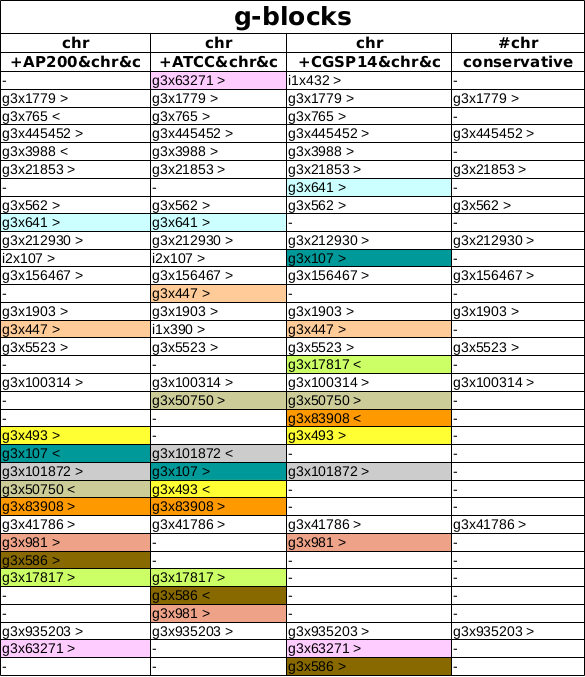
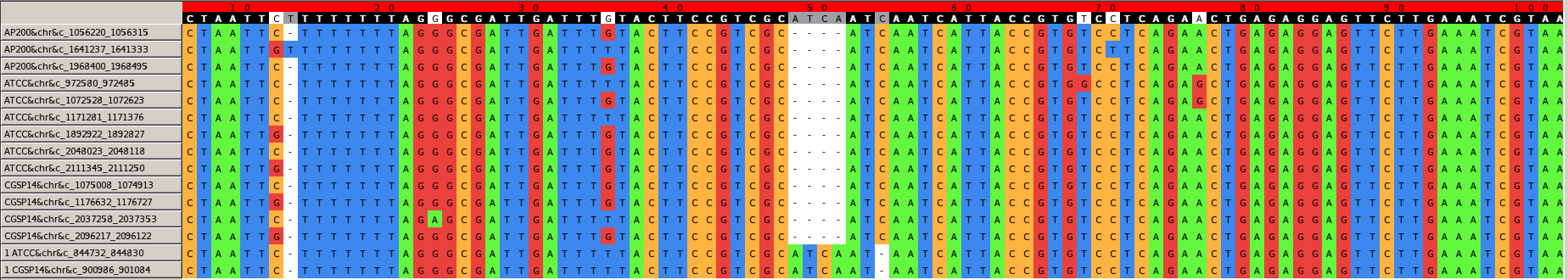
Обозначения блоков:
Ссылки:
[1] I. V. Kublanov, S. Kh. Bidjieva, A. V. Mardanov and E. A. Bonch-Osmolovskaya
(2009).
Desulfurococcus kamchatkensis sp. nov., a novel hyperthermophilic
protein-degrading archaeon isolated from a Kamchatka hot spring.
Int J Syst
Evol Microbiol. 59:1743-7.
[2]
Desulfurococcus // MicrobeWiki.
[URL]
[3]
Streptococcus pneumoniae // The kingdom of life.
[URL]
[4]
Пневмококк // Wikipedia: the free encyclopedia.
[URL]
[5] How to read a phylogenetic tree // epidemic: Molecular Epidemiology and
Evolution of Viral Pathogens.
[URL]
[6] Инсерционная последовательность // Wikipedia: the free encyclopedia.
[URL]
[7] ABC-Белки (ABC-транспортеры, суперсемейство транспортных белков ABC) // medbiol.ru.
[URL]
[8] Защитная функция белков // Wikipedia: the free encyclopedia.
[URL]