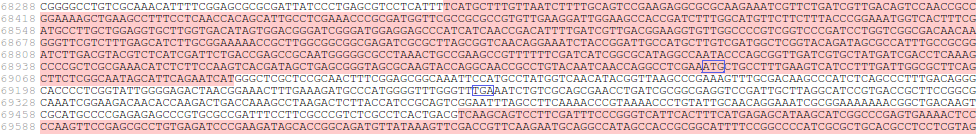Предсказание генов прокариот
1. Сравнение предсказаний генов в базе данных GenBank и по данным Prodigal для плазмиды
 Рис. 1.
Фотографии колонии Shinella sp.: SEM (слева), агар (в
центре и справа)1 Рис. 1.
Фотографии колонии Shinella sp.: SEM (слева), агар (в
центре и справа)1 |
Для выполнения задания мне была дана плазмида
Shinella sp. HZN7.
Систематика:
Домен: Bacteria
Тип: Proteobacteria
Класс: Alphaproteobacteria
Порядок: Rhizobiales
Семейство: Rhizobiaceae
Род: Shinella
Shinella sp. — плохо изученная аэробная, грам-отрицательная
бактерия, не образующая спор. Род открыт в 2006 году.
Штамм DD12, например, был выделен из зоопланктона
Daphnia magna.
Особенностью рода является способность использовать фосфиты
в качестве единственного источника фосфора для поддержания своего роста.
В настоящий момент род включает в себя 6 видов:
S. zoogloeoides , S. granuli , S. fusca , S. kummerowiae , S. daejeonensis,
S. yambaruensis1.
Для выполнения задания мне была дана кольцевая плазмида
pShin-12 длиной в 71917 п.н.
Сначала с помощью команд:
seqret embl:CP015748 fasta::cp015748.fasta
seqret embl:CP015748 gff::cp015748.gff -feature
была скачана последовательность выданной мне плазмиды в двух форматах:
cp015748.fasta и
cp015748.gff. При этом в итоговом файле
.gff были сохранены
особенности (features) с помощью квалификатора -features, т.к. они понадобятся
для дальнейшей работы.
Затем с помощью
Prodigal были предсказаны гены в данной плазмиде.
prodigal -i cp015748.fasta -o prodigal.fasta -f sco
При этом был выбран (-f sco) минималистичный формат
sco для записи
результата:
prodigal.fasta.
Из обоих файлов координаты генов (начало, конец, ориентация, разделенные символом
'_') были записаны в
отдельные файлы с помощью команд:
grep CDS cp015748.gff | cut -f 4,5,7 --output-delimiter='_'> genbank.out
grep '>' prodigal.fasta | cut -f 2,3,4 -d '_' > prodigal.out
Полученные файлы с координатами:
prodigal.out,
genbank.out.
Далее был создан скрипт
pr10.py, с помощью которого были
оценены количественно следующие показатели:
- число и % генов, которые аннотированы в GenBank и для которых
предсказание обоих концов гена с помощью Prodigal точно такое
же;
- число и % генов, для которых аннотация только N-конца белка не
совпадает с аннотацией Prodigal;
- число и % генов, для которых аннотация только С-конца белка не
совпадает с аннотацией Prodigal;
- число и % генов, для которых аннотация обоих концов не совпадает с
аннотацией Prodigal.
Прим. Процент считался от общего числа предсказанных
Prodigal
генов, т.к. их было больше аннотированных в
GenBank'е генов.
Краткое описание алгоритма работы скрипта:
- Скрипт запрашивает на вход названия трех файлов (GenBank и Prodigal с
координатами и файл, в который будут записаны результаты) и
создает два списка genbank и prodigal, содержащих строки
с координатами из исходных файлов (координаты разделены символом '_').
- Затем создаются по два списка с координатами С- и N-концов для
каждого из списков в п.1. Для этого в цикле из каждой строки создается
список (разделитель '_') и проверяется ориентация гена ('+' или
'-' как последний элемент полученного из строки списка). В зависимости
от этого в качестве координаты N-конца (С-конца) в случае '+' берется
первая (вторая) координата, а в случае '-' — вторая (первая).
В итоге получаем четыре списка (*_genbank, *_prodigal,
*=c/n) с координатами концов.
- После этого для каждой координаты N-конца в n_genbank ищется
такая же координата в n_prodigal. Если она найдена, проверяется
совпадает ли координата C-конца и в зависимости от ответа подсчитывается
число совпадающих генов (если координаты совпадают) или генов с разными
C-концами (у белка). Далее для каждой координаты С-конца в
c_genbank ищется
такая же в c_prodigal, так что соответствующие координаты N-концов
не совпадают. Таким образом подсчитывается число генов с по-разному
предсказанными N-концами белков.
- Все результаты записываются в указанный файл (в нашем случае это был
файл results.out) для удобства.
На рис. 2 и в табл. 1 представлены полученные результаты.
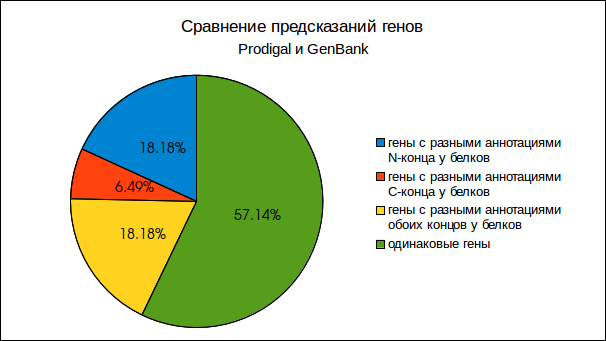 Рис. 2.
Диаграмма, отображающая результаты анализа Рис. 2.
Диаграмма, отображающая результаты анализа |
Таблица 1. Полученные результаты сравнения предсказаний
генов
| Число генов с одинаково предсказанными концами |
44 (57,14%) |
| Число генов с по-разному предсказанными C-концами |
5 (6,49%) |
| Число генов с по-разному предсказанными N-концами |
14 (18,18%) |
| Число предсказанных Prodigal генов, не аннотированных в
Genbank
|
14 (18,18%) |
| Число не предсказанных Prodigal генов, аннотированных в
Genbank |
1 |
| Общее число предсказанных Prodigal генов |
77 |
| Общее число аннотированных в GenBank генов |
62 |
Прим. Общее число хотя бы частично верно предсказанных генов
(первые три строчки в таблице) — 63 + один непредсказанный ген = 64. При
этом в GenBank аннотированных генов 62. Т.к. ошибки в скрипте я не нашла,
я объясняю это наличием в предсказании Prodigal нескольких генов с
совпадающим C-концом и N-концом (белка) для одного и того же гена в Genbank.
В файле results.out можно найти примеры.
Таким образом,
Prodigal для 44 генов предсказал координаты концов так же, как
они аннотированы в
GenBank (57,14%). Как мне кажется, это довольно низкий
процент. Тем не менее, только один ген, аннотированный в
GenBank, не был
найден этой программой, хоть и для многих из них (19) концы были предсказаны
по-другому.
Попробуем понять, почему для некоторых генов концы были предсказаны по-другому.
Пример 1. Не найденный
Prodigal ген, аннотированный в
Genbank.
Ген имеет координаты 68344-69094 (-). На рис. 3 данный ген выделен розовым. Синими
квадратами обозначены старт-кодон (ATG) и стоп-кодон (TGA) гена, который предсказал
Prodigal.
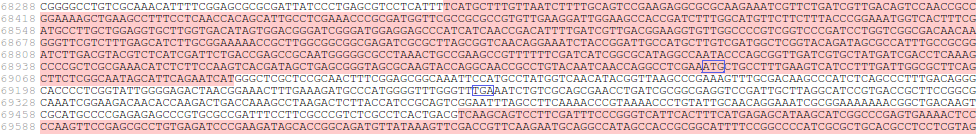 Рис. 3.
Анализируемый ген (верхний выделенный розовым) Рис. 3.
Анализируемый ген (верхний выделенный розовым) |
В записи
Genbank в строке "product" в аннотации данного гена указана
"transposase", которая уже обсуждалась в предыдущем практикуме. При этом в "note"
указано: "... Derived by automated
computational analysis using gene prediction method: Protein Homology" (рис. 5).
Prodigal, в свою очередь, на этом участке предсказал только один ген с
координатами 69030-69260(+), который короче аннотированного в
Genbank.
В общем, я больше склоняюсь к правильной аннотации данного гена в записи
Genbank.
Для того, чтобы это проверить, был запущен BLAST с этим
аннотированным
геном (discontiguous megablast). В итоге
было найдено 95 находок со статистически значимым e-value. Результаты поиска можно
посмотреть:
alignment1.txt. Для нескольких лучших
находок в качестве "product" были указаны: transposase, mobile element, hypothetical
protein. При этом для
"гена", найденного
Prodigal
, было найдено 55 находок (результаты поиска:
alignment2.txt), однако в соответствующих записях они не были никак
аннотированы. Таким образом, можно заключить, что
Prodigal тут все-таки
ошибся.
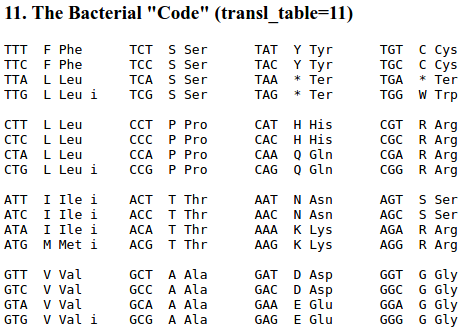 Рис. 4.
Таблица трансляции №112 Рис. 4.
Таблица трансляции №112
|
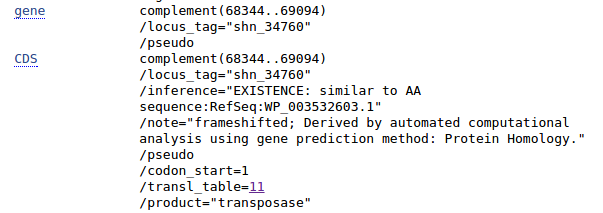 Рис. 5.
Аннотация гена в GenBank Рис. 5.
Аннотация гена в GenBank
|
При машинном анализе использовалась таблица 11, приведенная на рис. 4. В ней
старт-кодоны
обозначены прописной буквой
i, а стоп-кодоны — '* Ter'. Эта таблица
используется для анализа последовательностей бактерий, архей,
вирусов прокариот и хлоропластных белков. В ней помимо стандартного старт-кодона
ATG добавлены другие, встречающиеся у архей и бактерий
3.
Пример 2. В качестве второго примера расхождения предсказаний рассмотрим
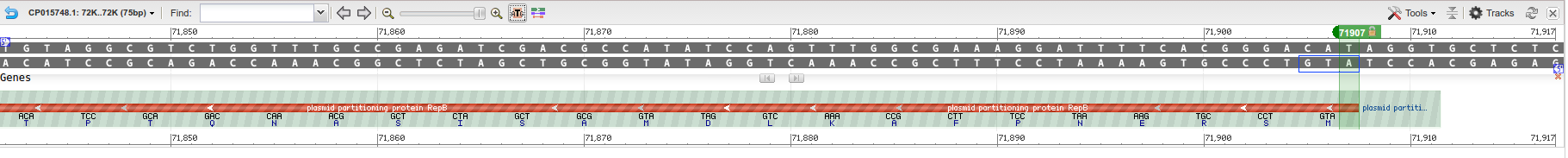
ген с координатами 70911-71907(-) в
Genbank и 70911-71402(-), предсказанными
Prodigal (т.е. программа определила N-конец по-другому).
На рис. 6, 7 показан ген в геномном браузере.
На рис. 6 также показано расположение гена в последовательности плазмиды. Как видно
на обоих рисунках, и в том, и в другом случае в качестве старт-кодона был взят ATG.
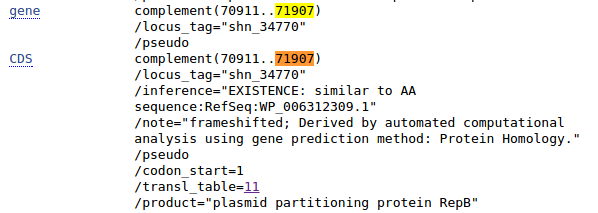 Рис. 8.
Аннотация обсуждаемого гена в записи GenBank Рис. 8.
Аннотация обсуждаемого гена в записи GenBank
|
На рис.8 приведена аннотация гена в
Genbank. В данном случае в качестве
продукта указан "plasmid partitioning protein RepB". Способ предсказания тот же
самый.
Plasmid partition proteins — белки, составляющие систему
разделения у плазмид RepABC, обеспечивающую их стабильность и репликацию. В систему
входят белки RepA, RepB, и центромеро-подобный белок parS. Мутации в этих белках
могут сильно уменьшить стабильность плазмид
4.
Как мне кажется, в этом случае стоит верить
Genbank'у, т.к.
описанные белки должны быть довольно консервативными. Для того, чтобы это проверить,
для аннотированного
гена тоже был запущен BLAST. В итоге
было найдено 13 сходных последовательностей, из которых для 10 покрытие составляет
99-100% (
результаты). Находки аннотированы также. Что
касается
гена, найденного
Prodigal, то для
него
результаты поиска BLAST выдали те же находки,
но с немного отличающимися параметрами. При этом найденные последовательности
были частью соответствующих аннотированных генов (тоже кодирующих RepB). Таким
образом, тут программа тоже ошиблась, но уже в определении N-конца.
2. Сравнение предсказаний генов в базе данных GenBank и по данным Prodigal для геномной записи
В данном задании необходимо было сравнить те же показатели, что в предыдущем пункте,
для геномов модельного организма
E. coli и археи
T. kodakarensis, с
которой я уже работала. Вручную с сайта NCBI были скачаны упомянутые геномы, для
которых потом были получены файлы с координатами и результаты анализа скриптом (все
аналогично предыдущему пункту):
1)
E. coli:
ecoli.fasta,
ecoli.gff;
координаты:
ecoli_gb.out,
ecoli_prod.out; результат анализа:
ecoli.result;
2)
T. kodakarensis:
thermo.fasta,
thermo.gff;
координаты:
thermo_gb.out,
thermo_prod.out; результат анализа:
thermo.result;
В таблице 2 приведены полученные результаты.
Таблица 2. Полученные результаты сравнения предсказаний
генов для двух заданных геномов
|
E. coli |
T. kodakarensis |
| Число генов с одинаково предсказанными концами |
3831 (88.7%) |
1910 (82.54%) |
| Число генов с по-разному предсказанными C-концами |
75 (1.74%) |
19 (0.82%) |
| Число генов с по-разному предсказанными N-концами |
319 (7.39%) |
325 (14.04%) |
| Число предсказанных Prodigal генов, не аннотированных в
Genbank
| 94 |
60 |
| Число не предсказанных Prodigal генов, аннотированных в
Genbank |
340 |
33 |
| Общее число предсказанных Prodigal генов |
4319 |
2314 |
| Общее число аннотированных в GenBank генов |
4386 |
2268 |
Как видно из таблицы, предсказания, в принципе, довольно близки. Для
E. coli,
однако, процент верно найденных генов выше, как и процент неверно
предсказанных C-концов и число непредсказанных генов, которые аннотированы в
GenBank. Последнее меня немного удивило, т.к. это модельный организм и
Prodigal, по идее, должен хорошо находить для его генома гены. Возможно, это
связано с большим размером генома у
E. coli (больше более чем в 2р.).