Филогенетические деревья
Общая информация о выбранных организмах
Таблица 1. Выбранные организмы
| Название |
Мнемоника |
| Bacillus anthracis |
BACAN |
| Clostridium botulinum |
CLOBA |
| Clostridium tetani |
CLOTE |
| Geobacillus kaustophilus |
GEOKA |
| Lactobacillus delbrueckii subsp. bulgaricus |
LACDA |
| Staphylococcus aureus |
STAAR |
| Streptococcus pneumoniae serotype 4 |
STRPN |
Филогенетическое дерево отражает эволюционные взаимосвязи между различными видами,
имеющими общего предка. Филогенетические деревья, как правило, реконструируются по
последовательностям белков или нуклеиновых кислот.
В таблице 1 перечислены выбранные бактерии, для которых было построено
филогенетическое дерево.
Все семь бактерий относятся к
фирмикутам (лат. Firmicutes) — типу бактерий,
представители которого характеризуются низким содержанием пар нуклеотидов G+C
(менее 50%) и строением клеточной стенки, характерным для грамположительных бактерий.
Представители данного типа являются как патогенами (
Bacillus anthracis,
Clostridium botulinum, Clostridium tetani, Staphylococcus aureus, Streptococcus pneumoniae
serotype 4), так и представителями нормальной микробиоты человека (
Lactobacillus
delbrueckii subsp. bulgaricus)
1.
Также было показано, что фирмикуты в кишечнике рыб
Danio rerio способствовали лучшему
усвоению жирных кислот
2, а у мышей количество этих бактерий было пропорционально степени
ожирения
3. Так как у упомянутых рыб метаболизм липидов очень похож на метаболизм
липидов у млекопитающих и других позвоночных, изучение фирмикут может быть важно для лучшего
понимания причин ожирения у людей.
1. Построение филогенетического дерева
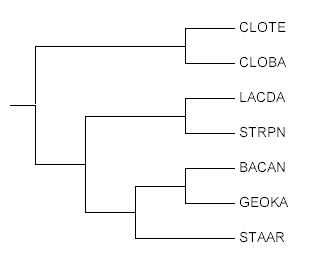 Рис. 1. Построенное филогенетическое
дерево Рис. 1. Построенное филогенетическое
дерево |
Для построения дерева использовалась программа
MEGA,
позволяющая построить дерево по скобочной формуле (в формате Newick). Дерево строилось на основе
дерева, данного в задании. Для построения был создан файл
.tre, содержащий скобочную формулу:
((CLOBA, CLOTE), ((LACDA, STRPN), ((BACAN, GEOKA), STAAR)));
Дерево на рис. 1 отражает эволюцию семи упомянутых бактерий.
Полученное дерево содержит четыре нетривиальные ветви (т.е. ветви, разбивающие множество листьев на
подмножества, в каждом из которых более одного элемента):
- {CLOBA, CLOTE} vs {LACDA, STRPN, BACAN, GEOKA, STAAR};
- {LACDA, STRPN} vs {CLOBA, CLOTE, BACAN, GEOKA, STAAR};
- {BACAN, GEOKA, STAAR} vs {CLOBA, CLOTE, LACDA, STRPN};
- {BACAN, GEOKA} vs {STAAR, CLOBA, CLOTE, LACDA, STRPN}.
2. Таксономия выбранных организмов
С использованием таксономического сервиса NCBI
http://www.ncbi.nlm.nih.gov/taxonomy/
были определены таксоны, к которым относятся выбранные бактерии. Они
приведены в таблице 2, начиная с таксона, лежащего ниже типа Firmicutes.
Таблица 2. Таксономия выбранных бактерий
| Название |
Таксономия |
| Bacillus anthracis |
Bacilli > Bacillales > Bacillaceae > Bacillus > Bacillus cereus group |
| Clostridium botulinum |
Clostridia > Clostridiales > Clostridiaceae > Clostridium |
| Clostridium tetani |
Clostridia > Clostridiales > Clostridiaceae > Clostridium |
| Geobacillus kaustophilus |
Bacilli > Bacillales > Bacillaceae > Geobacillus > Geobacillus thermoleovorans group |
| Lactobacillus delbrueckii subsp. bulgaricus |
Bacilli > Lactobacillales > Lactobacillaceae > Lactobacillus > Lactobacillus delbrueckii |
| Staphylococcus aureus |
Bacilli > Bacillales > Staphylococcaceae > Staphylococcus |
| Streptococcus pneumoniae serotype 4 |
Bacilli > Lactobacillales > Streptococcaceae > Streptococcus |
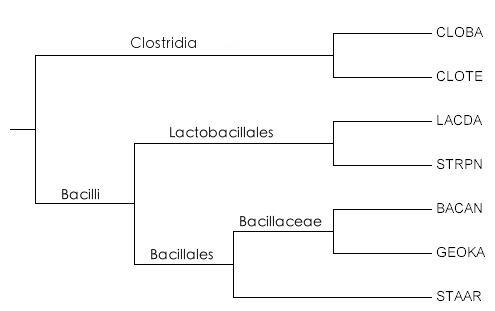 Рис. 2. То же
дерево с указанием таксонов для нетривиальных ветвей Рис. 2. То же
дерево с указанием таксонов для нетривиальных ветвей |
Как видно на рисунке, все нетривиальные ветви отделяют тот или иной таксон от
остальных ветвей. Таким образом ветви в порядке, в которым они указаны в
задании 1, отделяют следующие таксоны:
- ветвь отделяет класс Clostridia от другого класса Bacilli;
- ветвь отделяет отряд Lactobacillales от остальных таксонов;
- ветвь отделяет семейство Bacillaceae от остальных таксонов;
- ветвь отделяет класс Bacilli от другого класса Clostridia,
которые вместе входят в состав типа Firmicutes.
4. Реконструкция филогении
В этом задании я пыталась реконструировать филогенетическое дерево по заданному семейству
белков. В качестве белка была выбрана пептидил-тРНК-гидролаза (мнемоника: PTH). Соответствующие
последовательности были скачаны из БД
UniProt и выравнены с помощью
Jalview (Muscle). Затем по выравниванию было построено дерево методом "Neighbor Joining Using %
Identity". Оно было сохранено в Newick-формате и открыто в
MEGA. Полученное выравнивание в
.fasta:
task_2_alignment.fasta и проект:
task_2.jvp; дерево в Newick-формате:
task_2_tree.tre.
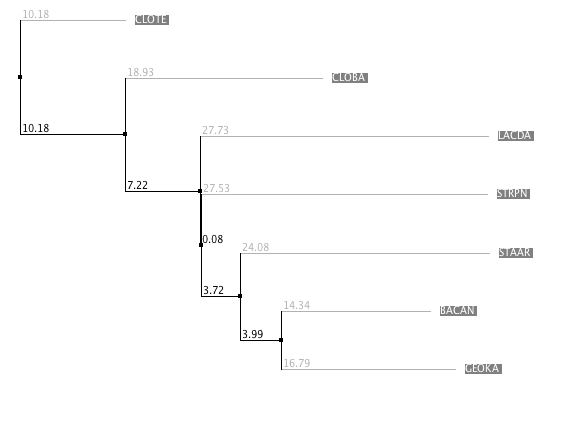 Рис. 2.
Построенное в Jalview дерево Рис. 2.
Построенное в Jalview дерево |
На рис. 3 представлено полученное с помощью
Jalview изображение построенного дерева.
Нетривиальные ветви у полученного дерева следующие:
- {CLOBA, CLOTE} vs {LACDA, STRPN, BACAN, GEOKA, STAAR};
- {CLOBA, CLOTE, LACDA} vs {STRPN, BACAN, GEOKA, STAAR};
- {CLOBA, CLOTE, LACDA, STRPN} vs {BACAN, GEOKA, STAAR};
- {BACAN, GEOKA} vs {STAAR, CLOBA, CLOTE, LACDA, STRPN}
Таким образом, если сравнить топологию с топологией дерева в задании 1, все нетривиальные ветви,
кроме второй, одинаковы. В то же время, вторая ветвь в данном случае делит бактерии на две группы,
поделив при этом отряд Lactobaciliales на две части.
Дополнительные задания представлены на
другой странице.
Ссылки:
[1] Briggs G.S., Smits W.K., Soultanas P. (2012). Chromosomal Replication Initiation Machinery of Low-G+C-Content Firmicutes. J Bacteriol. 194, 5162-70.
[2] Semova, I., Carten, J.D., Stombaugh, J., Mackey, L.C., Knight, R., Farber, S.A., and Rawls, J.F. (2012). Microbiota Regulate Intestinal Absorption and Metabolism of Fatty Acids in the Zebrafish. Cell Host Microbe 12, 277-288.
[3] Backhed, F., Ding, H., Wang, T., Hooper, L.V., Koh, G.Y., Nagy, A., Semenkovich, C.F., and Gordon, J.I. (2004). The gut microbiota as an environmental factor that regulates fat storage. Proc. Natl. Acad. Sci. USA 101, 15718–15723.
[4] Calculation of trees from alignments // Jalview documentation
[URL].