Домены и профили.
В первом задании требуется выбрать два домена Pfam и найти в UniProt составленные из них белки.
1.Это семейство содержит белки, связанные с алкилгидропероксидредуктазой (AhpC) и тиоловым
специфическим антиоксидантом (TSA).
ID:AhpC-TSA
AC:PF00578
Название:AhpC/TSA family
Число последовательностей среди бактерий: 36953
2.Это семейство состоит из трансмембранной (т. е. некаталитической) области белков биогенеза цитохрома с,
также известных как дисульфидные обменные белки. Эти белки обладают белковой дисульфидной изомеразой.
ID:DsbD
AC:PF02683
Название:ytochrome C biogenesis protein transmembrane region
Число последовательностей среди бактерий:9876
Поиск по UniProt был проведён со следующим запросом:
taxonomy:"Bacteria [2]" database:(type:pfam pf00578) database:(type:pfam pf02683)
Архитектура домена с описанием:

Pham нашел 150 последовательности с данной архитектурой.
В Uniprot представлена более свежая информация.
Всего было найдено 2710 последовательности, которые я скачала в формате Excel. Сразу же были убраны все белки,
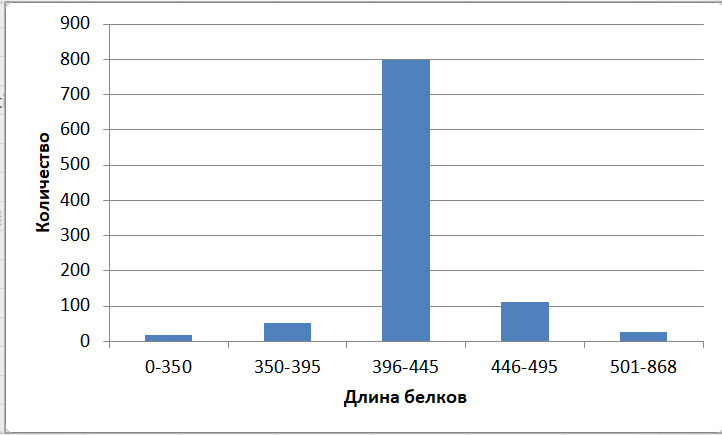
содержащие больше этих двух доменов.Осталось 1012 белков. По длине оставшихся белков была построена гистограмма, ее можно увидеть на
рисунке.Характерная длина белков с такими доменами лежит между 396 и 445 аминокислотными остатками.
Этот интервал и будем считать характерной длиной белка.
Из разных отделов и семейств было выбрано 50 белков(столбец select "+" в колонке).
Таблицу со всеми данными и гистограммой можно скачать по ссылке.
Таблицаф с белками.
Задание 3.
AC из колонки select были скопированы в окно программы Jalview.
Далее:File,Fetch sequences, Uniprot,OK. С помощью Mafft with Defaults последовательности были выровнены.
Я удалила 5 не ообо подходящих последовательностей.
С N-концевого конца были удалены 442 последовательности до первого консервативного блока и 4 с C-конца.
N-конец(красным выделены границы удаленных столбцов)
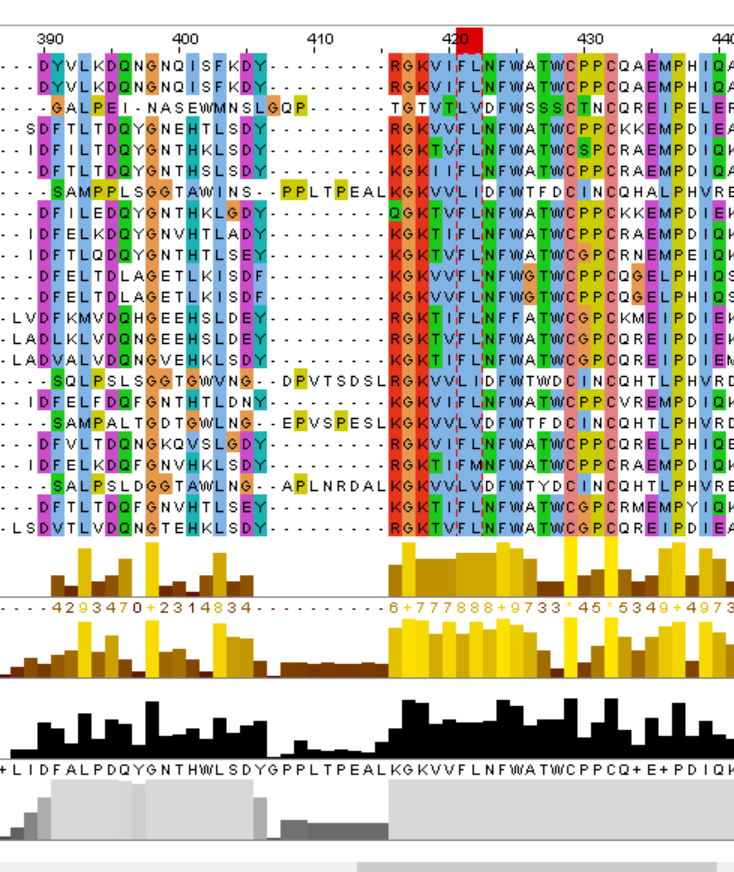
C-конец(Красный-границы удвленных столбцов)

Файл с выравниваниями после обработки:
Файл.
Из Uniprot домен pf02683, так как число бактерий среди бактерий с таким же доменом оказалось меньше, чем с другим.
Запрос в Uniprot -"taxonomy:"Bacteria [2]" database:(type:pfam pf02683)"(47,065 структур).
Белки.
Команды для построение профиля:
hmm2build -g build fail.fa
hmm2calibrate build
hmm2search -E 0.1 build download.fasta > table.txt - нужно, чтобы найти среди всех белков с первым доменом,
те, которые содержат заданную доменную архитектуру.
с E-value 0.1 (рекомендованное значение).
HMM профиль
Все вычисления
Сравнение исходной таблицы и таблицы белков (HMM профиля) я сравнивала с помощью методов Excel.Одинаковые AC выделены цветом,
а затем 1 отмечались одинаковые(колонка совпадения) AC, а 0 несовпадающие. Веса были отсортированы по убыванию.На основе вычисленных данных
были построены графики и таблица предсказаний(все есть в Excel в выделенном окне).
График весов.
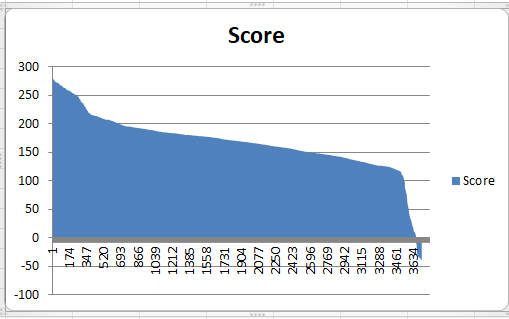
Моей целью было подобрать оптимальный порог веса для более менее точного предсказания находок с нужной
доменной архитектурой.На графике была выбрана точка с весом 185,3.
Она расположена в месте (на 1134 строке),
где график начинает стремиться к асимптоте.Чувствительность:0,68986694, 1- специфичность:0,002271617.
Определяя этут точку, я ориентировалась на max(есть в таблице) значение F1(0,638862559).
Ее значения и стали пороговыми, по которым заполнялась таблица предсказаний и истины.
ROS
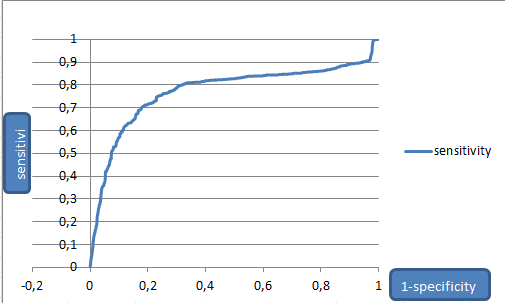
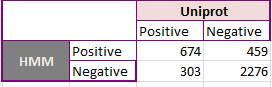
Таблица предсказаний.
Порог: вес 185,3.По ней можно судить о качестве предсказания.