| Идентификатор GenBank | Идентификатор RefSeq | Уровень сборки генома | Общий размер генома (п.н.) | Число фрагментов генома в сборке | Параметры N50 и L50 (контиги / скэффолды) |
|---|---|---|---|---|---|
| GCA_028858775.3 | GCF_028858775.2 | Chromosome | 3 177 739 762 п.н.; | Число контигов: 30, Число скэффолдов: 25 |
Contigs: N50 = 146 288 486 п.н.; L50 = 9; Scaffolds: N50 = 146 288 486 п.н.; L50 = 9 |
Пользуясь расширенным поиском на сайте NCBI, было выяснено:
- Всего записей по мРНК: 37690 (все мРНК, со словом в заголовке "Glutamine"). Запрос: (Glutamine[Title]) AND biomol_mrna[Properties]
- Всего записей по генам: 23894 (все типы нуклеотидных последовательностей, со словом в заголовке "Glutamine"). Запрос: Glutamine[Title] AND biomol_genomic[Properties]
-
Для мРНК:
- 33,488 - из базы RefSeqk
- 4,201 - из базы GenBank
-
Для генов:
- 8 - из базы RefSeq
- 23,885 - из базы GenBank
-
Для Homo sapiens по мРНК:
- 114 - из базы RefSeqk
- 494 - из базы GenBank
- Запрос: Glutamine[Title] AND biomol_mrna[Properties] AND "Homo sapiens"[Organism]
-
Для Homo sapiens по геному:
- 4 - из базы RefSeqk
- 158 - из базы GenBank
- Запрос: Glutamine[Title] AND biomol_genomic[Properties] AND "Homo sapiens"[Organism]
Пользуясь расширенным поиском сайта ENA, было выяснено:
- Всего записей по мРНК для Homo sapiens: 719. Запрос: description="Glutamine" AND scientific_name="Homo sapiens" AND mol_type="mrna"
- Всего записей по генам для Homo sapiens: 5. Запрос: description="Glutamine" AND scientific_name="Homo sapiens" AND mol_type="genomic dna"
- Всего записей по мРНК для Homo sapiens: 711. Запрос: definition="Glutamine" AND Organism="Homo sapiens" AND molecular type="mrna"
- Всего записей по генам для Homo sapiens: 5. Запрос: definition="Glutamine" AND Organism="Homo sapiens" AND molecular type="dna"
Пользуясь расширенным поиском сайта DDBJ, было выяснено:
В реальной ситуации я бы выбрал поиск NCBI или DDBJ, но скорее NCBI. Система поиска ENA совсем не понравилась, мне она показалась неудобной и интуитивно непонятной.
Последовательность белка в формате fasta тут
Идентификатор белка: XP_001138674.1
Идентификатор нуклеотидной записи: NC_072417

Для исследования на консервативность последовательности окрестности гена, проводилось выравнивание методами blastn и tblastn среди отряда Araneae. База данных - refseq genomes.
blastn в данном случае применим потому, что ищет последовательности с минимальным сходством среди геномов пауков. Среди стандартных параметров изменен только размер слова - 7, чтобы уменьшить длину совпадающего участка и найти больше совпадений.

Было найдено 92 совпадения среди всех 4 сборок в базе refseq_genomes среди пауков. Было найдено несколько действительно хороших совпадений с низким e-value(4e-30 - 4e-11).
tblastn используется для поиска гомологичных последовательностей исходной среди пауков.Т.к. одна аминокислота кодируется разными кодонами, то нуклеотидные последовательности могут сильно отличаться. Поэтому blastn не найдёт такие последовательности. А tblastn транслирует эти последовательности и сравнивает с последовательностью белка. Длина слова - 2, остальные параметры - по умолчанию.

Было найдено всего лишь два совпадения с высоким e-value(0.024 и 0.037).Можно сделать вывод, что в последовательности белка нет таких консервативных участков, которые бы сохранились у дальних друг от друга таксонов.
Предварительно перед поиском гомологов генов рРНК E.Coli в геноме P. troglodytes была создана локальная база данных "chim" при помощи команды:
В файлы 16S_E.Coli.txt и 23S_E.Coli.txt сохранены последовательности. При помощи локального blastn они были выравнены с последовательностями P. troglodytes из базы chim. Команды:
blastn -task blastn -db chim -query 16S_E.Coli.txt -out 16s_res.out -outfmt 7
blastn -task blastn -db chim -query 23S_E.Coli.txt -out 23s_res.out -outfmt 7
Ссылка на результат выравнивания 16sРНК
Ссылка на результат выравнивания 23sРНК
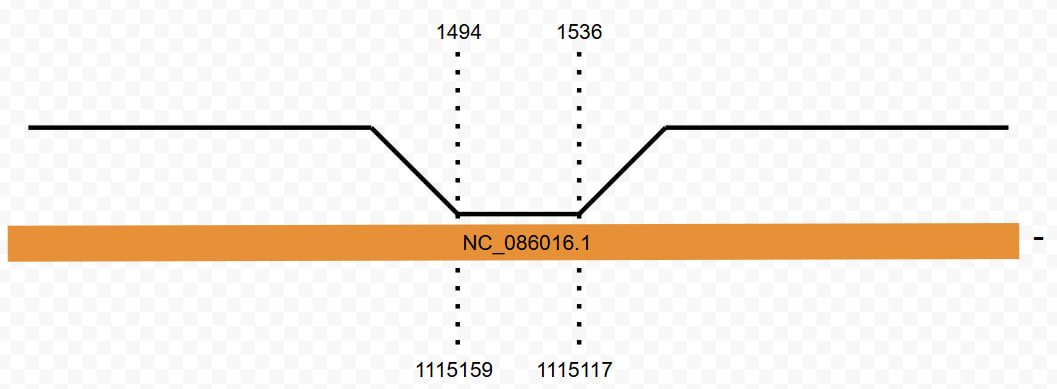
Было обнаружено 84 совпадения. Почти всегда E-value повторялись среди 1.03e-04 и 7.9. Результаты с E-value 1.03e-04 соответсвуют участку 1494 - 1536 рРНК E.Coli.
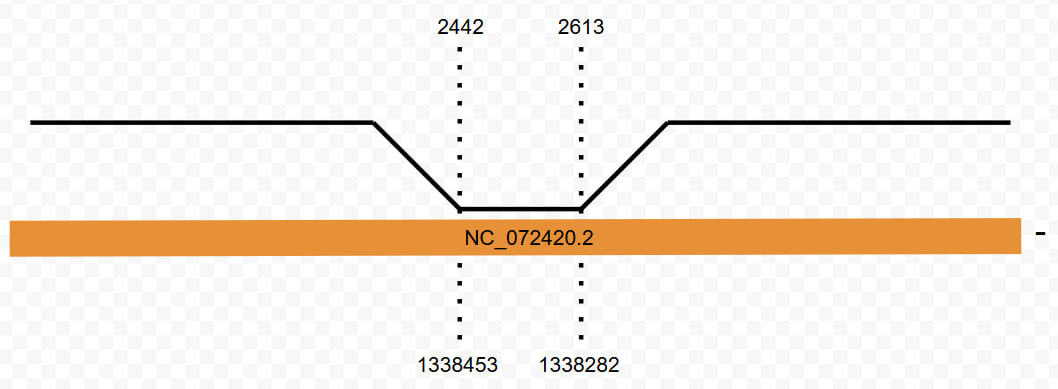
Было обнаружено 164 совпадения. Самый низкий E-value - 6.38e-17, соответствует участку 2442 - 2613.
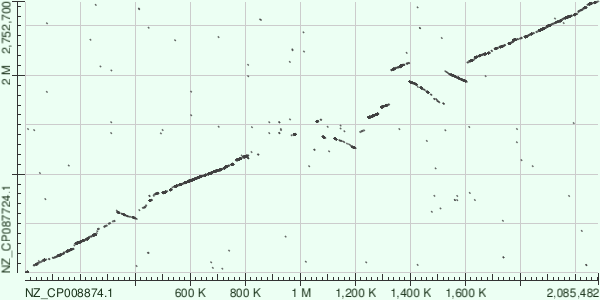
Для построения карт локального сходства были взяты археи разных штаммов: Halanaeroarchaeum sulfurireducens и Halanaeroarchaeum sp. HSR-CO. Коды доступа RefSeq NZ_CP008874.1 и NZ_CP087724.1 соответственно. Первую архею я описывал в своём миниобзоре.При использовании алгоритмов blastn и megablast плазмидные последовательности были получены при помощи всё тех же кодов доступа RefSeq.

blastn нашёл множество коротких совпадений, скорее всего являющиеся дупликациями. Этот алгоритм более чувствителен, чем megablast, т.к. последний ищет очень схожие последовательности.