Отчет по практикуму 4
1. Предсказание вторичной структуры заданной тРНК
1.1 Предсказание вторичной структуры тРНК при помощи поиска инвертированных повторов
Для предсказания инвертированных поворов в последовательности тРНК, найденной в банке PDB по идентификатору 2cv0, была использована программа einverted из пакета EMBOSS (параметры подобраны вручную в попытке получить наиболее достоверную вторичную структуру):
einverted -sequence 2CV0.fasta -gap 15 -threshold 10 -match 4 -mismatch -10
-outfile 2CV0.outfile -outseq 2CV0.outseq1.2 Предсказание вторичной структуры тРНК при помощи алгоритма Зукера
Предсказание вторичной структуры тРНК по алгоритму Зукера было выполнено с помощью инструмента ViennaRNA, запущенного в JupiterNotebook. Полученная структура тРНК представлена на Рисунке 1.
| Участок структуры | Позиции в структуре (по результатам find_pair ) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 1 – 7 и 71 – 65 | 3 – 7 и 69 – 65 | 1 – 7 и 71 – 65 |
| D-стебель | 10 – 14 и 25 – 21 | — | 9 – 13 и 22 – 26 |
| T-стебель | 48 – 53 и 64 – 57 | 48 – 52 и 64 – 60 | 48 – 52 и 60 – 64 |
| Антикодоновый стебель | 26 – 32 и 38 – 44 | — | 27 – 31 и 39 –43 |
| Общее число канонических пар нуклеотидов | 25 | 10 | 22 |
2. Поиск ДНК-белковых контактов
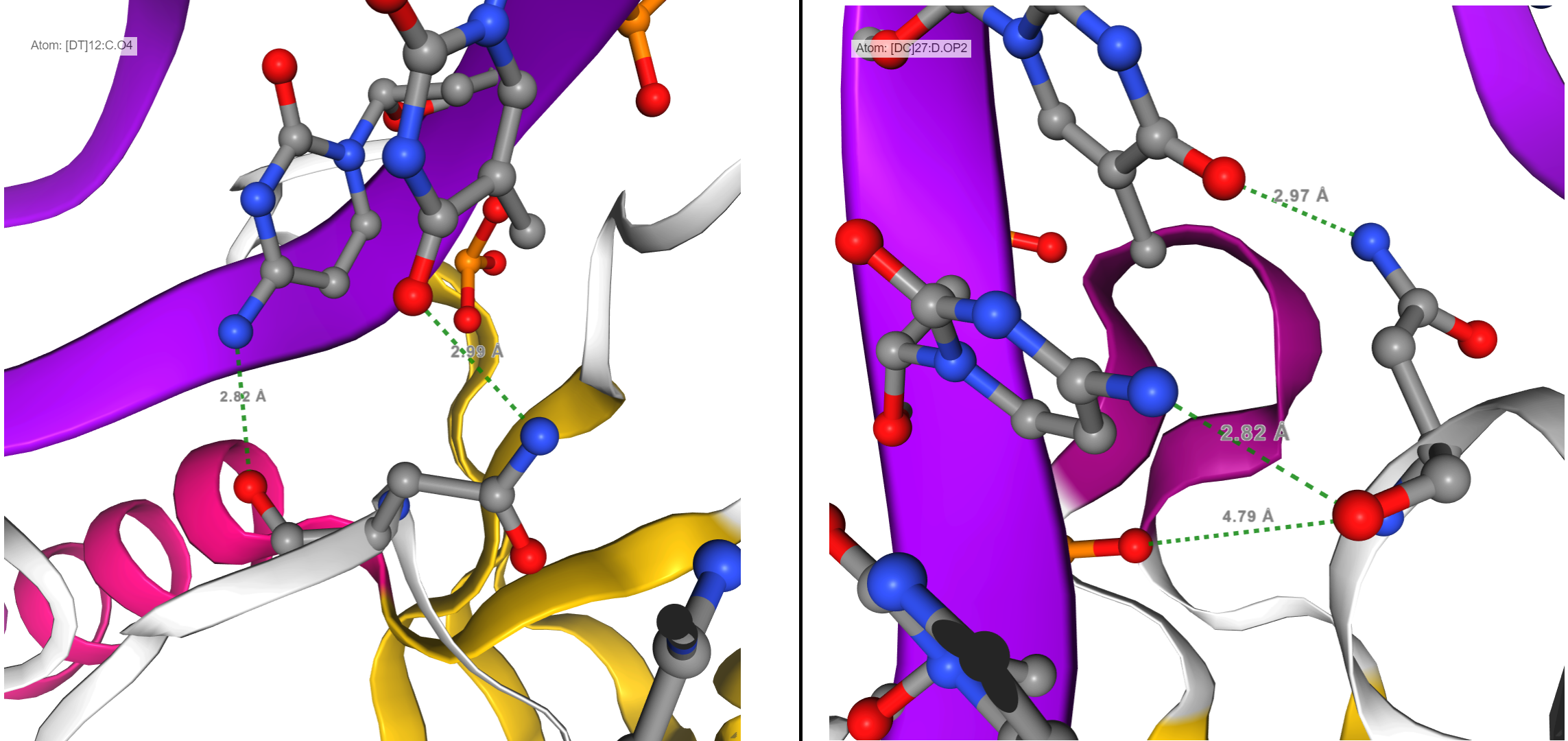
Для определения ДНК-белковых контактов комплекса с идентификатором 1DFM я сначала определил следующие группы атомов: атомы кислорода в составе дезоксирибозы в положении 2' (set1), атомы кислорода в составе остатков фосфорной кислоты (set2), атомы азота в составе азотистых оснований (set3). При помощи скрипта в JMol можно последовательно визуализировать полную структуру ДНК, затем ДНК в проволочной модели, потом ДНК в проволочной модели с последовательно выделенными при помощи шариков атомами групп set1, set2, set3.
Набор контактов ДНК-белок в комплексе 1DFM были получены при помощи скрипта в JMol.
| Участники взаимодействия с белком | Полярные | Неполярные | Всего |
| остатки 2'-дезоксирибозы | 7 | 47 | 54 |
| остатки фосфорной кислоты | 25 | 29 | 54 |
| азотистые основания со стороны большой бороздки | 6 | 8 | 14 |
| азотистые основания со стороны малой бороздки | 0 | 2 | 2 |
С помощью программы nucplot была получена схема взаимодействий ДНК-белок:
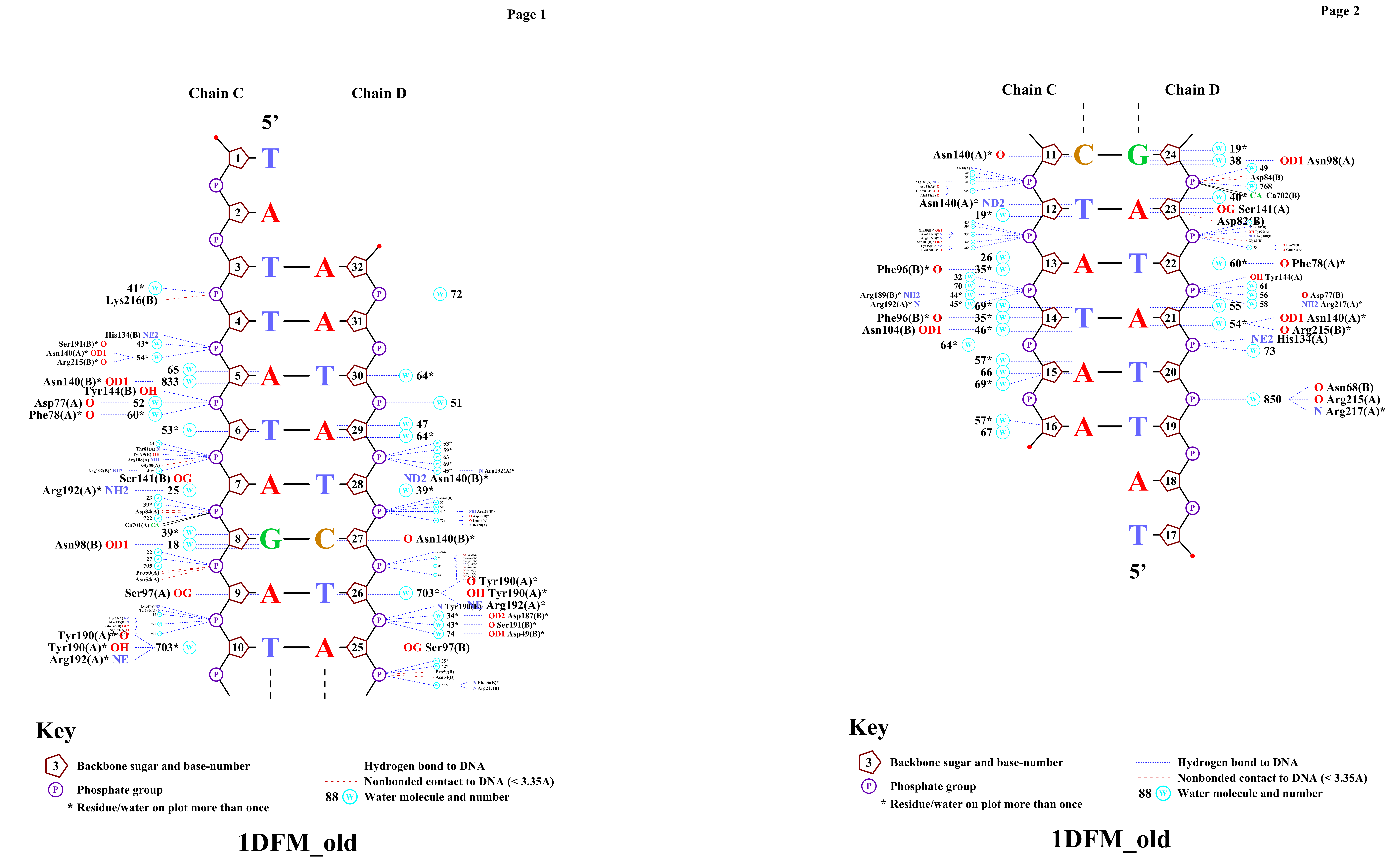
Наибольшее количество контактов с ДНК имеют аминокислоты аспарагин 140 в цепи A ([Asn]140:A) - 5 и аспарагин 140 из цепи B - 4 ([Asn]140:B). Вероятно, они являются наиболее важными для формирования комплекса.