Практикум 12
Сравнение выравниваний одних и тех же последовательностей тремя разными программами
В данном практикуме я решила сравнить выравнивания одних и тех же последовательностей тремя разными программами множественного выравнивания: MAFFT, MUSCLE и T-Coffee.
Для выполнения данного задания я воспользовалась программой, написанной Ксюшей Кирцовой.
Программу с инструкцией можно найти на её странице.
В практикуме 9 я уже строила множественное выравнивание при помощи программы MUSCLE ы Jalview. Для упрощения задачи возьмем те же самые последовательности и будем сравнивать их выравнивания тремя разными программами.
Для этого я открыла проект в Jalview, который создала в 9 практикуме. Далее дублировала окно для дальнейших манипуляций, удалила все гэпы в новом окне (edit -> remove all gaps). Затем перевыровняла последовательности оставшимися двумя программами (Web Service -> Alignment -> Mafft with Defaults/T-Coffee with Defaults).
Проект в Jalview
Полученные выравнивания я сохранила в формате fasta для того, чтобы подать на вход программе для сравнения выравниваний.
Я решила выбрать выравнивание MAFFT в качестве референсного и сравнить его с двумя другими. Мой выбор обусловлен данными, полученными в результате сравнения программ на BaliBase (база данных "идеальных" выравниваний). Учитывая эти данные, выравнивание программой MAFFT больше похоже на референсное по сравнению с выравниваниями двумя другими программами.
Список блоков одинаково выровненных колонок представлен в таблице 1.
Таблица 1. Список блоков одинаково выровненных колонок.
| № блока |
MAFFT с MUSCLE |
MAFFT с T-Coffee |
| 1 |
(1-337)=(1-337) |
(62-105)=(62-105) |
| 2 |
(378-491)=(380-493) |
(110-158)=(110-158) |
| 3 |
(507-541)=(509-543) |
(168-179)=(168-179) |
| 4 |
(544-555)=(546-557) |
(184-185)=(184-185) |
| 5 |
(582-593)=(583-594) |
(196-207)=(196-207) |
| 6 |
(595-599)=(596-600) |
(222-256)=(222-256) |
| 7 |
(601-654)=(602-655) |
(259-289)=(259-289) |
| 8 |
(664-792)=(663-791) |
(292-337)=(292-337) |
| 9 |
(804-1009)=(803-1008) |
(378-491)=(383-496) |
| 10 |
(1013-1025)=(1012-1024) |
(503-504)=(508-509) |
| 11 |
|
(507-542)=(512-547) |
| 12 |
|
(546-552)=(551-557) |
| 13 |
|
(582-654)=(586-658) |
| 14 |
|
(662-792)=(664-794) |
| 15 |
|
(804-904)=(806-906) |
| 16 |
|
(912-1008)=(914-1010) |
| 17 |
|
(1012-1020)=(1014-1022) |
| 18 |
|
(1022-1023)=(1024-1025) |
При сравнении выравниваний программами MAFFT и Muscle не было обнаружено одинаково выровненных колонок, не входящих в блоки. Процент выровненных колонок в случае MAFFT 85.46%, в случае Muscle – 85.55%. Разница в проценте выровненных колонок объясняется разницей в длине выравниваний (1025 и 1024 соответственно).
Аналогично нет одинаково выровненных колонок, не входящих в блоки, и при сравнении выравниваний программами MAFFT и T-Coffee. Процент выровненных колонок в выравнивании программой MAFFT – 85.46%, программой T-Coffee – 85.30% (длины выравниваний 1025 и 1027 соответственно).
Рассмотрим участок, на котором колонки выровнялись неодинаково при использовании трех разных программ (рис. 1, 2, 3). К сожалению, качество изображений оставляет желать лучшего, однако суть всё ещё остается уловимой.
На каждом изображении в начале и конце я оставила по фрагменту из блоков, на котором колонки выровнялись одинаково. После колонки лизинов выравнивания начинают различаться. Можно увидеть, что в выравнивании программой MAFFT приоритет отдается тому, чтобы колонка состояла из идентичных аминокислот (658 колонка, глицин), несмотря на появление гэпов.
При выравнивании программой Muscle гэпов на данном участке заметно меньше. Однако и абсолютно идентичных колонок на участке нет.
Выравнивание программой T-Coffee же занимает некоторое промежуточное положение: в нём тоже не так много гэпов, однако в колонках больше аминокислот с схожими свойствами.
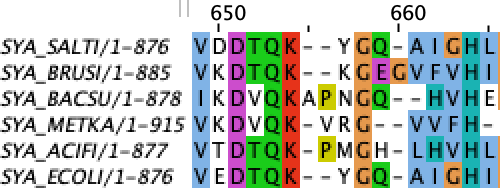 Рисунок 1. Участок выравнивания программой MAFFT.
Рисунок 1. Участок выравнивания программой MAFFT.
 Рисунок 2. Участок выравнивания программой Muscle.
Рисунок 2. Участок выравнивания программой Muscle.
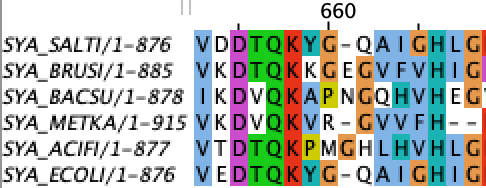 Рисунок 3. Участок выравнивания программой T-Coffee.
Рисунок 3. Участок выравнивания программой T-Coffee.
Выравнивание по совмещению структур и его сравнение с выравниванием MSA
Изначально для выполнения данного задания я хотела взять 3D структуры белков семейства PFAM, которое выбрала для выполнения предыдущего практикума. Однако я обнаружила, что из трёх представленных структур две относятся к одному и тоже же белку. По этой причине я выбрала другое семейство PFAM, а именно PF00045.
В данном семействе я выбрала три разных белка (1ck7, 1gxd, 3ba0).
С помощью Pairwise Structure Alignment на сайте PDB было построено выравнивание по совмещению структур. Результаты выдачи представлены в таблице 2 и на рисунке 4.
Таблица 2. Результаты структурного выравнивания.
| Entry |
Chain |
RMSD |
TM-Store |
Identity |
Aligned Residues |
Sequence Length |
Modeled Residues |
| 1CK7 |
A |
- |
- |
- |
- |
631 |
619 |
| 1GXD |
A |
2.82 |
0.92 |
93% |
561 |
631 |
624 |
| 3BA0 |
A |
2.08 |
0.3 |
35% |
188 |
365 |
365 |
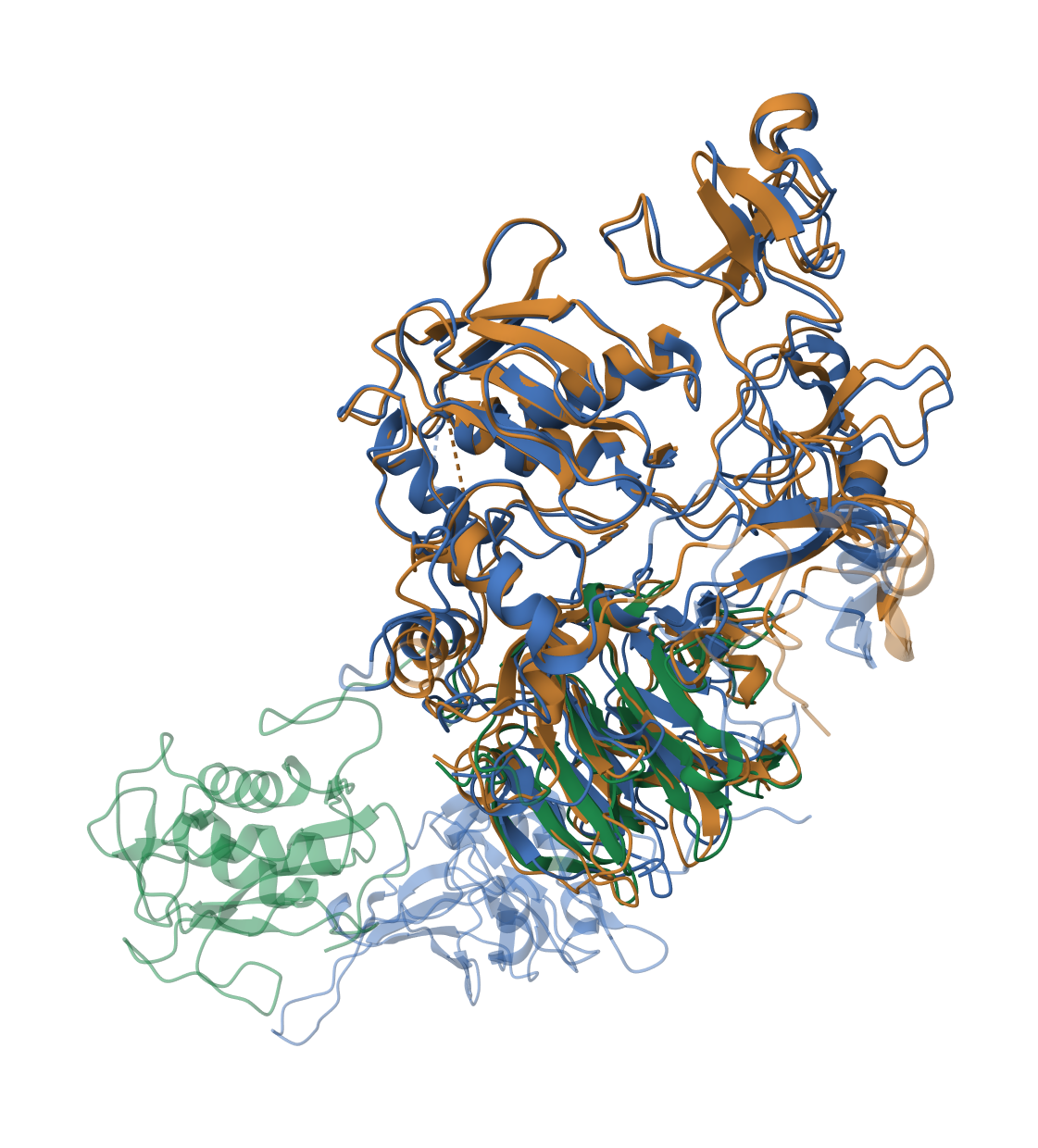 Рисунок 4. Структурное выравнивание. 1CK7 – оранжевым, 1GXD – синим, 3BA0 – зеленым.
Рисунок 4. Структурное выравнивание. 1CK7 – оранжевым, 1GXD – синим, 3BA0 – зеленым.
Также для этих же белковых последовательностей было построено множественное выравнивание MSA. Для этого я открыла нужные последовательности в программе Jalview и выровняла при помощи программы MAFFT.
Из двух парных выравниваний (1CK7 с 1GXD и 1CK7 c 3BA0) по совмещению структур я сделала одно множественное выравнивание путем добавления гэпов вручную в Jalview.
Выравнивание в Jalview
В начальном участке выравнивания по совмещению структур и MSA для 1CK7 и 1GXD отличаются, однако далее они одинаковы. Для 1CK7 и 3BA0 они отличаются в начальном и конечном участке, большинство позиций в "средней" части выровнялись одинаково.
В целом результаты выравнивания по совмещению структур и MSA сопоставимы.
Описание программы T-Coffee
T-Coffee (Tree-based Consistency Objective Function for Alignment Evaluation) – это программа множественного выравнивания последовательностей. Данная программа использует прогрессивное выравнивание. Проблемой прогрессивного выравнивания является то, что ошибка, допущенная в первых выравниваниях, сохраняется до конца. T-Coffee же сводит к минимуму эту проблему: эта программа позволяет на каждом этапе выравнивания учитывать информацию из всех последовательностей, а не только тех, которые были выровнены на данном этапе.
T-Coffee сочетает в себе свойства локальных и глобальных выравниваний. Такой метод выравнивания оказался более точным, чем его предшественники.
У T-Coffee есть несколько режимов работы в зависимости от поставленной задачи и необходимой точности.
К достоинствам программы можно отнести то, что она довольно точная. Однако существует и значительный недостаток: время работы T-Coffee существенно превышает время работы других часто используемых программ множественного выравнивания.
Используемые источники
1) Notredame C., Higgins D.G., Heringa J., 2000. T-Coffee: A novel method for fast and accurate multiple sequence alignment // Journal of Molecular Biology. V. 302. P. 205-217.
2) Tommaso P.D., Moretti S., Xenarios I., Orobitg M., Montanyola A., Chang J-M., Taly J-F., Notredame C., 2011. T-Coffee: a web server for the multiple sequence alignment of protein and RNA sequences using structural information and homology extension // Nucleic Acids Research. V. 39. P. 13-17.
3) Веб-сайт T-Coffee