Мини-обзор генома и протеома бактерии
Photobacterium ganghwense
Симоненкова С.А.
Факультет биоинженерии и биоинформатики, МГУ им. М. В. Ломоносова, 2023
Email: simonasmit@fbb.msu.ru
Аннотация
Мотивация: в рамках курса "Практическая информатика" в течении семестра студенты приобретают базовые навыки работы с биологическими данными. Данный опыт используется для написания мини-обзора по выбранному студентом микроорганизму.
Результаты: данный мини-обзор содержит анализ генома и протеома Photobacterium ganghwense, а именно информацию о длинах белков, расположении генов на репликонах, GC-skew репликонов.
1. Введение
Photobacterium ganghwense – вид морских грамотрицательных бактерии рода Photobacterium, семейства Vibrionaceae, класса Gammaproteobacteria. Представители данного рода являются биолюминесцентными, для них характерна положительная реакция на оксидазу и каталазу, подвижность, галофильность, палочковидная форма. Впервые вид Photobacterium ganghwense был выделен из образца морской воды с острова Канхва, Южная Корея (Park Y. D. et al., 2006). Также данный вид был выделен, охарактеризован и идентифицирован из морских губок района острова Святого Мартина в Бенгальском заливе, Бангладеш (Paul S. I. et al., 2021). Исходя из фенотипического и геномного анализа, использование Photobacterium ganghwense перспективно для производства ПГА (полигидроксиалканоатов) – биоразлагаемых и биосовместимых биопластиков, многообещающая экологичная альтернатива пластмассам на нефтехимической основе. (Lascu I. et al., 2022).
2. Материалы и методы
В работе исследуется геном и протеом штамма С2.2, выделенного из береговой линии Черного моря. Геном данного штамма является референсным, содержит 2 хромосомы и 1 плазмиду. Для получения результатов использовались данные из NCBI: таблица особенностей (Сопр. мат. 1). Для построения гистограммы распределе-ния длин белков и составления таблицы расположения генов на репликонах использовалась google таблица с особенностями генома (Сопр. мат. 2, лист feature_tables). По данной таблице была построена вспомогательная таблица с характеристиками кодирующий последовательностей (лист protein_CDS), на основании которой были построены гистограммы длин белков (лист protein_len_hist). Также по таблице особенностей для каждого из репликонов было посчитано количество генов белков и генов разных типов РНК (лист per-replicones). Из NCBI были получены полные последовательности в формате FASTA для каждого из репликонов (Сопр. мат. 3). Для подсчета GC-skew репликонов использовался скрипт, написанный в Visual Studio Code на python 3.11.7 (Сопр. мат. 4).
3. Результаты и обсуждение
3.1. Распределение длин белков
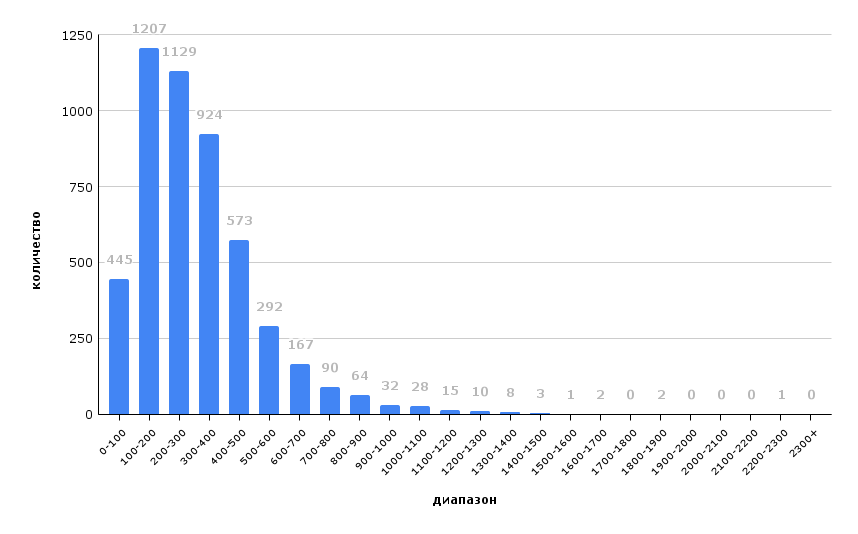
Была получена гистограмма длин белков P. ganghwense (Рис. 1). Всего геном бактерии кодирует 4984 белков, средняя длина белка – 315 аминокислот, медиана – 273 аминокислоты. Согласно распределению, самый частый диапазон длины белка составляет от 100 до 200 аминокислот. При этом максимальная длина – 2206 аминокислот. На гистограмме наблюдается один пик, резкий скачок между диапазонами 0-100 и 100-200 аминокислот. В целом, длина последовательности белка определяется его функцией, и широкий разброс в длинах белков организма отражает разнообразие специфических функциональных ролей этих белков (Lipman D. J. et al., 2022). Для P. ganghwense наблюдается небольшой разброс в длинах белков, большая часть из них сконцентрирована в диапазоне от 0 до 700 аминокислот, что, согласно утверждению выше, говорит о невысоком разнообразии специфических функциональных ролей белков бактерии.
3.2. Распределение генов по репликонвм
Геном P. Ganghwense состоит из трех репликонов:
1. хромосома 1 (RefSeq: NZ_CP071325.1);
2. хромосома 2 (RefSeq: NZ_CP071326.1);
3. плазмида pPGC22 (RefSeq: N_CP071327.1).
Для каждого из репликонов было посчитано количество генов белков и генов разных типов РНК (Табл. 1).
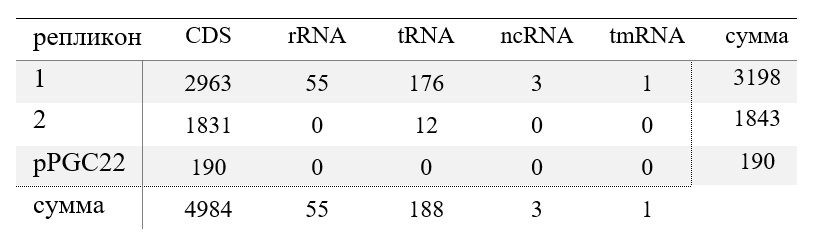
Из данных видно, что большая часть генов бактерии располагается на первой хромосоме. На второй хромосоме помимо генов белков закодировано 12 тРНК. С плазмиды может идти трансляция исключительно белков. Как показано для других видов рода, вторая хромосома и плазмида могут быть источником геномной пластичности и различий, специфичных для штаммов (Lascu I. et al, 2021). На первой хромосоме закодирована tmRNA – транспортно-матричная РНК, реализующая транс- трансляци (Keiler K. C. Ramadoss N. S, 2011).
3.3. GC-skew и примерное местонахождение орджина
Подсчет кумулятивного GC-skew позволяет примерно определить положение ориджина репликации (сайт начала репликации) и точки терминации репликации (сайт окончания репликации) по минимуму и максимуму параметра соответственно (Grigoriev, 1998). Результаты по каждому репликону представлены в таблицах (Табл.2, Табл.3).
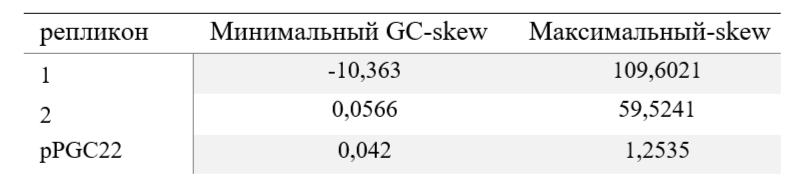
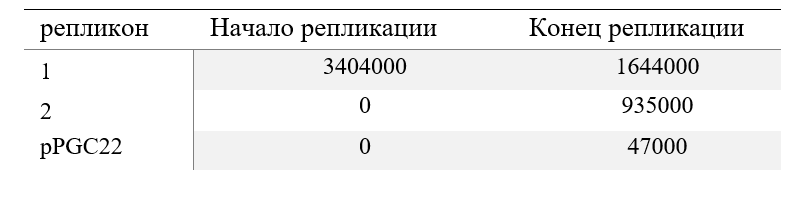
Метод кумулятивных диаграмм показывает, что нуклеотидный состав микробной хромосомы изменяется в двух точках, разделенных примерно половиной ее длины. Эти точки совпадают с сайтами начала и окончания репликации (Grigoriev, 1998). То есть модуль разницы координат этих точек будет примерно равен половине от длины хромосомы. Этому правилу соответствует только первая хромосома, длиной 3 515 384 п.н. Для второй хромосомы и плазмиды наблюдается странная ситуация, при которой репликация начинается в 0. Причина, вероятно, кроется в длине и GC-составе данных последовательностей, то есть при продвижении далее по последовательности значение GC-skew остается больше чем в начале последовательности. Локальные изменения, видимые искажения диаграммы, могут представлять собой существующие изменения в геноме.
4. Сопроводительные материалы
- Таблица особенностей генома Photobacterium ganghwense из NCBI: ссылка
- Таблица simonenkova_genome с расчетами по длинам белков и расположению генов на репликонах: ссылка
- Полные последовательности репликонов:
- Скрипт в google colab для подсчета GC-skew: ссылка
Список литературы
- Photobacterium ganghwense sp. nov., a halophilic bacterium isolated from sea water. Park Y. D. et al., 2006, International journal of systematic and evolutionary microbiology, Т. 56, № 4, 745-749.
- Identification of marine sponge-associated bacteria of the Saint Martin's island of the Bay of Bengal emphasizing on the prevention of motile Aeromonas septicemia in Labeo rohita. Paul S. I. et al., 2021, Aquaculture, Т. 545, 737156.
- Revealing the phenotypic and genomic background for PHA production from rapeseed-biodiesel crude glycerol using Photobacterium ganghwense C2.2. Lascu I. et al, 2022, Т. 23, № 22, 13754.
- The relationship of protein conservation and sequence length. Lipman D. J. et al., 2022, BMC evolutionary biology, Т. 2, 1-10.
- Complete genome sequence of Photobacterium ganghwense C2.2: A new polyhydroxyalkanoate production candidate. Lascu I. et al., 2021, MicrobiologyOpen, Т. 10, №. 2, e1182.
- Bifunctional transfer-messenger RNA. Keiler K. C. Ramadoss N. S, 2011, Biochimie, Т. 93, №. 11, 1993-1997.
- Analyzing genomes with cumulative skew diagrams. Grigoriev A., 1998, Nucleic acids research, Т. 26, №. 10, 2286-2290.