практикум №2
3DNA
Cравнительный анализ канонической ДНК и стеблей тРНК
Задание 1
Построенные программой fiber структуры ДНК: А-форма, В-форма, Z-форма.
Задание 2
По сравнению с предсказанной структурой ход остова эксперементальной структуры неровный, с небольшими смещениями (выравниваие выполнено в PyMOL).

Заданное основание - цитозин. Выбран цитозин A/9 в структуре 1bna.
В сторону большой бороздки обращены атомы (красные на рис.2):
C9.N1, C9.C2, C9.O2, C9.N3
В сторону малой бороздки обращены атомы (синие на рис.2):
C9.C6, C9.C5, C9.C4, C9.N4

| A-форма | В-форма | Z-форма | |
| Тип спирали | Правая | Правая | Левая |
| Шаг спирали (Å) | 25.9 | 30.9 | 42.3 |
| Число основания на виток | 11 | 10 | 12 |
| Ширина большой бороздки (Å) | 16.8 (A/DC'8/P - B/DT'35/P) |
17.2 (A/DT'11/P - B/DT'27/P) |
- |
| Ширина малой бороздки (Å) | 8.0 (A/DT'11/P - B/DC'24/P) |
11.7 (A/DT'11/P - B/DA'34/P) |
7.2 (A/DG'7/P - B/DG'37/P) |
В таблице 1 приведены числовые характеристики раличных форм ДНК, полученных с помощью программы fiber (поскольку в задании не уточнено, были выбраны предсказанные структуры как более удобные).
В Z-ДНК большая бороздка слабо различима, малая бороздка узкая и глубокая.
Задание 3
Для анализа была задана структура 1f7v.
3.1 Торсионные углы. С помощью конвейера
find_pair -t 1f7v.pdb stdout | analyze
был получен файл 1f7v.out, из которого взяты значения торсионных углов для цепей исследуемой тРНК. Торсионные углы ДНК имеют постоянные значения, из чего можно сделать вывод, что заданная структура тРНК не похожа ни на одну из них (но притянуто, методом пристального взгляда, с натяжкой, на А-форму).
3.2 Структура водородных связей. Также из файла 1f7v.out взята информация о водородных связях, реализованных в структуре исследуемой тРНК.
| Номер стебля | Начало | Последовательность | Конец |
| I | 901 | PUCCUCG | 907 |
| 966 | AAGGGGC | 972 | |
| II | 949 | cCAGG | 953 |
| 965 | GGUCC | 961 | |
| III | 939 | CCAGAA | 944 |
| 931 | GGUCPg | 926 | |
| IV | 910 | gCCC | 913 |
| 925 | CGGC | 922 |
Неканонические пары:
- P901-**--A972
- 905U-*---G968
- 954t-**--a958
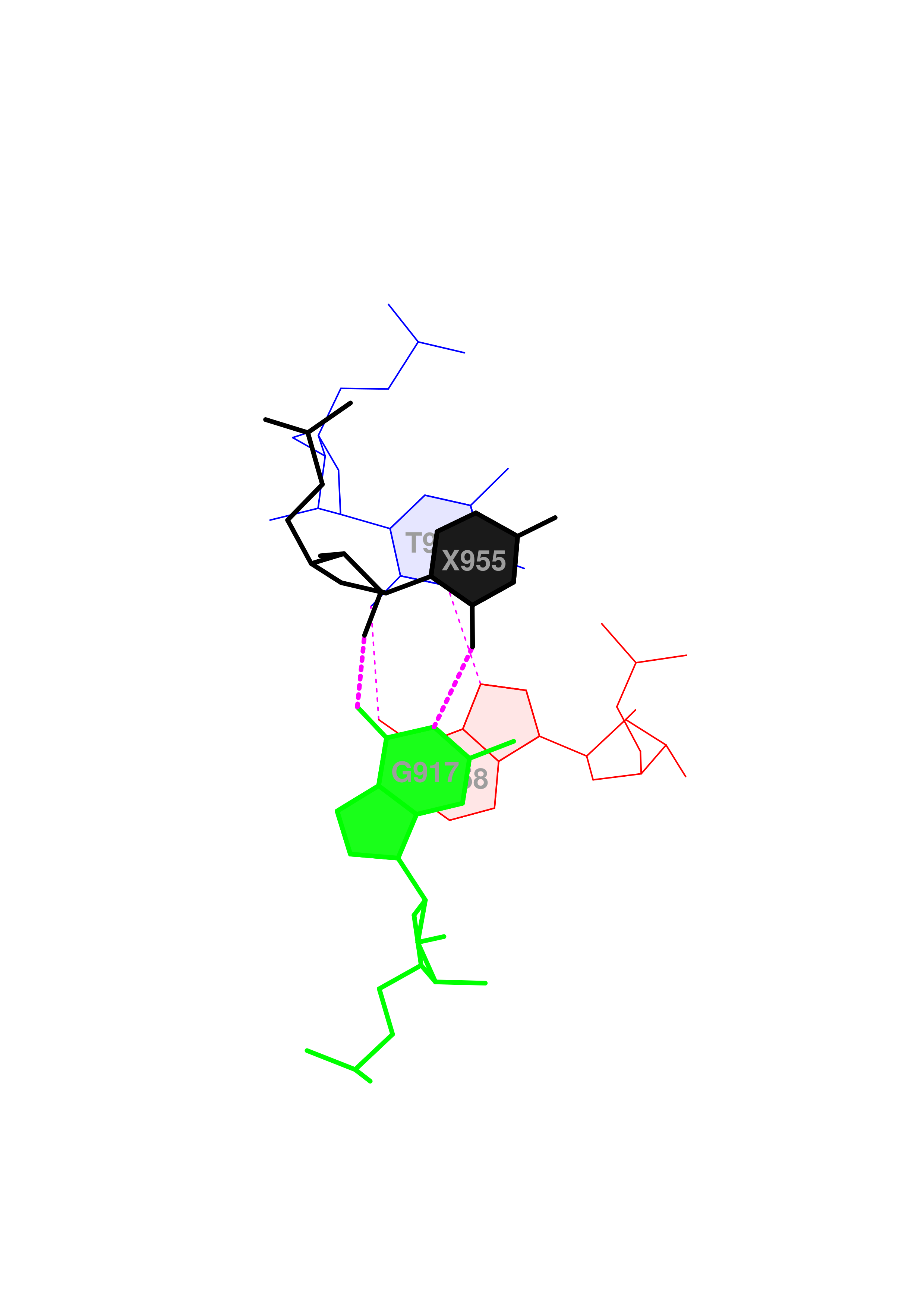
- 955P-**+-G917
- 943A-**--P927
- 944A-*---g926
- 913C-**--C922
- 914A-**--U908
- 915A-**+-U948
Дополнительные водородные связи, стабилизирующие структуру тРНК.
Следующие комплементарные пары нуклеотидов не входят в состав стеблей:
953G-C961, 952t-a958, 955P-G917, 914A-U908, 915A-U948, 918G-C956.
3.3 Стекинг-взаимодействия. Выбор нуклеотидов, для которых предполагается стекинг-взаимодействие, проводился на основе значений из столбца sum (данные о площади перекрывания оснований). Максимальное значение (13.25) имеет 13 пара tP/Ga, минимальное ненулевое (1.52) - 20 пара Ag/Cg. Для построения изображений использовались Section#0013 и Section#0020 соответственно.