практикум №3
/
Предсказание вторичной структуры заданной тРНК и анализ НК-белкового комплекса
1. Предсказание вторичной структуры заданной тРНК
Заданная тРНК - 1F7V. С предоставленными начальными параметрами (-gap 12 -threshold 10 -match 3 -mismatch -3) программа einverted даёт результат, представленный ниже - только один участок комплементарности, примерно соответствующий акцепторному стеблю. Изменение параметров (-gap 14 -threshold 12 -match 3 -mismatch -4) приводит к двум участкам комплементарности, но только один из них почти соответствует предсказанному ранее программой find_pair стеблю (einverted выдала на нуклеотид короче), поэтому для дальнейшего анализа будет использован начальный набор параметров. Предсказание алгоритом Зукера было проведено при помощи RNAfold.
Score 8: 5/6 ( 83%) matches, 0 gaps
2 tcctcg 7
||| ||
71 aggggc 66
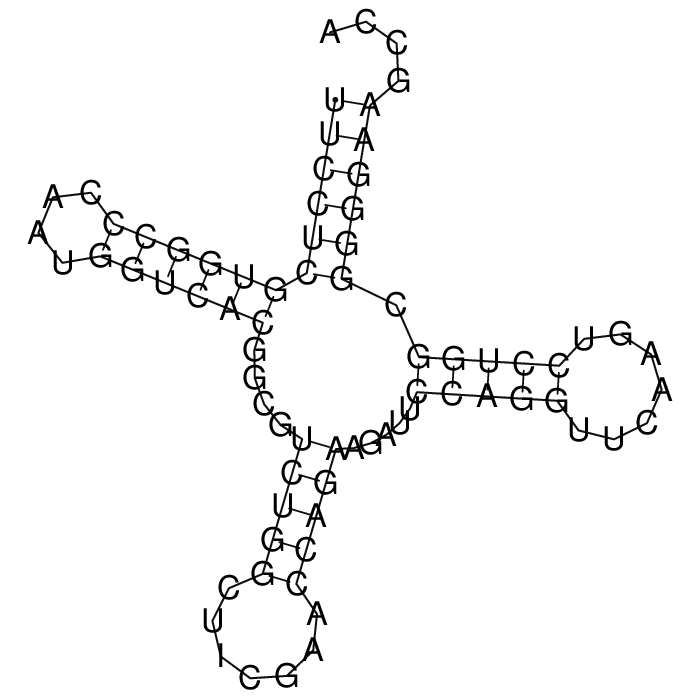
Команда einverted, входящая в пакет EMBOSS, принимает на вход нуклеотидную последовательность ДНК, поэтому для предсказания структуры была всзята последовательность 1F7V из банка NCBI. За счет наличия в последовательность знаков N, предсказанный программой einverted стебель смещен на одно положение (первый нуклеотид как потерян). Также использование подобной последовательности не предусматривает неканонические пары нуклеотидов, в отличии от алгоритма Зукера (пара 5U-68G). По табл. 1 видно, что алгоритм Зукера намного точнее предсказывает структуру РНК, чем программа einverted.
| Участок структуры | Позиции в структуре (find_pair) | Результаты предсказания с помощью einverted | Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 901-907 972-966 (всего 7 позиций) | 2-7 \ 5 71-66 \ 68 | предсказано 6 пар из 7 |
| D-стебель | 910-913 925-922 (всего 4 позиции) | - | предсказано 6 пар из 4 |
| Антикодоновый стебель | 939-944 931-926 (всего 6 позиций) | - | предсказано 5 пар из 6 |
| T-стебель | 949-953 965-961 (всего 5 позиций) | - | предсказано 5 пар из 5 |
| Общее число канонических пар нуклеотидов | 22 | 5 | 22 |
2. Поиск ДНК-белковых контактов.
Упражнение 1.
Заданный для анализа комплекс ДНК-белок - 1rio. Скрипт для JMol для определения множества атомов set1 (кислород 2'-дезоксирибозы), set2 (кислород в остатке фосфорной кислоты) и set3 (азот в азотистых основаниях), а также последовательного изображения ДНК c выделенными множествами атомов.
Упражнение 2.
Для анализа данных по контактам предложено взять цепь H белка. Принимается, что полярные атомы - O, N, неполярные атомы - C, S, P. Полярные контакты предполагаются при сближении полярных атомов в структуре на дистанции меньше 3.5 А, а неполярные - при сближении неполярных атомов на дистанции меньше 4.5 А.
| Контакты атомов белка с... | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 0 | 9 | 9 |
| остатками фосфорной кислоты | 5 | 4 | 9 |
| остатками азотистых оснований со стороны большой бороздки | 5 | 5 | 10 |
| остатками азотистых оснований со стороны малой бороздки | 0 | 0 | 0 |
Визуально можно сразу сказать, что преобладающими ДНК-белковыми контактами будут те, которые располагаются со стороны большой бороздки, т. к. одна из альфа-спиралей цепи Н располагается в углублении большой бороздки. В целом, цепь Н состоит всего из 4 небольших альфа-спиралей, поэтому контактов будет не так много. И, действительно, контактов малой бороздки ДНК с цепью белка не нашлось, контакты большой бороздки преобладают среди других контактов. Почти совпадает количество полярных и неполярных контактов белка с остатками фосфорной кислоты (5 и 4 соответственно). За счет того, что с остатками дезоксирибозы нашлись только неполярные контакты, именно они являются преобладающими (по сравнению с полярными).
Упражнение 3.
Схема ДНК-белковых контактов, полученная с помощью nucplot.
Упражнение 4.
Выбирались аминокислотные остатки цепи H белка.
Наибольшее число указанных на схеме контактов с ДНК
имели Glu399(H) и Thr397(H). Поиск контактов в PyMOL показал, что Thr397(H) взаимодействует с фосфатными остатками
непосредственно и через молекулу воды (рис. 3).

Далее были рассмотрены несколько аминокислотных остатков цепи H, взаимодействующих непосредственно с азотистыми основаниями. Gln414(H) находится в той альфа-спирали, которая входит в углубление большой бороздки, а также связывается с двумя азотистыми основаниями, что позволяет предположить важность его роли в распознавании ДНК (рис.4).
