практикум №10
Prosite, PSI-BLAST, cbcalc
Мотивы в белках: паттерны и PSSM для их поиска.
Поиск консервативных мотивов в выравнивании
Для выполнения данного практикума был выбран домен T-box с идентификатором PF00907. Выравнивание seed этого домена содержит 87 последовательностей, в базе данных SwissProt
104 белка имеют в архитектуре этот домен (Uniprot Curation: Reviewed). Выравнивание было открыто программой Jalview. В нем оказалось 6 похожих последовательностей, которые были
удалены (redundancy 90%), и осталась 81 последовательность. С помощью отображения цветом позиций по уровню идентичности было найдено 9 полностью консервативных колонок, причем они
разнесены по концам выравнивания: 4 в начале и 5 в конце. Благодаря снижению порога идентичности можно найти мотивы - вплотную или рядом расположенные консервативные позиции. Я ориентировалась
на участок с полностью консервативными колонками в конце выравнивания. Похожим на мотив с высоким IC является участок 305-311, который выделяется при понижении порога идентичности вплоть до 92%.
Паттерн предпологаемого мотива (формат Jalview): F.[AVTS]V[TCS].Y
Поиск по всему выравниванию показал, что данный паттерн встречается в 81 последовательности, то есть во всех. Следовательно, подобранный мотив консервативен во всех белках.
Паттерн найденного мотива (формат Prosite): F-x-[AVTS]-V-[TCS]-x-Y
Поиск мотива в базе данных SwissProt в Prosite выдал 721 последовательность, во всех он встречается один раз. При этом, в Pfam домена указано только 104 белка. Полные последовательности находок были выровнены в Jalview алгоритмом Mafft. В получившемся выравнивании паттерн был найден 721 раз. Подавляющее большинство мотивов в выравнивании содержало гэпы. Таким образом, подобранный паттерн не является специфичным для домена T-box.
Проект Jalview, в котором можно ознакомиться с выравниванием seed с выделенным мотивом, а также с выравниванием находок Prosite с также выделенным мотивом. Мотив выделен фиолетовым.
Поиск мотива, специфичного для одной клады филогенетического дерева
Для набора последовательностей из выравнивания seed (без очень похожих последовательностей, см. выше) в Jalview было построено дерево методом NJ (рис. 1). В нем была выбрана клада из 7 последовательностей (отмечена оранжевым) для поиска специфичного мотива. Найденный мотив содержит много консервативных колонок.
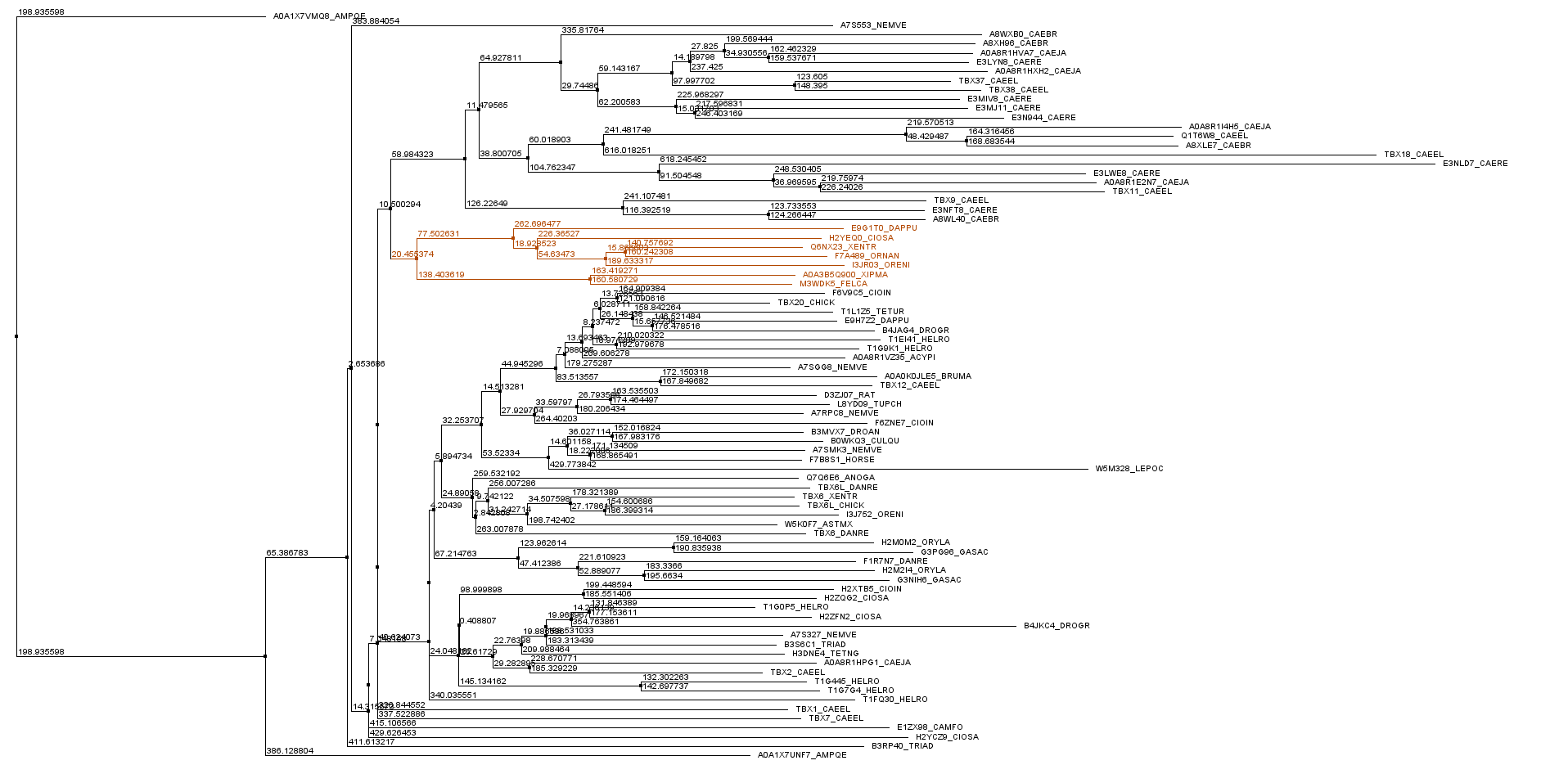
Паттерн предполагаемого мотива: F[KPTQ]ET[RQ]FIAVTAYQN. Найденный мотив является специфичным для клады, поскольку встречается во всех последовательностях клады и не встречается больше нигде в выравнивании.
Проект Jalview, в котором можно ознакомиться с филогенетическим деревом, последовательностями выделенной клады и распространением мотива по последовательностям всего выравнивания. Мотив выделен оранжевым.
PSI-BLAST
Для выполнения задания был выбран AC B2V8C0, по которому алгоритмом PSI-BLAST по бд Swiss-Prot (E 0.005 по умолчанию) выполнялся итеративный поиск дальних гомологов до тех пор, пока результаты этого поиска не стабилизировались (табл. 1).
| № Итерации | Число находок выше порога (0,005) | Идентификатор худшей находки выше порога | E-value худшей находки | Идентификатор лучшей находки ниже порога | E-value лучшей находки |
| 1 | 163 | Q88M41.2 | 0.004 | Q4US07.1 | 0.006 |
| 2 | 188 | Q9ZM51.1 | 6e-07 | A7H8E6.1 | 0.038 |
| 3 | 188 | Q9ZM51.1 | 2e-11 | A7H8E6.1 | 0.025 |
После третьего запуска PSI-BLAST в выдачу не было добавлено гомологов и разница между худшей «правильной» находкой и «лучшей» неправильной увеличилась, что говорит о вероятном нахождении белкового семейства. Все белки выше порога имеют одинаковое название, но принадлежат к различным организмам (выдача), что означает хороший результат поиска. За индентификатором B2V8C0 скрывается белок MinC бактерии Sulfurihydrogenibium sp., strain YO3AOP1. Это ингибитор клеточного деления, блокирующий формирование полярных Z-кольцевых перегородок.
Поиск мотивов de novo с помощью MEME
Для выполнения задания из бд Pfam были скачаны полные последовательности белков из Swiss-Prot, содержащие домен T-box (выборка из 104 последовательностей). На основе выборки было построено выравнивание алгоритмом Mafft в JalView, а также удалены высокопохожие последовательности (redundancy 90%), после чего их осталось 93. Выравание было подано на вход программе MEME следующим образом:
meme SW-pf00907-reduced-93.fa -o results -mod anr -minw 4 -maxw 8 -nmotifs 4С результатами можно ознакомиться по ссылке. Было найдено 4 мотива, все с приемлемо низким E-value. Почти во всех последовательностях был найден какой-то мотив, один мотив может встречаться несколько раз в последовательности. На рис. 2 представлено LOGO самого консервативного мотива, а на рис. 3 - LOGO мотива из первого задания практикума, который был также найден программой.
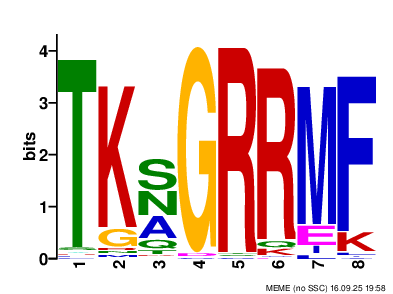

Оценка представленности сайта GATC в геноме бактерии
Оценка представленности сайта метилирования GATC в (полном) геноме Photobacterium ganghwense была проведена командой:
cbcalc -s sites.txt -o result_pr10_5 -M ~/term1/genome/GCF_017329545.1_ASM1732954v1_genomic.fnasites.txt - файл со всеми 24 вариантами сайта длины 4 из букв A, T, G, C без повторений. Программа cbcalc для вычисления контрастов основана на методе Карлина с соав.
С результатами можно ознакомиться по ссылке. На рис. 4 представлена гистограмма контрастов с выделенным сайтом GATC. Его obs/exp составляет 0,8, что значит, что данный сайт встречается в наблюдаемой последовательности реже, чем это ожидалось бы случайно. Наверное, из этого можно сделать вывод, что и в геноме исследуемой бактерии сайт GATC имеет важные регуляторные функции, поскольку не встречается случайно.