практикум №4
методы
Практические аспекты реконструкции филогении
Составление списка гомологичных белков, включающих паралоги.
Для выполнения практикума было выбрано 7 протеомов бактерий отдела Pseudomonadota: Aromatoleum aromaticum (AROAE), Brucella suis (BRUSU), Haemophilus influenzae (HAEIN), Pasteurella multocida (PASMU), Pseudomonas aeruginosa (PSEAE), Roseobacter denitrificans (ROSDO), Shewanella denitrificans (SHEDO). На основе данных протеомов командой makeblastdb была создана белковая база для поиска гомологов субъединицы сериновой протеазы из E. coli (CLPX_ECOLI). Поиск проводился программой blastp с порогом на e-value равным 0,0001. Ознакомиться со списоком находок из выдачи BLAST можно по ссылке. Из 16 находок только одна имеет статус непросмотренной (имеет в имени tr, что говорит о принадлежности к базе данных TrEMBL).
Последовательности находок с ID получены из общего файла протеомов, по которому создавалась белковая база, с помощью скрипта.
Реконструкция и визуализация.
Для реконструкции дерева найденных гомологов последовательности были выравнены программой muscle. Затем выравнивание было переведено в формат phylip-relaxed с помощью пакета BioPython. Непосредственно реконструкция проводилась программой FastME с использованием модели p-distance (формула Newick). Дерево было визуализировано в сервисе iTOL и укорененно в среднюю точку, после чего на нем были обозначены ортологические группы (рис. 1, 2).
| Ортологи | HSLU PASMU | CLPX PSEAE | CLPX BRUSU |
| HSLU HAEIN | CLPX SHEDO | CLPX ROSDO | |
| Паралоги | HSLU PASMU | HSLU ROSDO | HSLU AROAE |
| CLPX PASMU | CLPX ROSDO | CLPX AROAE |
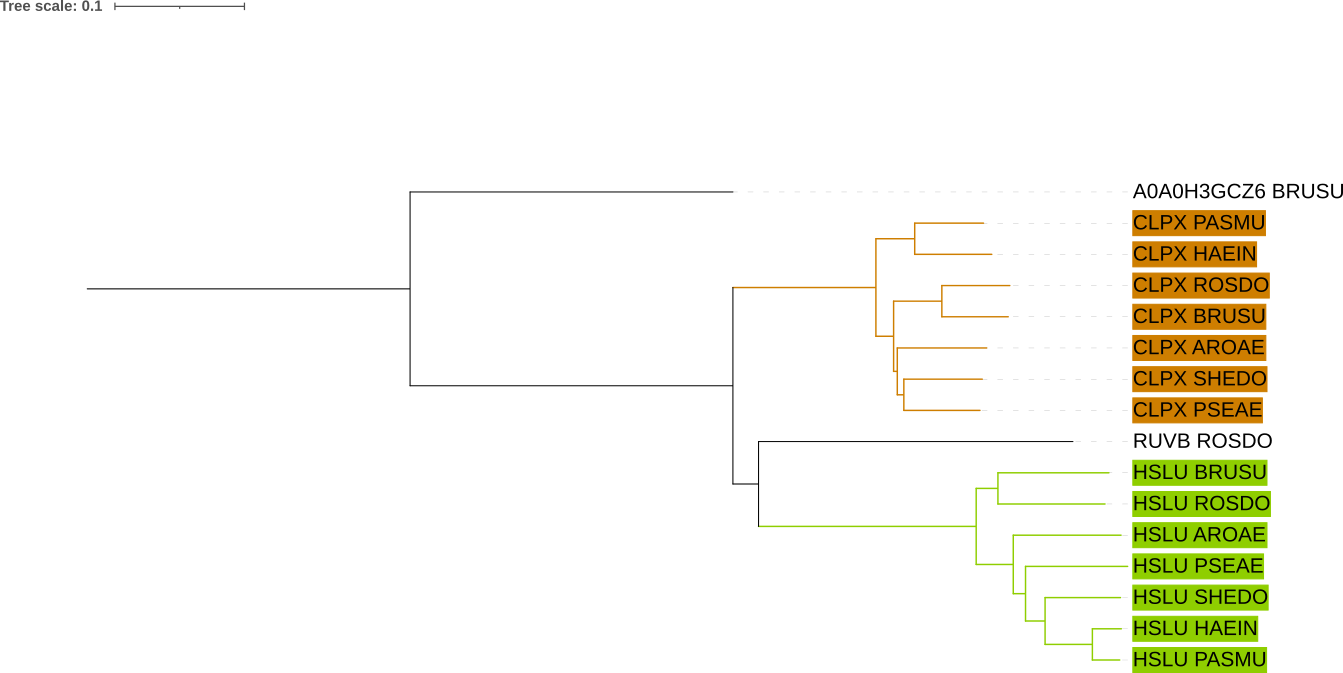

Обе ортологические группы включают 7 белков всех исследуемых бактерий, то есть являются полными. Орт. группа белка CLPX (отмечена коричневым цветом) соответствует принятой филогении бактерий лишь частично:
- Клада (CLPX_HAEIN,CLPX_PASMU) и положение AROAE и PSEAE друг относительно друга сохраняются;
- Клада (CLPX_HAEIN,CLPX_PASMU) реконструирована как сестринская группа к большей кладе остальных белков, однако по филогении бактерий является более поздно отделившейся. Также сестринским к рассматриваемой кладе должен быть белок CLPX_SHEDO, положение которого отдельно от данной клады сохраняется.
Орт. группа белка HSLU (отмечена зеленым цветом) соответствует принятой филогении бактерий.